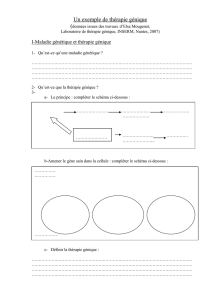
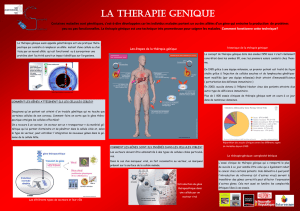
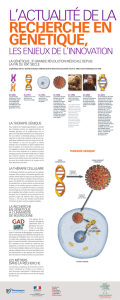
Les utilisations de la thérapie génique

VETAGRO SUP
CAMPUS VETERINAIRE DE LYON
Année 2013-2014 Thèse n°
Les utilisations de la thérapie génique
THESE
Présentée à l’UNIVERSITE CLAUDE-BERNARD - LYON I
(Médecine - Pharmacie)
et soutenue publiquement le Lundi 27 Octobre 2014
pour obtenir le grade de Docteur Vétérinaire
par
HELLEZ Justine
Née le 20 mai 1989
à Seclin (59)

2

3
Liste du corps enseignant
Civilité
Nom
Prénom
Unités pédagogiques
Grade
M.
ALOGNINOUW
A
Theodore
Pathologie du betail
Professeur
M.
ALVES-DE-
OLIVEIRA
Laurent
Gestion des elevages
Maitre de
conferences
Mme
ARCANGIOLI
Marie-Anne
Pathologie du betail
Maitre de
conferences
M.
ARTOIS
Marc
Sante Publique et Veterinaire
Professeur
M.
BARTHELEMY
Anthony
Anatomie Chirurgie (ACSAI)
Maitre de
conferences
Contractuel
Mme
BECKER
Claire
Pathologie du betail
Maitre de
conferences
M.
BELLI
Patrick
Pathologie morphologique et
clinique des animaux de
compagnie
Maitre de
conferences
Contractuel
Mme
BENAMOU-
SMITH
Agnes
Equine
Maitre de
conferences
M.
BENOIT
Etienne
Biologie fonctionnelle
Professeur
M.
BERNY
Philippe
Biologie fonctionnelle
Professeur
Mme
BERTHELET
Marie-Anne
Anatomie Chirurgie (ACSAI)
Maitre de
conferences
Mme
BONNET-GARIN
Jeanne-
Marie
Biologie fonctionnelle
Professeur
Mme
BOULOCHER
Caroline
Anatomie Chirurgie (ACSAI)
Maitre de
conferences
M.
BOURDOISEAU
Gilles
Sante Publique et Veterinaire
Professeur
M.
BOURGOIN
Gilles
Sante Publique et Veterinaire
Maitre de
conferences
M.
BRUYERE
Pierre
Biotechnologies et pathologie
de la reproduction
Maitre de
conferences
Stagiaire
M.
BUFF
Samuel
Biotechnologies et pathologie
de la reproduction
Maitre de
conferences
M.
BURONFOSSE
Thierry
Biologie fonctionnelle
Maitre de
conferences
M.
CACHON
Thibaut
Anatomie Chirurgie (ACSAI)
Maitre de
conferences
Stagiaire
M.
CADORE
Jean-Luc
Pathologie medicale des
animaux de compagnie
Professeur
Mme
CALLAIT-
CARDINAL
Marie-
Pierre
Sante Publique et Veterinaire
Maitre de
conferences
M.
CAROZZO
Claude
Anatomie Chirurgie (ACSAI)
Maitre de
conferences

4
Civilité
Nom
Prénom
Unités pédagogiques
Grade
M.
CHABANNE
Luc
Pathologie medicale des
animaux de compagnie
Professeur
Mme
CHALVET-
MONFRAY
Karine
Biologie fonctionnelle
Professeur
M.
COMMUN
Loic
Gestion des elevages
Maitre de
conferences
Mme
DE BOYER DES
ROCHES
Alice
Gestion des elevages
Maitre de
conferences
Mme
DELIGNETTE-
MULLER
Marie-
Laure
Biologie fonctionnelle
Professeur
M.
DEMONT
Pierre
Sante Publique et Veterinaire
Professeur
Mme
DESJARDINS
PESSON
Isabelle
Equine
Maitre de
conferences
Contractuel
Mme
DJELOUADJI
Zoree
Sante Publique et Veterinaire
Maitre de
conferences
Mme
ESCRIOU
Catherine
Pathologie medicale des
animaux de compagnie
Maitre de
conferences
M.
FAU
Didier
Anatomie Chirurgie (ACSAI)
Professeur
Mme
FOURNEL
Corinne
Pathologie morphologique et
clinique des animaux de
compagnie
Professeur
M.
FRANCK
Michel
Gestion des elevages
Professeur
M.
FREYBURGER
Ludovic
Sante Publique et Veterinaire
Maitre de
conferences
M.
FRIKHA
Mohamed-
Ridha
Pathologie du betail
Maitre de
conferences
Mme
GILOT-
FROMONT
Emmanuell
e
Sante Publique et Veterinaire
Professeur
M.
GONTHIER
Alain
Sante Publique et Veterinaire
Maitre de
conferences
Mme
GRAIN
Francoise
Gestion des elevages
Professeur
M.
GRANCHER
Denis
Gestion des elevages
Maitre de
conferences
Mme
GREZEL
Delphine
Sante Publique et Veterinaire
Maitre de
conferences
M.
GUERIN
Pierre
Biotechnologies et pathologie
de la reproduction
Professeur
Mme
HUGONNARD
Marine
Pathologie medicale des
animaux de compagnie
Maitre de
conferences
M.
JUNOT
Stephane
Anatomie Chirurgie (ACSAI)
Maitre de
conferences
M.
KECK
Gerard
Biologie fonctionnelle
Professeur
M.
KODJO
Angeli
Sante Publique et Veterinaire
Professeur
Mme
LAABERKI
Maria-
Halima
Sante Publique et Veterinaire
Maitre de
conferences
M.
LACHERETZ
Antoine
Sante Publique et Veterinaire
Professeur

5
Civilité
Nom
Prénom
Unités pédagogiques
Grade
Mme
LAMBERT
Veronique
Gestion des elevages
Maitre de
conferences
Mme
LATTARD
Virginie
Biologie fonctionnelle
Maitre de
conferences
Mme
LE GRAND
Dominique
Pathologie du betail
Professeur
Mme
LEBLOND
Agnes
Sante Publique et Veterinaire
Professeur
M.
LEPAGE
Olivier
Equine
Professeur
Mme
LOUZIER
Vanessa
Biologie fonctionnelle
Maitre de
conferences
M.
MARCHAL
Thierry
Pathologie morphologique et
clinique des animaux de
compagnie
Professeur
Mme
MIALET
Sylvie
Sante Publique et Veterinaire
Inspecteur en
sante publique
veterinaire
(ISPV)
Mme
MICHAUD
Audrey
Gestion des elevages
Maitre de
conferences
M.
MOUNIER
Luc
Gestion des elevages
Maitre de
conferences
M.
PEPIN
Michel
Sante Publique et Veterinaire
Professeur
M.
PIN
Didier
Pathologie morphologique et
clinique des animaux de
compagnie
Maitre de
conferences
Mme
PONCE
Frederique
Pathologie medicale des
animaux de compagnie
Maitre de
conferences
Mme
PORTIER
Karine
Anatomie Chirurgie (ACSAI)
Maitre de
conferences
Mme
POUZOT-
NEVORET
Celine
Anatomie Chirurgie (ACSAI)
Maitre de
conferences
Mme
PROUILLAC
Caroline
Biologie fonctionnelle
Maitre de
conferences
Mme
REMY
Denise
Anatomie Chirurgie (ACSAI)
Professeur
M.
ROGER
Thierry
Anatomie Chirurgie (ACSAI)
Professeur
M.
SABATIER
Philippe
Biologie fonctionnelle
Professeur
M.
SAWAYA
Serge
Anatomie Chirurgie (ACSAI)
Maitre de
conferences
Mme
SEGARD
Emilie
Anatomie Chirurgie (ACSAI)
Maitre de
conferences
Contractuel
Mme
SERGENTET
Delphine
Sante Publique et Veterinaire
Maitre de
conferences
Mme
SONET
Juliette
Anatomie Chirurgie (ACSAI)
Maitre de
conferences
Contractuel
M.
THIEBAULT
Jean-
Jacques
Biologie fonctionnelle
Maitre de
conferences
M.
VIGUIER
Eric
Anatomie Chirurgie (ACSAI)
Professeur
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
 46
46
 47
47
 48
48
 49
49
 50
50
 51
51
 52
52
 53
53
 54
54
 55
55
 56
56
 57
57
 58
58
 59
59
 60
60
 61
61
 62
62
 63
63
 64
64
 65
65
 66
66
 67
67
 68
68
 69
69
 70
70
 71
71
 72
72
 73
73
 74
74
 75
75
 76
76
 77
77
 78
78
 79
79
 80
80
 81
81
 82
82
 83
83
 84
84
 85
85
 86
86
 87
87
 88
88
 89
89
 90
90
 91
91
 92
92
 93
93
 94
94
 95
95
 96
96
 97
97
 98
98
 99
99
 100
100
 101
101
 102
102
 103
103
 104
104
 105
105
 106
106
 107
107
 108
108
 109
109
 110
110
 111
111
 112
112
 113
113
 114
114
 115
115
 116
116
 117
117
 118
118
 119
119
 120
120
 121
121
 122
122
 123
123
 124
124
 125
125
 126
126
 127
127
 128
128
 129
129
 130
130
 131
131
 132
132
 133
133
1
/
133
100%