LEXIQUE

LEXIQUE
Phénotype : ensemble des caractéristiques visibles ou analysables d’un individu. Le phénotype se décrit à plusieurs
échelles : moléculaire, cellulaire, macroscopique.
Phénotypes alternatifs : différentes versions d’un même caractère au sein d’une population. Exemple : dans l’espèce
humaine pour le caractère groupe sanguin, les phénotypes alternatifs sont [A], [B], [O], [AB].
Maladie génétique : maladie causée par la mutation d’un gène et la modification de la protéine que ce gène code.
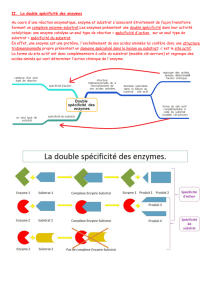
Enzyme : catalyseur biologique, de nature protéique, caractérisé par une double spécificité : de substrat et d’action. Elle
agit à faible dose et à une température compatible avec la vie.
Catalyseur : substance qui accélère une réaction chimique sans en changer le bilan et sans subir de modification elle-
même.
Substrat : molécule sur laquelle agit une enzyme.
Spécificité de substrat : une enzyme donnée ne peut agir que sur un seul substrat.
Spécificité d’action : pour un substrat donné une enzyme ne peut catalyser qu’un seul type de réaction chimique.
Séquence d’un polypeptide : c’est la succession des acides aminés qui constituent la protéine. La séquence est
caractéristique de la protéine. Elle détermine ses propriétés.
Acide aminé : molécule portant une fonction acide (COOH), une fonction amine (NH2) et un radical qui détermine ses
propriétés (hydrophobe, polaire, chargé). Il en existe 20 différents.
Structure primaire d’un polypeptide : enchaînement linéaire d’acides aminés reliés par des liaisons peptidiques.
Structure secondaire d’un polypeptide : disposition d’acides aminés voisins en hélices
ou feuillets
.
Structure tertiaire d’un polypeptide : repliement de la protéine dans l’espace du fait que des acides aminés plus ou moins
lointains établissent des liaisons entre eux (liaisons hydrogène, ponts disulfures...).
Liaison hydrogène : interaction faible entre atomes d’hydrogène porteurs de charges partielles opposées.
Interaction électrostatique : interaction entre charges nettes.
Pont disulfure : liaison covalente stabilisant la structure tertiaire des polypeptides. Elle s’établit entre deux atomes de
soufre portés par deux acides aminés cystéine.
Structure quaternaire d’une protéine : assemblage dans certaines protéines (PAH, hémoglobine) de plusieurs polypeptides
qui sont appelés sous-unités.
Hème : association d’un atome de fer et d’une portion du polypeptide. Il est indispensable à sa fonctionnalité : transport de
l’oxygène dans le cas de l’hémoglobine, catalyse dans le cas de la PAH.
Complexe enzyme-substrat : association étroite entre l’enzyme et son substrat au niveau du site actif. Une fois la réaction
terminée, il se dissocie en libérant les produits formés et l’enzyme.
Site actif : sillon ou cavité d’une enzyme où s’effectue la catalyse. On y distingue un site de reconnaissance permettant la
spécificité de substrat et un site catalytique responsable de la spécificité d’action.

LEXIQUE (SUITE)
Transcription : synthèse d’un ARNm à partir du brin transcrit du gène. Cette réaction a lieu dans le noyau. Elle est
catalysée par une ARNpolymérase. Elle est basée sur la complémentarité des bases azotées.
ARNm : Acide RiboNucléique messager. Formé dans le noyau puis exporté dans le cytoplasme, cet intermédiaire porte le
message servant à la synthèse des protéines. Il diffère de l’ADN par sa nature chimique (sucre = ribose au lieu de
désoxyribose, base U au lieu de T), sa taille (plus court), sa durée de vie (très éphémère).
ARN polymérase : complexe enzymatique qui catalyse la transcription : séparation des deux brins d’ADN, désenroulement
de la double hélice, synthèse de l’ARNm.
Séquence d’un gène : succession des nucléotides sur un brin d’ADN du gène. La séquence est caractéristique du gène.
C’est un message codé (elle contient toutes les informations pour la synthèse d’un polypeptide ayant une séquence en
acides aminés donnée) mais elle n’est pas utilisée directement.
Triplet de nucléotides : trois nucléotides successifs dans l’ADN.
Brin transcrit : brin d’ADN qui sert de modèle pour la synthèse de l’ARNm.
Traduction : synthèse d’un polypeptide dans le cytoplasme à partir de l’information contenue dans l’ARNm correspondant.
Ribosome : complexe moléculaire qui se déplace le long de l’ARNm de codon en codon, permet la mise en place des acides
aminés correspondants et crée des liaisons peptidiques entre eux.
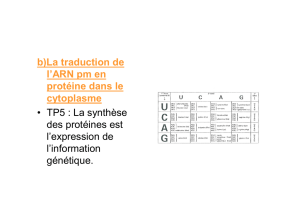
Code génétique : règle de correspondance entre codon de l’ARNm et acide aminé du polypeptide codé. Il est universel, non
ambigu et dégénéré.
Codon : 3 nucléotides successifs surl’ARNm.
Codon initiateur : codon AUG commandant le début de la synthèse du polypeptide.
Codon stop : codon commandant l’arrêt de la synthèse du polypeptide.
Universel : valable pour tous les êtres vivants.
(code génétique) Non ambigu : un codon donné ne désigne qu’un seul acide aminé.
(code génétique) Dégénéré ou Redondant : dans le code génétique, plusieurs codons désignent le même acide aminé.
(allèle) Dominant : allèle qui, même en un seul exemplaire, détermine le phénotype de l’individu.
(allèle) Récessif : allèle qui ne détermine le phénotype de l’individu que s’il est présent en deux exemplaires.
(caractère) Multigénique : qui dépend de l’expression de plusieurs gènes. C’est le cas le plus fréquent.
Gènes de prédisposition : gènes qui ne sont pas responsables directement d’un phénotype mais qui codent des protéines
dont l’activité influence la réalisation du phénotype.
1
/
2
100%