ADN

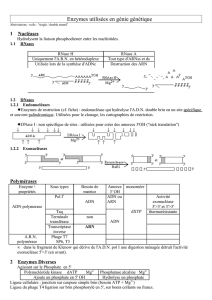
Transfert de l'information génétique
PROTEINES ACTIVES
MODIFICATIONS
POST TRADUCTIONNELLES
TRADUCTION
PROTEINES
REPLICATION
ADN
TRANSCRIPTION
ARN ARN
Réplication de l’ADN
Eléments nécessaires
Mécanisme
Caractères
Biosynthèse d’une double chaîne d'ADN
identique au double brin d'ADN parental
PACES UE1-3 Biologie Moléculaire
Marc DENIS
1/27

Réplication : éléments nécessaires
ADN qui sera répliqué (copié)
=ADN matrice sous forme simple brin
Polynucléotide qui sera prolongé =ARN amorce
Enzymes : complexe "réplicase"
principales : ADN polymérases ADN dépend.
autres :
primase
gyrase (topo-isomérase)
hélicase
ligase
RNase (exonucléase 5' →3')
Cosubstrats (ou coenzymes)
= désoxyribonucléosides tri P
dATP, dGTP, dCTP, dTTP
l'élément transféré est le reste
Nucléotidyl
Protéines auxiliaires spécifiques
•protéines de reconnaissance
•protéines de stabilisation de l'ADN monobrin
•protéines de la fourche de réplication
PACES UE1-3 Biologie Moléculaire
Marc DENIS
2/27

ADN – polymérases
Différentes chez procaryotes et eucaryotes
Caractères
toutes nécessitent un modèle-support simple brin
une amorce avec un group
t
3’ OH libre
toutes assurent la synthèse de la chaîne dans le sens 5’ vers 3’
certaines ont une activité de relecture - correction
5’ 3’
Amorce ARN
5’
ADN polymérase
3’
ADN nouvellement synthétisé
5’
Topoisomérases
Topoisomérase II
Caractères
– Agissent sur l’enroulement ou le désenroulement
– 2 possibilités
– Coupure d’un brin - Coupure des 2 brins
Topoisomérase I
PACES UE1-3 Biologie Moléculaire
Marc DENIS
3/27

Double hélice
3 super tours
Coupure
ADN
2 super tours
Liaison
Topo I
Passage
du brin 3’
sous le
monobrin
intact Dissociat°
Topo I
Double hélice
Intermédiaire
ADN-enzyme
Topoisomérase I
suite
Topoisomérase II
Coupure
du
double
brin
Passage
par
devant
et
ligation
Réplication : mécanisme
Energie dépendant : ATP
Importance des interactions ADN-protéines
7 étapes
Reconnaissance de(s)
l'origine(s) de la
réplication, intervention
de protéines d’initiation
Protéines
d’initiation
origine
PACES UE1-3 Biologie Moléculaire
Marc DENIS
4/27

Stabilisation des
simples brins par des
protéines spécifiques
protéines SSB
Synthèse des amorces
d’ARN par la primase
polymérase
ARN
primase
Détorsion et séparation
des brins par la gyrase et
l’hélicase
gyrase
hélicase
Addition des
désoxyribonucléosides =
polymérisation de l'ADN
Hydrolyse des
amorces ARN et remplac
t
par des dXTP
RNase
Soudure et
assemblage des fragments
ligase
PACES UE1-3 Biologie Moléculaire
Marc DENIS
5/27
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
1
/
27
100%