Travaux dirigés de Biologie Moléculaire 5

Travaux dirigés de
Biologie Moléculaire
5
(semaine 7)

Exercice 14 : Phage λ: microscopie électronique


Bactériophage T5 attaquant une bactérie E. coli.
En haut, à dte : bactériophage T4 injectant son ADN dans E. coli.

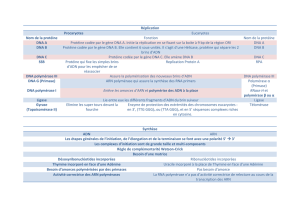
Les ADN POLYM
Les ADN POLYMÉ
ÉRASES
RASES
n
né
écessitent
cessitent
¾
¾un brin d'ADN matrice
un brin d'ADN matrice
¾
¾une amorce
une amorce oligonucl
oligonuclé
éotidique
otidique
r
ré
éalisent
alisent
¾
¾une synth
une synthè
èse du brin nouveau 5
se du brin nouveau 5‘
‘vers 3'
vers 3'
5’ 3’
3’ 5’
amorce
matrice
nouveau brin
3’
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
1
/
23
100%