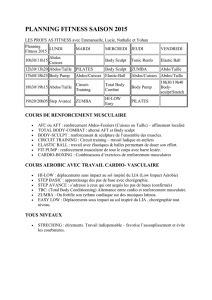
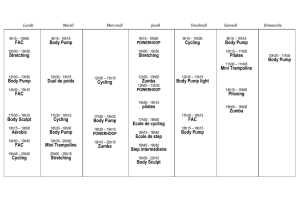
Evolution de la masse corporelle chez les mammifères : Patrons

Evolution de la masse corporelle
chez les mammifères :
Patrons & Processus
benoit.nabholz@univ-montp2.fr

I. Comme étudier l’évolution de la masse corporelle :
a) Les fossiles
b) Les reconstructions phylogénétiques
II. Analyses de travaux passées
a) Nabholz & Lartillot in prep
b) Romiguier et al. 2012
c) Smith et al. 2010
III. Implication pour la loi de Cope
PLAN

Les données fossiles
●Extrapolation de la masse par régression linaire1 :
masse = β (longueur m1 x larguer m1) + α [Carnivora, Insectivora,
Primates, Rodentia]
masse = β longueur m1 + α [Artiodactyla, Perrisodactyla]
●Utilisation de régressions allométriques (log-log) spécifique a une
famille ou un ordre2, mais aussi restreinte à la même gamme de taille3
●Utilisation de données post-craniales, si disponible, est conseillé3
1 Alroy 1998 ; 2 Smith et al. 2010 ; 3 Damuth & McFaden 1990

Les données fossiles
●Exemple chez les carnivores (n=72)1 :
1 Van Valkenburgh 1990 in Damuth J, MacFadden BJ
Groupe Pente R %PE
Total 2.97 0.83 97
Canidae 1.82 0.87 27
Ursidae 0.49 0.43 46
Mustelidae 3.48 0.93 45
Felidae 3.05 0.95 28
PE (Predicted Error) = (Observed – Predicted) / Predicted

● Utilise un modèle de « mouvement brownien » :
I. La moyenne des mouvements entre la racine et les taxons
terminaux est nulle
II. La variance des mouvements (σ²) est constante et
proportionnelle au longueur de branches
III. La direction du mouvements est indépendantes entre lignées
et ne dépend pas non plus de l'état ancestral
Méthodes phylogénétiques
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
1
/
42
100%