les acides aminés et les protéines

1
LES ACIDES AMINÉS ET LES
PROTÉINES
A- LES PROPRIÉTÉS ACIDO-BASIQUES DES ACIDES AMINÉS ET DES PEPTIDES
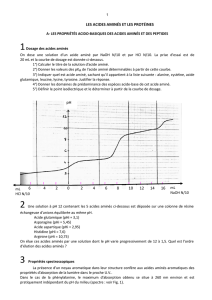
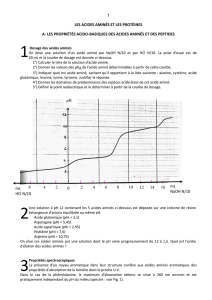
1 Dosage des acides aminés
On dose une solution d’un acide aminé par NaOH N/10 et par HCl N/10. La prise d'essai est de
20 mL et la courbe de dosage est donnée ci-dessous.
1°) Calculer le titre de la solution d'acide aminé.
2°) Donner les valeurs des pKA de l'acide aminé déterminables à partir de cette courbe.
3°) Indiquer quel est acide aminé, sachant qu'il appartient à la liste suivante : alanine, cystéine, acide
glutamique, leucine, lysine, tyrosine. Justifier la réponse.
4°) Donner les domaines de prédominance des espèces acide-base de cet acide aminé.
5°) Définir le point isoélectrique et le déterminer à partir de la courbe de dosage.
2 Une solution à pH 12 contenant les 5 acides aminés ci-dessous est déposée sur une colonne de résine
échangeuse d'anions équilibrée au même pH.
Acide glutamique (pHi = 3,1)
Asparagine (pHi = 5,45)
Acide aspartique (pHi = 2,95)
Histidine (pHi = 7,6)
Arginine (pHi = 10,75)
On élue ces acides aminés par une solution dont le pH varie progressivement de 12 à 1,5. Quel est l'ordre
d'élution des acides aminés ?
3 Propriétés spectroscopiques
La présence d'un noyau aromatique dans leur structure confère aux acides aminés aromatiques des
propriétés d'absorption de la lumière dans le proche U.V..
Dans le cas de la phénylalanine, le maximum d'absorption obtenu se situe à 260 nm environ et est
mL
HCl N/10
mL
NaOH N/10
pH
2
2
4
4
4
4
6
4
6
4
8
4
10
4
12
14
16
0

2
pratiquement indépendant du pH du milieu (spectre : voir Fig. 1).
Dans le cas de la tyrosine, le maximum d'absorption se situe à une longueur d'onde différente suivant que la
solution de tyrosine (75 mg.L-1) est préparée dans l'eau ou dans un milieu NaOH
0,1 M (spectres : voir Fig. 2).
1°) Écrire les domaines de prédominance des espèces acide-base de la tyrosine et de la
phénylalanine. Essayer d'interpréter l'observation faite ci-dessus.
Cette variation de l'absorption en fonction du pH peut être utilisée pour déterminer
expérimentalement la valeur du pKA de la chaîne latérale de la tyrosine.
On prépare pour cela 2 solutions de tyrosine à la concentration de 75 mg.L-1 en milieu tampon bicarbonate
l'une à pH 9,5 et l'autre à pH 10,5 (ces valeurs encadrent le pKA de la chaîne latérale de la tyrosine). Les
spectres d'absorption de ces deux solutions sont présentés sur la Fig. 2.
2°) Indiquer quelles sont les espèces acide-base de la tyrosine présentes dans chacune des 2
solutions à une concentration ≥ 10% du total.
3°) Sachant que, lorsque deux espèces, ici, Φ-OH et Φ-O-, présentes en solution absorbent la lumière
simultanément à une longueur d'onde donnée, l’absorbance lue s'exprime ainsi
A = εΦ-OH . cΦ-OH + εΦ-O- . cΦ-O- .
à quelle longueur d'onde sera-t-il judicieux de se placer pour calculer le plus simplement possible, à partir de
l’absorbance lue, les concentrations de Φ-OH et Φ-O- présentes dans chacune des 2
solutions ?
4°) Calculer ces concentrations de Φ-OH et Φ-O- et en déduire la valeur de pKA de la chaîne latérale
de la tyrosine.
4 Le diagramme ci-dessous représente la courbe de neutralisation du trichlorate (forme la plus acide) de
l'hexapeptide (A) ou (B).
(A) Phe - Gly - Lys - Phe - Gly - Lys
(B) Phe - Gly - Arg - Phe - Gly - Arg
Fig. 1
Fig. 2
A (U.A.)
A(U.A.)
0,2
0,4
0,6
0,8
1,0
0,2
0,4
0,6
0,8
1,0
240
260
280
270
290
250
(a) Tyrosine dans la soude
(b) Tyrosine dans l'eau
(c) Tyrosine à pH 10,5
(d) Tyrosine à pH 9,5
O
H
C
H
2
C
H
C
O
O
-
N
H
3
+
pK = 2,2
pK = 9,1
pK = 10,1
a
c
b
a
b
d
c
d
λ
λ

3
Données : pKA des groupements des acido-basiques des chaînes latérales des acides aminés :
Asp
Glu
His
Tyr
Lys
Cys
Arg
3,9
4,1
6
10,1
10,5
8,3
12,5
a) Relever les pKA et les attribuer aux différentes fonctions acido-basiques.
b) Relever et justifier les nombres d'équivalents versés pour neutraliser les fonctions.
c) S'agit-il de l'hexapeptide (A) ou (B) ?
5 Soit le peptide P : Asp-Leu-Lys-Met-Trp-Cys-Ser-Val
1°) Comment migrera-t-il lors d’une électrophorèse à pH 7 ? à pH 12 ?. Justifiez votre réponse en
précisant quels sont les résidus d’acides aminés chargés dans les deux cas.
2°) Peut-on détecter ce peptide par une mesure d’absorbance ? Pourquoi ?
B- DÉTERMINATION DES STRUCTURES
6 Méthode du finger-print
L'analyse d'une protéine naturelle purifiée donne les résultats suivants :
* Le traitement court par le PITC, permet de caractériser le PTH-Ala.
* Une incubation brève avec une carboxypeptidase détache une leucine.
* Après hydrolyse trypsique, les peptides libérés sont séparés par la méthode du finger-print
(électrophorèse à pH 6,4 suivie d'une chromatographie dans un sens perpendiculaire, réalisée à l'aide
d'une phase mobile organique). Après révélation, on obtient 4 taches numérotées 1,2,3 et 4
correspondant à 4 peptides différents.
Équivalents de
OH- ajoutés
pH
1
2
3
4
0
2
4
6
8
10
12

4
* Après élution de ces taches et hydrolyse totale et détermination de la composition en acides
aminés, on obtient les résultats suivants :
- peptide "a" : 3 Ser, 4 Leu, 1 Ala, 1 Met, 1 Tyr
- peptide "b" : 5 His, 2 Ser, 1 Arg, 1 Thr, 1 Pro, 1 Leu
- peptide "c" : 1 Ala, 5 Glu, 2 Cys, 4 Asp, 1 Lys, 1 Leu
- peptide "d" : 1 Tyr, 1 Arg, 2 Thr, 3 Val, 1 Phe
1°) Quel est l'emplacement de ces peptides sur le finger-print ?
2°) Parmi ces peptides, l'un a un pH isoélectrique de 6,4 et un autre de 4,5. De quels peptides s'agit-il ?
3°) a) Quel est le peptide contenant le résidu N-terminal ?
b) Quel est le peptide contenant le résidu C-terminal ?
7 Soit un octapeptide P dont on veut déterminer la structure primaire. Pour cela, on réalise un certain
nombre d’expériences. Pour chacune d’entre elles, il est demandé de rappeler l’action des réactifs utilisés et
d’en tirer le maximum de conclusions.
1°) L’action brève de l’aminopeptidase libère un Asp. Une hydrolyse HCl 6N à chaud sur le peptide
restant permet d’obtenir un mélange des acides aminés suivants en proportions stoechiométriques : Arg,
Gly, Glu, Val, Leu, Lys.
2°) La chymotrypsine est sans action sur ce peptide P.
3°) L’action de la trypsine sur P permet de libérer deux tripeptides A et B et un dipeptide C. L’action
courte de l’aminopeptidase sur l’hydrolysat trypsique permet de libérer Asp, Gly et Leu.
4°) Une hydrolyse chlorhydrique sur C fournit un mélange de Asp et Arg.
5°) La même expérience sur A ne permet d’identifier que Gly et Glu.
6°) Le peptide A mige vers l’anode lors d’une électrophorèse à pH = 7.
Quelle structure de P pouvez-vous déduire de l’ensemble de ces résultats ?
8 Soit un peptide P dont on veut déterminer la structure :
1°) On fait agir l’aminopeptidase sur ce peptide pendant un temps très court : elle détache la valine.
On hydrolyse le peptide restant en milieu acide. Les acides aminés obtenus sont analysés et dosés par
chromatographie. On obtient : Ala, Asp, Cystine, Gly et Tyr . Tous les acides aminés sont à la même
concentration sauf la tyrosine qui est à une concentration double et la cystine qui est à la concentration
moitié de celle des autres. Par ailleurs, l'action de la carboxypeptidase pendant un temps très court libère
Gly. Que peut-on déduire de ces résultats ?
2°) On traite le peptide par la chymotrypsine puis l’aminopeptidase pendant un temps très court :
celle-ci détache l’alanine et la valine.
3°) Le séquençage d'Edman, appliqué au peptide P, permet de mettre en évidence et dans l'ordre,
Val, Asp puis Tyr. Quelle est la séquence du peptide initial ?
3
4
2
1
(dépôt
)
chromatographie
- électrophorèse à pH 6,4 +

5
9 Un peptide est composé de 30 résidus d'acides aminés. Sa composition, déterminée après hydrolyse
acide, est la suivante :
Gly 3 Ser 2 Phe 2 Glu 3
Ala 3 Thr 1 Pro 2 Lys 1
Val 3 Cys 1 Tyr 1 Arg 2
Leu 3 Ile 1 Met 1
Traité par la trypsine, il donne 4 peptides A, B, C et D dont les séquences sont déterminées
indépendamment.
1°) La composition en acides aminés du peptide A est la suivante : 2 Ala, Tyr, Ile et Lys. Quelle est la
nature de l'extrémité C-terminale de ce peptide ?
Un traitement au PITC donne un PTH-Ile et l'action de la chymotrypsine libère deux peptides : un dipeptide et
un tripeptide.
- À l'aide de ces résultats, déterminer la ou les séquences du peptide A.
- Quelle autre méthode permettrait de résoudre entièrement cette séquence ?
2°) Les séquences de peptides B, C et D sont obtenues partiellement par la méthode récurrente
d'Edman. Les résultats obtenus sont les suivants :
B (10 aa) : Met - Ala- Ser - (4) - Leu - Thr - Arg
C (12 aa) : Gly - Trp - Leu - (4) - Pro - Gly - Phe - Ser - Arg
D ( 3 aa) : Val - Glu - Leu
L'action de la chymotrypsine sur le peptide initial libère 5 peptides dont les séquences sont les suivantes :
E (8 aa) : Leu - Pro - Gln - Cys - Val - Pro - Gly - Phe
F (7 aa) : Val - Glu - Leu - Thr - Arg - Ile - Tyr
G (5 aa) : Met - Ala - Ser - Gly - Phe
H (5 aa) : Ala - Ala - Lys - Gly - Trp
I (5 aa) : Ser - Arg - Val - Glu – Leu
- Quelle est la séquence initiale ?
10 Un octapeptide I absorbe la lumière ultraviolette à 280 nm. L'hydrolyse acide donne Ala, Met, Leu,
Arg, Val, Lys, Gly. L'action de la carboxypeptidase donne Val. L'action courte de l'aminopeptidase donne Leu.
L'hydrolyse trypsique donne 2 peptides II et III et un acide aminé libre.
L'analyse du peptide II donne les résultats suivants :
Ce peptide n'absorbe pas à 280 nm. L'hydrolyse acide totale donne Lys, Leu, Ala.
1°) Quelle est la séquence du peptide II ?
L'analyse du peptide III donne les résultats suivants :
Ce peptide absorbe à 280 nm. L'action du bromure de cyanogène donne 2 peptides IIIa et IIIb. Le peptide IIIa
absorbe à 280 nm. L'hydrolyse acide de IIIb donne Arg et Gly.
2°) Quelle est la séquence du peptide III ?
3°) Quelle est la séquence du peptide I ?
11 (examen juin 2002) Pour déterminer la structure primaire d’un pentapeptide P, on réalise les
expériences suivantes. Pour chacune d’entre elles, vous préciserez clairement le rôle de chacun des réactifs
utilisés et vous indiquerez les conclusions que l’on peut tirer du résultat obtenu :
1°) L’hydrolyse acide totale aboutit au mélange d’acides aminés suivant : Arg, Glu, Leu, Met à la même
concentration. De plus, l’action ménagée du réactif d’Edman libère un PTH-Leu.
2°) L’action de la trypsine sur ce peptide permet la formation d’un dipeptide P1 et d’un tripeptide P2. De plus,
l’action ménagée du réactif d’Edman sur P1 libère un PTH-Leu.
3°) L’action du bromure de cyanogène sur P2 libère un acide aminé et un dipeptide P3 qui absorbe la lumière
à 280 nm. Quels sont les acides aminés qui absorbent la lumière à 280 nm ? Comment les appelle-t-on ?
4°) Quelle est la séquence du peptide P ? Quelle l’expérience proposez-vous pour lever l’ambiguïté ?
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
1
/
11
100%