Liste des figures

Liste des figures
Liste des figures
Figure 1-1. Anatomie du cœur selon [Netter-69] : Vue ouverte du cœur selon un plan de coupe ........................ 17
Figure 1-2. Représentation schématique des ventricules du coeur et des repères couramment utilisés. ............... 17
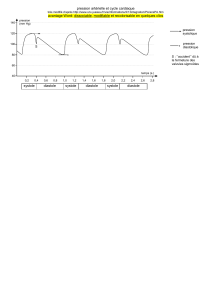
Figure 1-3. Illustration d’un électrocardiogramme ............................................................................................... 18
Figure 1-4. La vascularisation du cœur................................................................................................................. 19
Figure 1-5. Définition de l’orientation des plans de coupe du cœur en petit axe et en grand axe [Cerqueira-02]. 21
Figure 1-6. Dénominations préconisées des segments cardiaques pour les 3 niveaux de coupe en petit axe
[Cerqueira-02]............................................................................................................................................ 21
Figure 1-7. Exemple d’angiographie des coronaires............................................................................................. 22
Figure 1-8. Coupe du cœur obtenue en tomodensitométrie (Philips system, CHU L. PRADEL Lyon)
résolution= 0.44 x 0.44 mm, épaisseur de coupe=0.67mm....................................................................... 23
Figure 1-9. Image ultrasonore cardiaque de mode B (vue 4 cavités).................................................................... 24
Figure 1-10. Acquisition d’images par RM cardiaques synchronisée à l’ECG : t représente le décalage entre le
pic de l’onde R et le début de l’enregistrement des données ; t représente la résolution temporelle de la
série dynamique
delai
offset
......................................................................................................................................... 27
Figure 1-11. Exemple d’images par RM de marquage tissulaire. (a) télé-diastole, (b) télé-systole...................... 28
Figure 1-12. Étude de l’anatomie et des fonctions cardio-vasculaires en imagerie .............................................. 29
Figure 2-1. Le fantôme 4D MCAT où les organes sont définis à l’aide de primitives géométriques (issue de
[Segars-01a]).............................................................................................................................................. 37
Figure 2-2. Coupe en grand axe du cœur battant du modèle 4D MCAT en fin de diastole et en fin de systole (issu
de [Segars-01a]). Les ventricules et les oreillettes sont définis par deux ellipsoïdes, un pour l'intérieur et
un pour la paroi externe. Pour simuler la contraction radiale et l'épaississement des parois pendant la
systole, l’axe principal et l’axe secondaire (y et z) définissant l’ellipsoïde intérieure sont modifiés. Pour
simuler la contraction longitudinale, le plan des valves (plan ⊥ passant par l’axe z) entre les oreillettes et
les ventricules est abaissé. Le mouvement de torsion est modélisé en tournant le modèle autour du grand
axe du VG (axe (x) dans l'image ci-dessus) dans le sens horaire au niveau de la base et dans le sens
inverse au niveau de l’apex........................................................................................................................ 38
Figure 2-3. Version initiale du modèle NCAT (issu de [Segars-99]. À gauche, la phase diastolique et à droite la
phase systolique de ce modèle................................................................................................................... 39
Figure 2-4. Génération des modèles d’oreillettes au cours du cycle cardiaque (issu de [Segars-01a]). (a) Modèles
d'oreillettes adaptés en télé-diastole. (b) Modèles d'oreillettes (a) avec les modèles de ventricule en télé-
systole. La translation vers le bas des modèles d'oreillettes en prenant en compte le mouvement de la
contraction longitudinale des ventricules est illustrée en (c). Les modèles sont alors mis à l’échelle
longitudinalement en gardant l'apex des oreillettes constant au cours du cycle. Les oreillettes sont mises à
l’échelle en 2D afin qu'elles aient un volume défini (d). ........................................................................... 39
Figure 2-5. Le fantôme NCAT complet. (a) fin d’expiration. (b) fin d’inspiration. (c) points de contrôle des
surfaces rationnelles pour la simulation du mouvement respiratoire (issu de [Segars-01a, Segars-02a]). 40
Figure 2-6. Version féminine du modèle de thorax à base de superquadriques [Peter-99]................................... 41
Figure 2-7. Modèle déformable des deux ventricules basé sur des primitives géométriques superellipsoïdes
[Park-05].................................................................................................................................................... 42
Figure 2-8. (a) Modèle de l’épicarde d'un coeur de chien obtenu à partir d’images en tomodensitométrie à rayons
X à l’instant de fin de diastole. (b) les quatre cavités et l’abouchement des gros vaisseaux du coeur
humain obtenu à partir d’Images par RM en fin de diastole (issu de [Wierzbicki-04a])........................... 43
Figure 2-9. (à gauche) les surfaces cardiaques triangulées. (à droite) Modèle résultant avec le réseau des
coronaires (issu de [Lorenz-05])................................................................................................................ 43
Figure 2-10. (a) Modèle 3D surfacique des quatre cavités cardiaques de Lötjönen et al. en fin de diastole
[Lötjönen-04]. (b) Modèle moyen spatio-temporel [Delhay-05] (vue en fin de diastole) ......................... 44
Figure 2-11. Coupes anatomiques axiales du cœur de l’homme (gauche) et de la femme (droite) issues du VHP
................................................................................................................................................................... 45
Figure 2-12. Modèle de cœur féminin issu des données du VHP (issu de [Hurmusiadis-05]).............................. 45
Figure 2-13. (a) Vues du modèle de cœur basé sur l’imagerie du VHP développé par Sachse et al. [Sachse-04].
(b) Orientation des fibres incluse dans ce modèle ..................................................................................... 45
Figure 2-14. Position des marqueurs implantés chirurgicalement dans la paroi du ventricule gauche d’un chien
dans [Arts-92]............................................................................................................................................ 47
8

Liste des figures
Figure 2-15. (a) Modèle d’ellipsoïdes du ventricule gauche du cœur. (b) Modèle 3D du VG à six phases du cycle
cardiaque (issu de [Waks-96]) ................................................................................................................... 47
Figure 2-16. (a) Modèle maillé en tétraèdres du VG+VD+épicarde de Pham et al. (b) le modèle avec VG et VD
individualisés. (c) exemple de la segmentation du muscle cardiaque au cours du cycle (t=0, 80, 160 et 240
ms), vue 2-D à mi-hauteur du VG [Pham-02]. .......................................................................................... 48
Figure 2-17. (a) Modèle de VG de forme ellipsoïdale. (b) Modèle des ventricules gauche et droit (issu
de [Kerckhoffs-03a]).................................................................................................................................. 49
Figure 2-18. Stratégie proposée pour estimer les déformations du VG dans [Papademetris-02].......................... 50
Figure 2-19. Ajustement du modèle bicavité de Sermesant et al. à des données patient en IRM (issu de
http://www-sop.inria.fr/asclepios/personnel/Maxime.Sermesant/gallery.php).......................................... 51
Figure 2-20. Vue photographique du fantôme physique présenté dans [Wierzbicki-03] avec marqueurs............ 52
Figure 3-1. Principe général de construction du modèle de cœur battant ............................................................. 58
Figure 3-2. Visualisation 3D des données statiques acquises. (a) En haut, visualisation du volume de données
constitué de l’empilement des coupes transverses avec interpolation linéaire. En Bas, 3 plans orthogonaux
(transverse, sagittal et coronal). (b) Situation des plans d’acquisition par rapport au cœur....................... 60
Figure 3-3. Géométrie d’acquisition des données anatomiques et deux coupes au niveau inférieur (coupe 16) et
au médian de la pile de coupes (coupe 60) ................................................................................................ 61
Figure 3-4. (a) Coupe transverse du thorax et du cœur de la série anatomique statique. (b) Coupe de la série
dynamique en milieu de diastole. (c) Superposition des deux images....................................................... 61
Figure 3-5. Géométrie d’acquisition des séries dynamiques et images acquises en télédiastole à un niveau
inférieure, à un niveau médian et à un niveau supérieure, respectivement. ............................................... 62
Figure 3-6. 8 niveau coupe des séries dynamiques à cinq phases du cycle cardiaque. Phase 1 en fin de
diastole, phase 9 et phase 11 au cours de la systole, phase 17 en fin de systole et phase 25 en milieu de
diastole.
ième
...................................................................................................................................................... 63
Figure 3-7. Données IRM de marquage tissulaire acquises. A gauche, coupe en milieu de diastole. A droite,
coupe en milieu de systole. ........................................................................................................................ 63
Figure 3-8. Visualisation 3D illustrant la couverture partielle du cœur et des gros vaisseaux dans le premier jeu
de données acquis ...................................................................................................................................... 64
Figure 3-9. (a) Reconstruction d’un volume à partir des données dynamiques acquises en petit axe avec une
faible résolution spatiale selon l’axe longitudinal du cœur (17mm d’espacement entre coupes), (b) Report
d’images en Ciné-IRM dans le référentiel statique. Des défauts de mise en correspondance des structures
anatomiques sont identifiés par des flèches dans l’image.......................................................................... 65
Figure 3-10. Pile de coupes acquises en IRM dynamique en axe transverse illustrant la forte anisotropie spatiale.
La résolution dans les plans de coupe est de 1.25 x 1.25 mm² et la distance inter-coupes est de 7 mm.... 65
Figure 3-11. les différents noyaux d’interpolation expérimentés sur les données acquises .................................. 67
Figure 3-12. (a) Interface du logiciel de segmentation «ManSegTool» et contours dessinés sur une coupe
médiane du cœur. (b) Empilement des contours extraits en 3D pour le VG, VD, OG et OD.................... 69
Figure 3-13. A gauche : une cellule de Voronoï. Au milieu : diagramme de Voronoï d’un ensemble de n points.
A droite : diagramme de Voronoï et triangulation de Delaunay en traits pleins........................................ 70
Figure 3-14. Les différentes étapes de la construction du modèle statique. (a) Extraction des contours. (b)
Reconstruction 3D des surfaces des différentes structures cardiaques. ..................................................... 71
Figure 3-15. Construction du réseau des coronaires. (a) Lignes centrales du réseau. (b) Modèle binaire. ........... 72
Figure 3-16. Rehaussement des images par RM anatomiques en vue de l’extraction des coronaires. A gauche :
une coupe par RM transverse du cœur. A droite : la même image après filtrage multi-échelle................. 75
Figure 3-17. Extraction de l’arbre coronaire dans les images IRM 3D filtrées. A droite : extraction du tronc
coronaire droit, à gauche : extraction du tronc coronaire gauche. ............................................................. 75
Figure 3-18. Comparaison du modèle des coronaires extrait sans et avec filtrage multi-échelle dans les mêmes
images 3D IRM. (a) le modèle 3D binaire de l’arbre coronaire sans filtrage multi-échelle préalable. (b) le
modèle 3D binaire de l’arbre coronaire obtenu après filtrage multi-échelle.............................................. 76
Figure 3-19. Modèle anatomique du cœur et des coronaires................................................................................. 77
Figure 3-20. Notre modèle (à droite) en regard de vues d’artiste (à gauche) telles qu’on peut les rencontrer dans
des planches anatomiques (http://www.chups.jussieu.fr/polys/cardio/anat/index.html)............................ 77
Figure 3-21. Positionnement du modèle anatomique dans les coupes IRM natives. (a) Modèle 3D, (b) Contours
d’intersection du modèle 3D avec trois plans de coupes orthogonaux ...................................................... 78
Figure 3-22. (a-b) à gauche, le modèle de VG obtenu sans remaillage, à droite le même modèle après remaillage.
(c) Triangulations respectivement sans et avec remaillage........................................................................ 79
Figure 3-23. Modèle statique de cœur final avec identification des différentes structures segmentées (en haut).
Vue du coté gauche et du coté droit du cœur anatomique (en bas)............................................................ 80
Figure 3-24. Changement de volume du ventricule gauche au cours du cycle cardiaque. Le volume en fin de
systole est environ la moitié du volume en fin de diastole......................................................................... 82
9

Liste des figures
Figure 3-25. Principaux composants d’une méthode de recalage entre deux images qui seront ici des images
consécutives d’une séquence dynamique................................................................................................... 83
Figure 3-26. Recalages successifs réalisés au cours du cycle cardiaque. Les t indiquent les instants des phases
cardiaques, les T - représentent les transformations entre deux instants.i
j k .................................................. 84
Figure 3-27. Principe des déformations de forme libre FFD. À gauche : grille de points de contrôle et objet non
déformé. À droite : le déplacement de certains points de contrôle de la grille entraîne la déformation de
l’objet inclus. ............................................................................................................................................. 86
Figure 3-28. Détermination des coordonnées locales d’un point x....................................................................... 87
Figure 3-29. Représentation des fonctions B-Splines pour les quatre premiers ordres (ordre=degré+1).............. 88
Figure 3-30. Principe de la méthode de recalage non rigide basée sur les déformations de forme libre et la double
pyramide portant sur l’image (à gauche) et la transformation de forme libre (à droite)............................ 95
Figure 3-31. Illustration du champ de mouvement estimé en 3D (coupe 2D à mi-VG) à trois instants du cycle
cardiaque.................................................................................................................................................... 96
Figure 3-32. Illustration du champ de mouvement 3D estimé dans la direction longitudinale sur une coupe
coronale 2D 4 cavités. On agrandit le champ de mouvement au niveau du VG et de l’OD. ..................... 97
Figure 3-33. Variations des formes des deux cœurs en milieu de diastole et de systole et courbes de volumes
associées. ................................................................................................................................................... 99
Figure 3-34. Illustration des variations de formes de l’ensemble des structures cardiaques aux différents instants
du cycle cardiaque (1 instant / 2 de la séquence complète), obtenues à partir des champs de déplacement
calculés. ................................................................................................................................................... 101
Figure 4-1. Anatomie du Thorax......................................................................................................................... 103
Figure 4-2. Évolution des volumes des poumons au cours de la respiration....................................................... 104
Figure 4-3. Déformation du diaphragme pendant la respiration. L’image présente une radiographie des poumons
en fin d’expiration forcée et au maximum d’inspiration.......................................................................... 105
Figure 4-4. Variation du volume des poumons pendant le cycle respiratoire ([Segars-01a]) ............................. 106
Figure 4-5. Principe de construction du modèle de thorax respirant avec intégration du cœur battant............... 107
Figure 4-6. Trois coupes extraites des données IRM thoraciques à l’instant de fin d’inspiration....................... 108
Figure 4-7. Étapes de construction du modèle anatomique 3D de thorax. (a) exemple de contours segmentés, (b)
exemple de surface 3D reconstruites et (3) modèle résultant de l’assemblage des différents structures. 109
Figure 4-8. Remaillage des structures................................................................................................................. 109
Figure 4-9. Données IRM après interpolation en 3D avec un noyau B-splines (degré 3) à l’instant de fin
d’inspiration............................................................................................................................................. 110
Figure 4-10. Application des transformations calculées aux modèles 3D des poumons (droit en bleu) et gauche
en rouge)) issus de la segmentation (b) entre (a) l’instant intermédiaire et l’instant de fin d’inspiration et
(c) l’instant intermédiaire et l’instant de fin d’expiration ........................................................................ 111
Figure 4-11. Courbe de la variation du volume des poumons au cours de la respiration. Les 3 instants pour
lesquels on dispose d’images sont indiqués............................................................................................. 111
Figure 4-12. Synchronisation des cycles respiratoire et cardiaque ..................................................................... 112
Figure 4-13. Déplacement du cœur et déformations des poumons pendant l’inspiration ................................... 113
Figure 4-14. Déplacement du cœur et déformations des poumons pendant les cycles respiratoire et cardiaque 113
Figure 4-15. Volumes des structures représentatives au cours des cycles respiratoire et cardiaque................... 114
Figure 5-1. Comparaison structurelle entre notre modèle et les modèles de Wierzbicki et al. [Wierzbicki-04] (a)
et Lorenz et al. [Lorenz-05] (b)............................................................................................................... 117
Figure 5-2. Volume des structures cardiaques. A gauche : volumes issus des contours extraits. Au milieu :
volume des structures remaillées. A droite : volumes des structures remaillées et lissées ...................... 118
Figure 5-3. Superposition des contours initiaux et après re-maillage sur les données anatomiques ................... 118
Figure 5-4. Cartographies d’erreur entre les surfaces issues des contours extraits et le modèle anatomique 3D,
calculées par le logiciel Metro. A gauche le péricarde et à droite le VG................................................. 119
Figure 5-5. Courbes de volume au cours du cycle cardiaque. Comparaison entre la segmentation manuelle des
structures et les structures obtenues par application du mouvement calculé ........................................... 121
Figure 5-6. Superposition de la trace du modèle dynamique sur 3 plans de coupe et à 3 instants du cycle
cardiaque.................................................................................................................................................. 122
Figure 5-7.Cartographies d’erreur pour le modèle dynamique calculé comparé à la segmentation (milieu de
systole)..................................................................................................................................................... 123
Figure 5-8 . Superposition sur une coupe IRM 4- cavités du cœur des structures segmentées manuellement et des
structures déformées obtenues par l’application du mouvement calculé en milieu de systole (mouvement
le plus important)..................................................................................................................................... 124
Figure 5-9 . A gauche, champs de mouvement estimé par notre méthode en fin de diastole où l’on constate un
mouvement de faible amplitude. Au milieu, mouvement de contraction du VG en milieu de la systole. A
10

Liste des figures
droite, mouvement d’expansion du VG en fin de systole. En bas, agrandissement du champ dans la région
antérieure du myocarde............................................................................................................................ 125
Figure 5-10 . A gauche, mouvement calculé par InTag. A droite, mouvement calculé par notre méthode en
milieu de la systole .................................................................................................................................. 125
Figure 6-1. Structures anatomiques associées aux labels (à gauche) et coupe transverse d’une image 3D
labellisée de thorax (à droite)................................................................................................................... 127
Figure 6-2. Valeurs de T1 et T2 utilisées lors de la simulation du Thorax pour un B0 de 1.5T ...................... 129
Figure 6-3. (a) Image par RM en pondération T1/T2. (b) Image par RM en pondération .............................. 130
Figure 6-4. Coupe IRM simulée à 117 mm d’élévation à différents instants des cycles cardiaque et respiratoire
................................................................................................................................................................. 130
Figure 6-5. Valeurs d’intensité relative moyenne des différentes structures exprimées en pourcentage ........... 131
Figure 6-6. Temps de simulation avec SIMRI sur un P4 3GHz – 2Go de RAM ................................................ 131
Figure 6-7. Valeurs de fixation du Fluor-18 et choix des milieux atténuants utilisés pour la simulation du
Thorax avec SORTEO............................................................................................................................. 134
Figure 6-8. A gauche : Image TEP réelle de transmission. Au milieu : Image TEP de transmission simulée
obtenue par N. Pauna [Pauna-03, Pauna-04]. A droite : Image TEP de transmission simulée que nous
avons obtenue pour un cas sain................................................................................................................ 134
Figure 6-9. A gauche : Image TEP d’émission simulée obtenue par N. Pauna [Pauna-03, Pauna-04]. A droite :
Image TEP d’émission simulée que nous avons obtenue ........................................................................ 135
Figure 6-10. A gauche : Image TEP d’émission simulée pour un modèle sain. A droite : Image TEP d’émission
simulée pour un modèle ischémique (captation du F-18 dans le myocarde à 40% de sa valeur initiale). 135
F
igure 6-11. Temps de simulation avec Sorteo sur une grappe de 7 machines P4 3GHz – 2Go de RAM.......... 136
Figure A-1. (a) Mouvement de rotation d’un noyau autour du moment angulaire J appelé spin. (b) Mouvement de
précession d’un noyau atomique autour de l’axe du champ B0............................................................... 141
Figure A-2. Perturbation de l’aimantation M par le champ créé par une impulsion RF. L’angle de bascule est
fonction de la durée de cette impulsion ................................................................................................... 142
Figure B-1. Caractéristiques physiques des principaux émetteurs de positon utilisés en TEP........................... 146
Figure B-2. (a) Annihilation électron/positon, (b) Détection en coïncidence des photons.................................. 148
F
igure B-3. Coïncidences fortuite et diffusée ..................................................................................................... 148
Figure C-1. Plusieurs niveaux de coupes des données cardiaques statiques ....................................................... 150
Figure C-2. Données cardiaques dynamiques à plusieurs instants du CC (1 instant/2) ...................................... 151
Figure C-3. Données thoraciques à 3 instants du cycle respiratoire.................................................................... 152
11
1
/
4
100%