n°18 - Institut de Biosciences et Biotechnologies de Grenoble (BIG)

LETTRE SCIENTIFIQUE
Cytosquelette!1
AT_Chloro
Jacques Joyard
Page 2
!Lettre n° 18 - Mars 2010
iRTSV
Résultats scientifiques
Institut de Recherches en Technologies et Sciences pour le Vivant
Direction des Sciences du Vivant
Immunoprotéomique
Thierry Rabilloud
Page 3
En bref
Revue
Page 4
Moteurs moléculaires
Danielle Gulino
Pages 3 - 4
Cytosquelette
Rajaa Boujemaa-Paterski
Page 1
Pour mieux comprendre les phénomènes
allant de l’acquisition de forme au mouve-
ment cellulaire, l'équipe dirigée par Laurent
Blanchoin du laboratoire de Physiologie Cel-
lulaire Végétale a observé en microscopie à
onde évanescente (méthode hautement réso-
lutive), la génération d’un réseau spécifique
de filaments d’actine en temps réel. In vivo,
c’est l’assemblage coordonné de ces polymè-
res biologiques qui est directement
responsable de la production de force de
poussée qui déforme puis propulse la mem-
brane plasmique et engendre ainsi le dépla-
cement de la cellule.
Ces chercheurs ont utilisé comme outil biomiméti-
que des fibres de verre ou des billes de polystyrène
rendues motiles grâce à l’auto-assemblage sponta-
né d’un réseau d’actine à leur surface (Figures 1 et
2). De façon surprenante, l’observation et la modé-
lisation mathématique extensive des données ex-
périmentales (Figure 2), ont démontré sans ambi-
guïté et contrairement au modèle préétabli, que la
production de force ne résultait pas nécessaire-
ment d’une architecture imposée et contrainte de
ce réseau. Bien au contraire, la génération de force
repose sur des lois physiques simples et fonda-
mentales. Elle est la conséquence de l’allongement
spontané selon une orientation libre des polymères
d’actine issus d’un réseau précurseur. L’augmenta-
tion de matière due à l’assemblage de ce polymère
biologique au sein d’un volume limité et in fine
saturé, engendre la force de poussée de la particule
biomimétique et, par extension, de la membrane
cellulaire.
Figure 2 : L'observation en temps réel en microscopie à
onde évanescente (TIRFM) montre que la coque de fila-
ments d'actine assemblée autour d'une bille de polysty-
rène dans les mêmes conditions qu'en figure 1 finit par
se rompre, propulsant alors l'objet vers l'extérieur (à
t=12 minutes). L'existence de cette première coque cata-
lyse alors l'assemblage des suivantes. On observe alors
que la motilité reconstituée de la bille résulte de la rup-
ture des coques de réseau de filaments polymérisés suc-
cessivement à la surface de l'objet bio-mimétique. Le
code couleur utilisé pour les têtes de flèche correspond à
celui des coques successives de réseau d’actine matériali-
sées dans le dessin modèle.
Un modèle mathématique prenant en considération la
cinétique d'assemblage des filaments d'actine ainsi que
leurs caractéristiques mécaniques rend compte des résul-
tats expérimentaux, notamment que les filaments d'ac-
tine possèdent une orientation libre au sein du réseau.
De l’organisation architecturale du
cytosquelette au mouvement cellulaire
Contact : Rajaa Boujemaa-Paterski
UMR 5168 CEA - CNRS - Inra - UJF
Références
Achard et al. A "primer"-based mechanism un-
derlies branched-actin filament networks forma-
tion and motility. Current Biology, 2010, 20(5):
423-428
Discuté dans News and Views, 2010, 464: 365-366
Figure 1 : Reconstitution in vitro de l'assemblage d'un
réseau de filaments d'actine à la surface d'une fibre de
verre, dans un milieu constitué de protéines purifiées. Ce
réseau, observé simultanément en lumière transmise et en
épifluorescence, génère une force suffisante pour propulser
l'objet bio-mimétique dans ce milieu. Les images révèlent
le réseau polymérisé en 58 minutes. La flèche indique le
sens de déplacement de la baguette de verre.
Voir également lettres scientifiques de l'iRTSV n° 6, n°!8 et n°!12

LETTRE SCIENTIFIQUE
2!AT_Chloro
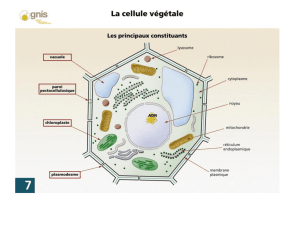
L’essentiel du carbone renouvelable est fixé
par les organismes photosynthétiques grâce à
leurs chloroplastes. Les plastes sont des or-
ganites semi-autonomes présents dans les
plantes terrestres, les algues et quelques pro-
tistes. Site de la photosynthèse, les chloro-
plastes réalisent de nombreuses autres fonc-
tions essentielles comme la synthèse d’acides
gras, de lipides, d’acides aminés, de vitami-
nes, etc. Ces biosynthèses nécessitent le fonc-
tionnement coordonné de trois comparti-
ments!: les thylacoïdes, le stroma et l’enve-
loppe. L’approfondissement des connaissan-
ces des voies métaboliques du chloroplaste et
de leur régulation est considéré comme dé-
terminant dans la perspective d’une
meilleure valorisation de la biomasse végé-
tale. Enfin, comprendre les processus méta-
boliques du chloroplaste est essentiel si l’on
veut analyser l’impact des modifications de
l’environnement sur les plantes. Dans ce
contexte, l’analyse protéomique s’est révélée
être un outil de choix pour faire progresser
de manière significative la connaissance du
chloroplaste.
Depuis plusieurs années, les chercheurs des labo-
ratoires de Physiologie Cellulaire Végétale et
d'Étude de la Dynamique des Protéomes contri-
buent de manière significative à l’établissement de
répertoires protéiques pour
divers compartiments de la
cellule d’Arabidopsis thaliana.
Cependant, un des enjeux
actuels de la protéomique est
la réalisation d’études compa-
ratives afin de suivre des pro-
cessus dynamiques. Parmi les
stratégies d'analyses protéo-
miques quantitatives qui ont
été développées, la méthode
AMT (Accurate Mass and time
Tags) (lettre thématique de
l'iRTSV sur l'Analyse protéo-
mique) et celle dite de Spectral
count ont permis à ces cher-
cheurs d'établir la première
base de données AMT pour un
organite spécifique de la cel-
lule végétale, le chloroplaste.
La base de données AT_Chlo-
ro a été établie en réalisant
près de 500 analyses sur des fractions hautement
purifiées des compartiments majeurs du chloro-
plaste (enveloppe, stroma et thylacoïdes). Des
informations précises ont ainsi été obtenues pour
10654 peptides distincts (Figure 1) correspondant à
1323 protéines validées. L'ensemble des données a
ensuite été structuré dans AT_Chloro, une base de
données spécifique du chloroplaste d’Arabidopsis
thaliana. La création de cette base de données per-
mettra de réaliser les
analyses ultérieures par
nanoLC-FT-MS, en évi-
tant le recours systéma-
tique à la MS/MS, ce qui
permet d’augmenter
significativement le débit
d’analyse et la couver-
ture du protéome étudié.
Dans l’optique d’analy-
ses protéomiques com-
paratives, la méthode
AMT présente l'avantage
de pouvoir comparer
directement entre elles
les intensités des si-
gnaux MS obtenus lors
de l’analyse de différents
échantillons protéiques
complexes.
L'association de l'AMT
et du Spectral Count a
permis d'obtenir à partir
d’une seule et même
analyse, les données
d’identification et de
quantification. Cette
stratégie mise en œuvre
pour la première fois a permis de déterminer la
localisation sub-chloroplastique des protéines
identifiées pour construire la base AT_Chloro.
Dans cette étude, un effort majeur d’annotation a
été réalisé plus spécifiquement pour les protéines
de l’enveloppe des chloroplastes, site privilégié
des interactions
entre les plastes et
le reste de la cel-
lule. Ainsi, près
de 500 protéines
(sur les 1323 pro-
téines identifiées
dans le chloro-
plaste) ont été
identifiées dans
l’enveloppe alors
qu’elles ne repré-
sentent (en masse)
que 1 à 2% des
protéines du chlo-
roplaste. Ces ré-
sultats confirment
l’importance fonc-
tionnelle de ce
système membra-
naire. L’analyse
protéomique réalisée a permis de replacer les pro-
téines identifiées dans le contexte fonctionnel des
voies métaboliques et du rôle que jouent l’enve-
loppe, le stroma ou les thylacoïdes dans le fonc-
tionnement du chloroplaste (Figure 2).
AT_Chloro : première base de données AMT
des protéines du chloroplaste
Contacts : Norbert Rolland
UMR 5168 CEA - CNRS - Inra - UJF
Myriam Ferro, Christophe Masselon
Unité mixte CEA - Inserm U880 - UJF
Figure 2 : Compartimentation du métabolisme des composés terpéniques
dans le chloroplaste déterminée à partir des données de protéomique semi-
quantitative. L’analyse protéomique par «!spectral count!» montre que les premiè-
res étapes (voies du méthylérythritol phosphate, du shikimate ou du protoporphyri-
nogène IX, par exemple) se déroulent dans le stroma (les protéines sont indiquées
par des ronds oranges)!; puis, dès que les molécules deviennent hydrophobes, les
voies métaboliques impliquent les membranes : soit les thylacoïdes (ronds verts), soit
l’enveloppe (ronds jaunes). Les ronds bleus correspondent à des protéines dont la
localisation fine n’a pas été possible. Les résultats démontrent en particulier le rôle
majeur de l’enveloppe dans la biosynthèse des caroténoïdes (précurseurs entre autres
de l’hormone ABA), des prénylquinones (plastoquinone, α-tocopherol) et de la pro-
tochlorophyllide, précurseur de la chlorophylle.
Figure 1 : Carte peptidique massique des ions (pep-
tides) détectés par LC-MS dans le chloroplaste. Axe
des abscisses : temps de rétention des peptides sur la
colonne nanoLC (min) ; Axe des ordonnées : masse isoto-
pique des différents peptides (Da). Les différents états de
charge (z) des peptides sont représentés par différentes
couleurs : z=1 en bleu ; z=2 en rouge ; z=3 en vert.
Références
Ferro et al. AT_CHLORO: A comprehensive
chloroplast proteome database with
subplastidial localization and curated
information on envelope proteins. Molecular and
Cellular Proteomics, 2010
Joyard et al. Chloroplast proteomics and the
compartmentation of plastidial isoprenoid
biosynthetic pathways. Molecular Plant, 2009, 2:
1154-1180
Joyard et al. Chloroplast proteomics highlights
the subcellular compartmentation of lipid
metabolism. Progress in Lipid Research, 2010,
49(2): 128-158
!La base AT_Chloro va au-delà d’un simple
répertoire de protéines en présentant des don-
nées semi-quantitatives qui permettent de dé-
terminer dans quel compartiment (enveloppe,
stroma, thylacoïdes) se trouve une protéine du
chloroplaste. Il est donc possible de préciser le
rôle de chacun des compartiments dans les
voies métaboliques du chloroplaste.
!La base AT_Chloro, qui renferme une
somme importante d’informations sur les pro-
téines de l’enveloppe des chloroplastes, consti-
tue une base de connaissance unique sur ce
système membranaire.
!AT_Chloro représente une avancée significa-
tive en ouvrant la voie à des études quantitati-
ves systématiques permettant de comparer les
protéomes chloroplastiques de mutants ou de
suivre la dynamique de ces protéomes au cours
du développement et dans des plantes soumises
aux modifications de leur environnement.
Thylacoïdes EnveloppeStroma

LETTRE SCIENTIFIQUE
Immuno-protéomique - Moteurs moléculaires!3
certains sucres particuliers, comme l'héparane que
l'on retrouve dans la matrice extracellulaire de nos
tissus, mais aussi le chitosane qui est un consti-
tuant des parois de certains champignons, mais
aussi des parois de formes enkystantes de certains
protozoaires. Cette découverte ont immédiatement
amené ces chercheurs à réaliser certaines expérien-
ces visant à comprendre l'effet du chitosane sur les
cellules dendritiques.
Ces expériences ont alors montré que le chitosane
était capable d'induire une réponse partielle des
cellules dendritiques. Celles ci actionnent leur
programme d'activation membranaire, comme
pour tout démarrage d'une réponse immune, mais
s'avèrent incapables de sécréter les
cytokines nécessaires. En conséquence,
les cellules dendritiques traitées par le
chitosane sont incapables de stimuler
les lymphocytes T, et donc d'activer
une réponse immune efficace. Cepen-
dant, cette immunosuppression n'est
pas dominante : des cellules dendriti-
ques traitées en même temps par du
chitosane et des produits bactériens
comme le lipopolysaccharide, activateur puissant,
sont capables de stimuler normalement les lym-
phocytes T.
Replacés dans un contexte physiologique, ces ré-
sultats suggèrent que le chitosane peut induire une
immunodulation négative et localisée aux sites où
il est présent en fortes concentrations, typiquement
les sites d'infection par des organismes en conte-
nant dans leur paroi, et donc participer aux phé-
nomènes de survie de ces organismes en présence
du système immunitaire. Ceci pourrait contribuer
à expliquer la persistance de ces parasites, par un
mécanisme d'adaptation fin jouant sur les méca-
nismes de contrôle interne du système immuni-
taire, tout en évitant une immunosuppression
massive qui serait dommageable pour l'hôte et
pour le parasite.
Les mycoses cutanées, comme le pied
d'athlète, font partie des ces petites affections
dont il est difficile de se débarrasser, mais qui
restent bénignes. Ce n'est hélas pas le cas de
toutes les mycoses, qui peuvent être fatales
aux sujets immunodéprimés, ou d'autres in-
fections par des parasites comme les amibia-
ses. Ce qui caractérise ces affections, c'est leur
grande résistance au système immunitaire.
Celui-ci arrive souvent à les contrôler, mais
difficilement à les éradiquer, alors même que
ces parasites, non intracellulaires
et à croissance lente, devraient être
facilement éliminés par notre sys-
tème immunitaire.
Cette question peut sembler éloi-
gnée des analyses fondamentales
en immunologie, et pourtant le lien
est plus court qu'il n'y paraît.
L'équipe dirigée par Thierry Rabilloud du labora-
toire Biochimie et Biophysique des Systèmes Inté-
grés et des chercheurs de l'Institut Albert Bonniot, se
sont en effet intéressés au fonctionnement des cellu-
les dendritiques. Ces cellules jouent un rôle clé dans
la réponse immune en étant capables d'activer les
cellules effectrices de la réponse immune, en parti-
culier les lymphocytes T, qui activent à leur tour les
autres cellules effectrices de la réponse immune.
En étudiant par des techniques d'analyse protéomi-
que les protéines sécrétées par les cellules dendriti-
ques (analyse du secrétome), ces chercheurs ont
retrouvé les cytokines communément décrites, mais
aussi trouvé des protéines peu caractérisées, comme
la lectine YM1. Cette protéine a la capacité de lier
Pourquoi vous avez mal aux pieds, ou les surprises
de l'analyse immunoprotéomique
Référence
Villiers C, Chevallet M, Diemer H, Couderc R,
Freitas H, Van Dorsselaer A, Marche PN and
Rabilloud T. From secretome analysis to
immunology: Chitosan induces major
alterations in the activation of dendritic cells via
a TLR4-dependent mechanism. Molecular and
Cellular Proteomics, 2009, 8(6): 1252-1264
Contact : Thierry Rabilloud
UMR 5092 CEA - CNRS - UJF
Analyse protéomique des protéines sécrétées par les
cellules dendritiques.
Gel 2D.
La plasticité tissulaire se traduit par la mi-
gration des cellules les unes par rapport aux
autres sans que l’intégrité de ce tissu soit
rompue. Ce remodelage est vraisemblable-
ment corrélé au comportement dynamique
des jonctions intercellulaires et plus particu-
lièrement à leurs connexions au cytosque-
lette d'actine.
Le tissu modèle utilisé par les chercheurs de
l'équipe dirigée par Danielle Gulino du laboratoire
Angiogenèse et Physiopathologie Vasculaire est
l’endothélium vasculaire dont le rôle est de contrô-
ler les échanges entre le sang et les tissus. Sa cohé-
sion est maintenue en partie par la VE-cadhérine,
un récepteur adhésif localisé spécifiquement aux
jonctions intercellulaires endothéliales. La VE-
cadhérine élabore, via sa partie extracellulaire, des
interactions homophiliques entre molécules appar-
tenant à des cellules voisines et s’associe, par son
domaine cytoplasmique, à des protéines appelées
caténines. La connexion des complexes à base de
cadhérine au réseau d’actine via les caténines a
pour effet de renforcer la cohésion de l’endothé-
lium.
Depuis plusieurs années ces chercheurs étudient
les mécanismes à l’origine de la formation des
complexes jonctionnels à base de VE-cadhérine. Ils
crées pour cela des lésions au sein de monocou-
ches endothéliales qui miment l’endothélium vas-
culaire et étudient la réparation de ces lésions. Ces
chercheurs ont observé que sur le pourtour des
lésions, les cellules explorent leur environnement
en émettant des filopodes (i.e. de fines et longues
extensions membranaires) sur lesquels circulent
très rapidement des «!patches!» de VE-cadhérine.
Moteurs moléculaires
Transport du complexe à base de VE-cadhérine le
long des filaments d’actine intrafilopodiaux.
La protéine moteur Myosine-X se déplace à une vi-
tesse moyenne de 700 nm/s vers l’extrémité barbée des
filaments d’actine intrafilopodiaux (mouvement anté-
rograde A) mais également à une vitesse moyenne de
30 nm/s vers la base des filopodes lorsqu’elle est sou-
mise au flux rétrograde d’actine (mouvement rétro-
grade R). Elle transmet ces deux types de mouve-
ments au complexe de VE-cadhérine en interagissant
directement, par l’intermédiaire de son domaine
FERM, avec la partie cytoplasmique de la VE-cadhé-
rine. βcat!: β-caténine!; αcat!: α-caténine.

LETTRE SCIENTIFIQUE
4!Moteurs moléculaires - En bref
Lorsqu’un filopode touche une cellule voisine, les
molécules de VE-cadhérine sont projetées à l’ex-
trémité de ce filopode et interagissent avec leurs
homologues de la cellule cible. Ce type de trans-
port permet de multiplier les contacts adhésifs
intercellulaires et d’arrimer les cellules les unes
aux autres. Ces chercheurs ont montré que la myo-
sine X, une protéine moteur de la famille des myo-
sines, régissait le transport des molécules de VE-
cadhérine le long des filopodes. (Détails dans la
légende de la Figure page précédente).
Le concept du transport de la VE-cadhérine par la
Myosine-X semble pouvoir être étendu à d’autres
cadhérines qui s’expriment dans des tissus va-
riés. Ceci suggère que l’accumulation des cadhé-
rines à l’extrémité des filopodes semble un pro-
cessus général utilisé par les cellules pour cons-
truire de nouvelles jonctions intercellulaires ou
remanier celles déjà existantes. Ces connaissan-
ces apportent un éclairage nouveau sur les méca-
nismes à la base du remodelage, de la réparation
tissulaires et de l’angiogenèse.
Contact : Danielle Gulino
Unité mixte CEA - Inserm U882 - UJF
Plant Systems Biology
"En bref
Référence
Almagro et al. The motor protein Myosin-X
transports VE-cadherin along filopodia to allow
the formation of early endothelial cell-cell con-
tacts. Molecular and Cellular Biology, 2010, 30(7):
1703-1717
Publication d'un numéro spécial (Focus
Issue) sur le thème Plant Systems Biology de
la revue américaine Plant Physiology.
La complexité des
systèmes biologiques
et la production en
masse de données
expérimentales ont
mené au développe-
ment de nouvelles
approches intégratives
et pluridisciplinaires et
à l’utilisation de la
modélisation pour
chercher à comprendre
le fonctionnement des organismes vivants placés
dans diverses conditions environnementales. Cette
manière d'aborder l'étude des questions biologi-
ques n'en est qu'à ses premiers pas, en particulier
dans le domaine végétal.
Dans un numéro spécial de Plant Physiology, "Focus
Issue on Plant Systems Biology", édité par Jacques
Joyard du laboratoire de Physiologie Cellulaire
Végétale et Sheila McCormick (UC Berkeley), une
série de mini revues (Updates) et des articles origi-
naux traitent de ces questions qui vont de!la quan-
tification et l'intégration de données moléculaires
complexes (expression de gènes, protéomique, etc.)
à l'identification de réseaux de régulation (signali-
sation, métabolisme) et leur modélisation.!Ce nu-
méro vient enrichir la série Focus Collections dispo-
nible en ligne. Ainsi, Plant Systems Biology 2010
recensera à terme tous les articles publiés sur ce
thème dans Plant Physiology depuis février 2008 et
jusqu'à février!2012.
Référence
Joyard J and McCormick S. Plant Systems Biol-
ogy. Plant Physiology, 2010, 152(2): 401

LETTRE SCIENTIFIQUE
Contacts : [email protected]!5
Les laboratoires de l’iRTSV
Directeur de la publication
Dr. Jérôme Garin
Éditeur et format électronique
Pascal Martinez — [email protected]
Comité de rédaction
Rajaa Boujemaa-Paterski, Danielle Gulino, Jacques Joyard,
Thierry Rabilloud.
Institut de Recherches en Technologies et
Sciences pour le Vivant
http://www-dsv.cea.fr/irtsv
http://www-dsv.cea.fr/irtsv/lettres
CEA Grenoble
17 rue des Martyrs
38 054 Grenoble cedex 09
Responsable : Jérôme Garin
Tel. : 04 38 78 45 01
Fax : 04 38 78 51 55
© CEA [2010]. Tous droits réservés. Toute reproduction totale ou partielle sur quelque support que ce soit ou utili-
sation du contenu de ce document est interdite sans l’autorisation écrite préalable du CEA
LAPV
Laboratoire Angio-
genèse et Physiopa-
thologie Vasculaire
LBBSI
Laboratoire Biochi-
mie et Biophysique
des Systèmes Inté-
grés
Biopuces
Laboratoire Biopu-
ces
LCBM
Laboratoire Chimie
et Biologie des Mé-
taux
LEDyP
Laboratoire Étude
de la Dynamique
des Protéomes
LBIM
Laboratoire Biolo-
gie, Informatique et
Mathématiques
LPCV
Laboratoire Physio-
logie Cellulaire Vé-
gétale
LTS
Laboratoire Trans-
duction du Signal
GPC
Groupe Physiopa-
thologie du Cytos-
quelette
GIPSE
Groupe Informati-
que Pour les Scien-
tifiques du sud-Est
GPMS
Groupe Plate-forme
et Moyens Scientifi-
ques et techniques
communs
1
/
5
100%