Projet de recherche Diagnostic du cancer du sein basé sur la

Projet de recherche
Diagnostic du cancer du sein basé
sur la technologie microarray
Benjamin Haibe-Kains
Promoteur : Gianluca Bontempi Co-promoteur : Christos Sotiriou

Table des matières
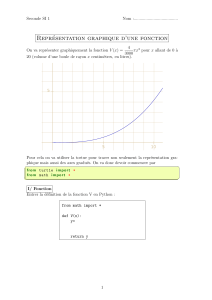
Problème biologique
Classification par apprentissage supervisé
Etat de l’art
Objectifs de la recherche
Stratégie scientifique
Plan de la recherche
Perspectives industrielles
Pr´
esentation F.R.I.A. – p.1/15

Problème biologique
Le cancer du sein représente un tiers des cancers
diagnostiqués
Détection au stade précoce ➙traitement
systémique (hormono/chimiothérapie) pour les
patients à haut risque
Evaluation du risque
basé sur des critères histo-pathologiques
défini par des consensus reconnus (St Gallen,
NIH, NPI)
important dans la pratique clinique
Pr´
esentation F.R.I.A. – p.2/15

Problème biologique(2)
Taux de sur-traitement élevé
haut risque = 80% des patients selon les
consensus actuels
récidive = 30% des patients (métastases à
distance)
Amélioration de l’évaluation du risque
éviter les effets secondaires non-désirables
diminuer les coûts du suivi médical
Pr´
esentation F.R.I.A. – p.3/15

Classification par appr. supervisé
Problème biologique →problème de classification par
apprentissage supervisé
entrée : données
microarray dérivées
de tissus tumoraux
sortie (oui/non) :
apparition de mé-
tastases à distance
endéans les 5 pre-
mières années de
suivi médical
Pr´
esentation F.R.I.A. – p.4/15
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
1
/
18
100%