Demande de photocopie
publicité

Master de Sciences et Technologies Mention Biologie Intégrative et Physiologie Spécialité : Neurosciences Responsable : Professeur Régis LAMBERT Fiche à renvoyer avant le 14 mars 2016 à [email protected] et [email protected] Proposition de Stage M2 S4 NEUROSCIENCES Année Universitaire 2016-2017 1. Equipe d’Accueil de Master (EAM) : Intitulé et numéro de l’Unité : CIRB – Collège de France, CNRS UMR 7241, INSERM U1050 Nom du Responsable de l’Unité : Professeur Alain PROCHIANTZ Nom du Responsable de l’Équipe : Professeur Alain PROCHIANTZ Intitulé de l’équipe d’accueil : Chaire des Processus Morphogénétique Développement et Neuropharmacologie Adresse : CIRB – Collège de France, 11 Place Marcelin Berthelot, 75231 Paris Cedex 05 Nom du responsable de l’encadrement : Rajiv JOSHI / Julia FUCHS Tél. : 01 44 27 14 55 / 01 44 27 15 18 Fax. : 01 44 27 14 65 E-mail : [email protected] / [email protected] 2. Titre du sujet : Etude des mécanismes d’action d’Engrailed dans la survie et la physiologie des neurones dopaminergiques. 3. Description du sujet : Le laboratoire s’intéresse depuis plusieurs années au rôle des facteurs de transcription de la famille des homéoprotéines dans le développement, la plasticité et certaines pathologies du système nerveux. Les différents projets de l’équipe sont tous basés sur les propriétés de transfert intercellulaire des homéoprotéines, ce qui représente un nouveau mode de signalisation et de communication intercellulaire (Prochiantz & Joliot, Nat. Rev. Mol. Cell. Biol. 4, 814-819, 2003). Le laboratoire a démontré ces dernières années l’importance physiologique d’un tel transfert dans le guidage axonal, la plasticité corticale, la migration neuronale ainsi que le potentiel thérapeutique de la transduction par les homéoprotéines. (Prochiantz & Di Nardo, Neuron 8, 911-925, 2015). Le groupe « Engrailed et la maladie de Parkinson » s’intéresse au rôle du facteur de transcription Engrailed (Engrailed-1/Engrailed-2, collectivement En1/2) dans la survie et la physiologie des neurones dopaminergiques (mDA) chez l’adulte. Le laboratoire avait précédemment montré que l’invalidation d’un allèle En1 chez la souris conduit à la dégénérescence progressive des neurones mDA à partir de l‘âge de 6 semaines (Sonnier et al., J. Neurosci. 27, 1063-1071, 2007). Cette dégénérescence pourrait être rétrograde et impliquer une altération de l’autophagie (Nordström et al., Neurobiol. Dis. 73, 70-82, 2015). Ces souris représentent donc un modèle très intéressant de la maladie de Parkinson. De plus, la transduction des neurones mDA par En1/2 permet de stopper la mort de ces neurones. Le potentiel thérapeutique d’En1/2 a maintenant été démontré dans différents modèles expérimentaux de la maladie de Parkinson (Alvarez-Fisher, Fuchs et al., Nature Neurosci. 14, 1260-1266, 2011). Cette activité d’Engrailed passe en partie par la stimulation de la traduction par En1/2 de certaines sous unités du complexe I mitochondrial codées par le génome nucléaire. Engrailed protège aussi contre des altérations de la chromatine et les cassures d’ADN induites par le stress oxydatif, et agit comme une protéine anti-vieillissement (Rekaik et al. Cell Reports 13, 1-9, 2015). Plus récemment nous avons aussi exploré un lien entre En1/2, la répression d’éléments rétrotransposables (LINEs) et la stabilité du génome (Blaudin de Thé et al., soumis). Mis en forme : Français (France) Master de Sciences et Technologies Mention Biologie Intégrative et Physiologie Spécialité : Neurosciences Responsable : Professeur Régis LAMBERT Le programme de recherche que nous poursuivons a pour objectif de mieux comprendre les mécanismes moléculaires et les voies de signalisation impliquées dans l’activité d’Engrailed sur la survie et la physiologie des neurones mDA en relation avec la maladie de Parkinson. Le stage de Master M2 portera sur deux aspects : 1) Etude des mécanismes épigénétiques impliqués dans l’activité d’Engrailed. Nous avions précédemment montré qu’Engrailed permet non seulement de sauver les neurones mDA dans des modèles toxicologiques de la maladie de Parkinson mais aussi d’augmenter l’activité physiologique des neurones mDA résiduels. Cet effet d’Engrailed perdure plusieurs semaines après l’arrêt du traitement par Engrailed et pourrait impliquer une régulation épigénétique. Nous recherchons les cibles transcriptionnelles et des cibles épigénétiques d’Engrailed pouvant être modifiées par méthylation de l’ADN ou des changements de l’état de leur chromatine (méthylation/déméthylation CpH et des histones en particulier). Les données préliminaires montrent qu’Engrailed pourrait réguler la méthylation/déméthylation d’un certain nombre de gènes longs, ainsi que de certains éléments rétrotransposables (i.e. LINE-1). 2) Etude du rôle possible d’ARN non codants dans l’activité d’Engrailed, Nos résultats préliminaires sur la recherche de cibles transcriptionnelles d’Engrailed avaient suggéré que le gène H19, codant pour un long ARN non-codant, pourrait être une cible d’Engrailed. Certaines études récentes ont montré que l’ARN H19 pourrait être un régulateur de l’état de méthylation globale du génome. Le gène H19 se trouve à proximité du gène codant pour la tyrosine hydroxylase (TH), enzyme clé pour la synthèse de la dopamine. Nous allons donc examiner si l’ARN H19 ne pourrait pas être un régulateur de l’expression du gène Th. Par ailleurs, nos avons aussi observé qu’Engrailed peut lier certains petits ARN non codants (miARN/piARN). Nous allons également examiner le rôle possible de ces ARN dans l’activité d’Engrailed dans la survie ou la physiologie des neurones mDA. Méthodologie : Ces études sont menées en utilisant des modèles de souris invalidées pour En1 ou des cultures de neurones primaires mésencéphaliques. Les méthodologies employées incluent des expériences d’injections stéréotaxiques dans le cerveau de la souris, la mise en culture des neurones primaires, des approches en d’immunohistologie et microscopie confocale, des analyses par western blots, l’immunonoprécipitation de la chromatine (ChIP) suivie de séquençage (et PCR) et l’analyse de l’expression de gènes par séquençage et qRT-PCR. Publications de l’équipe relatives au sujet : Sonnier et al., J. Neurosci. 27, 1063-1071, 2007. Alvarez-Fisher, Fuchs et al., Nature Neurosci. 14, 1260-1266, 2011. Nordstrom et al., Neurobiol. Dis. 73, 70-82, 2015. Rekaik, Blaudin de Thé et al., Cell Reports 13, 242-250, 2015. Rekaik et al., FEBS Lett. 589, 3786-3894, 2015. (Invited review)




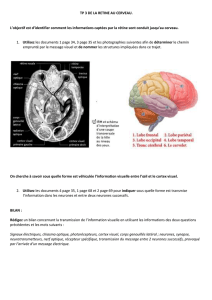

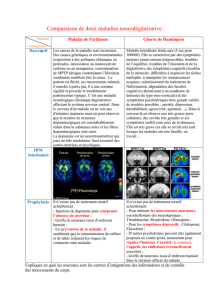




![Découverte d un nouveau centre cérébr[...]](http://s1.studylibfr.com/store/data/001261824_1-044b689d1e2faad91148811640c2eb34-300x300.png)