Bertolla - Spiral Connect

PROPOSITION SUJET de MASTER 2016-2017
TITRE : Etiologie de la bactériose de la laitue
Nom, Prénom du Maitre de Stage : Bertolla Franck
Qualité : Maître de Conférences
Téléphone : 04 72 43 27 58 E-mail : franck.bertolla@univ-lyon1.fr
Nom, Prénom du co-encadrant éventuel : Burlet Alexandre
Qualité : Chargé d’expérimentation
Téléphone : 04 78 87 97 59 E-mail : burlet.serail@orange.fr
Laboratoire d’accueil, Responsable et équipe : Ecologie Microbienne
Adresse : 16, rue Raphaël Dubois / Bâtiment Gregor Mendel
F-69622 Villeurbanne cedex, France
Station SERAIL
123 Chemin du Finday
69126 BRINDAS
Nom du candidat éventuellement proposé :
S'il n'est pas retenu, acceptez-vous un autre candidat ? Oui - Non
Description du sujet au verso

Sujet (objectif, démarche et technique, collaboration(s),...) :
Enjeux et problématique :
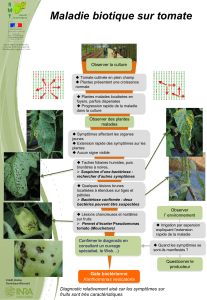
Sur cultures de laitues, une maladie infectieuse bactérienne provoque des dégâts pouvant aller jusqu'à la
destruction de la récolte et des pertes économiques importantes pour les maraîchers. La bactériose de laitue
est aujourd’hui la plus grosse crainte sanitaire aussi bien chez les producteurs spécialisés, que les producteurs
diversifiés, qu’ils soient en agriculture conventionnelle ou biologique, car jusqu’à aujourd’hui aucune solution
n’existe pour lutter contre ces attaques de bactériose. Dès lors cette filière professionnelle exprime une forte
demande pour travailler sur des solutions de lutte contre cette bactériose.
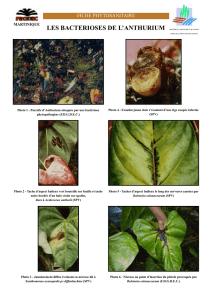
Cependant, de nombreux paramètres étiologiques sont encore inconnus. La nomenclature exacte de l’agent
pathogène n'est pas clairement établie, même si de précédentes études ont identifiées des souches
appartenant au groupe Xanthomonas campestris pv. vitians. Les rôles de l’origine de plants, des itinéraires
techniques utilisés, des caractères pédologiques des parcelles, du climat et de l’irrigation dans l’apparition de
cette maladie sont également méconnus et constituent des informations déterminantes pour l’étude de
l’étiologie de bactériose de la laitue.
Objectifs :
Le projet aura donc pour but de rechercher et de vérifier l’identité et l’éventuelle diversité du bioagresseur.
Les sources d’inoculum de X. campestris pv vitians seront également recherchées. Ainsi, des prélèvements
seront effectués sur les laitues, dans le sol proche des laitues attaquées puis en s’éloignant jusqu’à une zone
saine (eau, mauvaises herbes potentiellement hôtes du pathogène) dans des exploitations de la région Rhône
Alpes.
Démarches expérimentales :
Le travail consistera à broyer les échantillons végétaux naturellement infectés puis d’isoler sur les milieux de
culture les plus adéquats la bactérie phytopathogène afin de réaliser des extractions d’ADN sur des cultures
pures. Dès lors des analyses génétiques moléculaires seront réalisées, tels que des amplifications PCR avec
des amorces spécifiques pour certains gènes bactériens conservés (rpoD, gyrB). Leurs séquences permettront
d’aborder plus précisément l’identification des isolats. Suivant la diversité génétique rencontrée, trois isolats
seront séquencés par les nouvelles techniques de séquençages à haut débit.
Grâce aux génomes et à la comparaison de ceux-ci aux bases de données de génomes bactériens, nous
identifierons des marqueurs génétiques spécifiques à cette ou ces bactéries pathogènes de la laitue.
L’élaboration d’amorces moléculaires spécifiques de ces marqueurs nous permettra plus facilement de
détecter et d’identifier le ou les pathogènes dans les tissus de plantes infectées et dans les réservoirs
possibles (sols, eaux, laitues sauvages).
Une fois la caractérisation réalisée et le bioagresseur identifié, des essais de reproduction de symptômes de
bactériose seront mis au point en conditions contrôlées. Ces tests permettront d’identifier les leviers
favorables à l’expression de la maladie (orage, température, hygrométrie…). Ils seront affinés selon les
souches de X. campestris pv vitians retrouvées sur les différentes zones et selon la concentration de
l’inoculum de chaque souche.
Collaborations :
Ce projet sera réalisé en collaboration avec la Station d’Expérimentation Rhône Alpes Information Légumes
« SERAIL » et plus particulièrement Alexandre Burlet. Le SERAIL, fort de son réseau de producteurs, apportera
toute son expertise dans le suivi sur le terrain de la maladie et dans l’expérimentation de type lutte
biologique.
Ce projet est financé par la région Rhône Alpes et l’Europe.
1
/
2
100%