Technologie de l`ADN Recombinant CHMI 4226 F

E.R. Gauthier, Ph.D. CHMI 3216 F –A2009 1
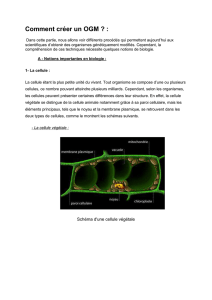
Boiningénierie de l’ADN
CHMI 3216 E
Semaine du 14 Septembre 2007
Boîte à outils, 2ième partie.
Plasmides, clonage d’ADN 101

E.R. Gauthier, Ph.D. CHMI 3216 F –A2009 2
Plasmides
•Plasmides:
– Molécules d’ADN circulaires extrachromosomales
–Caractéristiques de base:
•Origine de réplication: permet au plasmide de se répliquer
de manière autonome;
•Gène de résistance à un antibiotique: permet la sélection
de bactéries possédant le plasmide;
•Site de clonage multiple (multiple cloning site -MCS): une
petite région du plasmide crée en laboratoire et contenant un
certain nombre de sites de coupure uniques pour des
enzymes de restrictions.

E.R. Gauthier, Ph.D. CHMI 3216 F –A2009 3
Plasmides –origine de réplication
• Origine de réplication (Ori): courte séquence d’ADN permettant à l’ADN
polymérase bactérienne de lier et initier la réplication du plasmide;
• Il existe plusieurs types d’Ori. ColE1 celui rencontré le plus souvent;
•Certaines Ori permettent au plasmide de se répliquer fréquemment
(plasmides à nombre de copies élevées – jusqu’à 100 copies par
bactérie); d’autres Ori ne permettent qu’un taux relativement faible
d’initiation de la réplication (plasmides à nombre de copies faible–
seulement quelques copies de plasmide par bactérie);

E.R. Gauthier, Ph.D. CHMI 3216 F –A2009 4
Plasmides –gènes de sélection
•Gènes codant des protéines permettant la résistance bactérienne à
un antibiotique particulier;
•Gènes le plus souvent rencontrés:
–Ampr: résistance à l’ampicilline
–Kanr: résistance à la kanamycine
–Tetr: résistance à la tétracycline
•Les bactéries possédant un plasmide ayant le gène Amprseront
capable de survivre lorsque cultivées dans un milieu de culture
contenant de l’ampicilline;

E.R. Gauthier, Ph.D. CHMI 3216 F –A2009 5
Plasmides –sites de clonage
multiples
•MCS: une région limitée du plasmide crée artificiellement
et qui possède plusieurs sites de coupure uniques pour
un ensemble d’enzymes de restriction;
•Les sites de restriction retrouvés dans le MCS ne sont
pas retrouvés ailleurs dans le plasmide en question.
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
1
/
18
100%