Décryptage du contrôle génétique des réponses immunitaires

Décryptageducontrôlegénétiquedesréponses
immunitairesinnéesetadaptativeschezleporcLargeWhite:
uneétudecombinantdesapprochesgénétiques
etfonctionnelles
ClaireROGEL‐GAILLARD(1,2,3),LaurenceFLORI(1,2),YuGAO(1,2),IsabelleOSWALD(4),FrançoisLEFEVRE(5),
MarcelBOUFFAUD(6),Marie‐JoséMERCAT(7),Jean‐PierreBIDANEL(1,2)
(1)INRA,UMRdeGénétiqueAnimaleetBiologieIntégrative,Jouy‐en‐Josas,France
(2)AgroParisTech,UMRdeGénétiqueAnimaleetBiologieIntégrative,Jouy‐en‐Josas,France
(3)CEA,DSV,iRCM,LaboratoiredeRadiobiologieetEtudeduGénome,Jouy‐en‐Josas,France
(4)INRA,LaboratoiredePharmacologieToxicologie,Toulouse,France
(5)INRA,LaboratoiredeVirologieetImmunologieMoléculaires,Jouy‐en‐Josas,France
(6)INRA,Stationdecontrôledeperformances,UE450,LeRheu,France
(7)IFIP‐BIOPORC,Pôlegénétique,LaMotteauVicomte,France
claire.rogel‐[email protected]
AveclacollaborationtechniquedeGaëtanLEMONNIER(1,2,3),AngéliqueTEILLAUD(1,2,3),Jean‐JacquesLEPLAT(1,2,3),
StephanBOUET(1,2,3),FabriceANDREOLETTI(1,2,3),Anne‐MarieCOSSALTER(4),JoelleLAFFITTE(4),
ChristianedeVAUREIX(5)etlesoutienfinancierdel’AgenceNationaledelaRecherche
(ProjetIMMOPIG,programmeGenanimal2007‐2009)
DécryptageducontrôlegénétiquedesréponsesimmunitairesinnéesetadaptativeschezleporcLargeWhite:uneétude
combinantdesapprochesgénétiquesetfonctionnelles
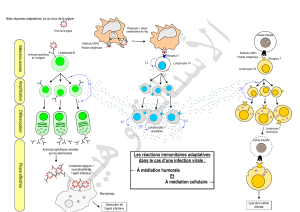
Unprogrammed'analysefineducontrôlegénétiquedelaréponseimmunitaire(RI)combinantdesapprochesgénétiqueset
fonctionnellesaétédéveloppé.Plusde400animauxderaceLargeWhitemesuréspourdescaractèresdeproductionontété
caractériséspourunlargeéventaildeparamètresdel'immunitétroissemainesaprèsvaccinationcontreMycoplasma
hyopneumoniae.Cesparamètresincluent:i)unenumération‐formulesanguineetuncomptageparcytométriedefluxdecinq
sous‐populationsleucocytairescaractériséesparlaprésenceàleursurfacedesmarqueursIgM,TCRγδ,CD4/CD8,CD16/CD2et
CD16/CD172a/MHCII,ii)desparamètresdelaRIinnée(phagocytoseetproductioninvitrodescytokinesIL1B,IL6,IL8,TNF,IL12et
IFNαaprèsstimulationdusangtotal),iii)desparamètresdeRIadaptative(proliférationlymphocytaire,productioninvitrodes
cytokinesIL2,IL4,IL10etIFNγ,dosagessériquesdesimmunoglobulines(Ig)totalesA,G,MetdesIgGspécifiquesanti‐Mycoplasma
hyopneumoniae)etiv)deuxprotéinesdelaphaseinflammatoireaiguë(protéineC‐réactiveethaptoglobine).Lesestimations
d'héritabilitéatteignent0,4enmoyenne(SE=0,1)et42des54paramètresmesurésontmontrédesvaleursd'héritabilitéestimées
modéréesàélevées(≥0,2).Cesvaleursconfirmentquedenombreuxparamètressontsouscontrôlegénétiqueetpourraientêtre
inclusdanslesprogrammesdesélection.Lesanalysesfonctionnellesontrévéléqueletranscriptomesanguinestinformatifpour
unepartiedescaractèresd'immunitémesurésetpourraitêtreutilepouraffinernosconnaissancessurl'immunocompétencechez
leporc.
DecipheringthegeneticcontrolofinnateandadaptiveimmuneresponsesinLargeWhitepigs:acombinedgeneticandgenomic
study
Improvinganimalrobustnessandresistancetopathogensbyaddinghealthcriteriatoselectionschemesisoneofthechallenging
objectivesofthenextdecade.InordertobetterunderstandthegeneticcontrolofimmunityinFrenchLargeWhitepigs,wehave
launchedaprogramcombininggeneticandgenomicstudiesthatdonotfocusonanyparticularpathogen.Morethan400animals
recordedforproductiontraitswerescoredforawiderangeofimmunityparametersthreeweeksaftervaccinationagainst
Mycoplasmahyopneumoniae:i)totalwhitebloodcellsandlymphocytecountsandproportionsofvariousleucocytesubsets
includingcellsharbouringIgM,γδTCR,CD4/CD8,CD16/CD2andCD16/CD172a/MHCII,ii)innateimmuneresponseparameters
(phagocytosisandinvitroproductionofIL1B,IL6,IL8,TNF,IL12andIFNαafterbloodstimulation),iii)adaptiveimmuneresponse
parameters(lymphocyteproliferation,invitroproductionofIL2,IL4,IL10andIFNγafterbloodstimulation,totalIgG,IgA,IgMand
specificIgGlevels)andiv)twoacutephaseproteins(C‐reactiveproteinandhaptoglobin).Heritabilityestimateswere0.4on
average(SE=0.1)and42ofthe54measuredparametersshowedmoderatetohighheritabilities(≥ 0.2),confirmingthatmany
parametersareundergeneticcontrolandcouldbeincludedinselectionprotocols.Functionalanalysesrevealedthatblood
transcriptomeisinformativeforsomeoftheimmunitytraitsandcouldberelevanttorefineourknowledgeonpig
immunocompetence.
2011. Journées Recherche Porcine, 43.
27

INTRODUCTION
Chezleporc,l’élaborationdeprogrammesdesélection
efficacessurlescaractèresdeproductionetl’applicationde
règlessanitairesstrictesontconduitàuneamélioration
hautementsignificativedesperformanceszootechniques
pendantles30dernièresannées.Or,denouvelles
considérationsliéesaubien‐êtreanimal,àlasécuritédu
consommateuretlamiseenplacedenouvellesdirectivesà
l’échelleeuropéenneontconduitàsupprimerl’usagedes
antibiotiquesàdesfinsdecroissanceetàlimiterla
prophylaxiemédicamenteuse.Deplus,despathologies
chroniquesetémergentesexistentenélevageetreprésentent
desperteséconomiquesimportantesquelaprofession
souhaiteréduire.Danscecontexte,améliorerlarobustesseet
larésistancedesanimauxauxpathogènesdevientunepriorité
pourlaplupartdesanimauxderente.Introduiredescritères
desantédanslesfutursschémasdesélectionpourproduire
desanimauxglobalementplusrésistantsàdespathologies
diversesestundesenjeuxmajeursdelaprochainedécennie.
L’identificationetl’utilisationdecritèresquicaractérisentune
multi‐résistancedesanimauxàdesagentspathogènesvariés
estunequestioncomplexe.Plusieursoptionssontpossibles
pouraborderlesujet.Parmicesoptions,deuxapproches
peuventêtredistinguées:explorerlesrésistancesdel’hôteaux
agentspathogènesoucaractériserl’immunocompétencedes
animauxsansciblerunagentpathogèneparticulier. Cette
dernièreapprocheestsous‐tendueparunehypothèseforte
selonlaquelleleniveaud’immunocompétencepourraitêtre
uncritèreprédictifderésistance/susceptibilitéauxinfections.
Caractériserl’immunocompétencesignifiechoisirdes
paramètresdelaréponseimmunitaire(RI)considérésensuite
commedescritèresdesanté(WilkieetMallard,1999,2000).
Cesdernierspourraientêtreensuiteintroduitsdansles
objectifsdesélection.Poureffectuercechoix,ilestnécessaire,
d’unepart,d’identifierdesparamètresimmunshéritableset
positivementcorrélésaveclasantéetlarésistanceaux
maladieset,d’autrepart,d’analyserlescorrélationsavecles
paramètreszootechniquessoussélection.Quelquesétudes
ontdéjàmontrédeshéritabilitésmoyennesàélevéespour
plusieursparamètresdelaRI,notammentlenombrede
leucocytesetlesproportionsdesous‐typesleucocytaires
(Edfors‐Liljaetal.,1994;Henryonetal.,2006;Clappertonet
al.,2008,2009),l’hypersensibilitéretardée(Jolingetal.,1993;
WilkieetMallard,2000),laproliférationlymphocytaireetla
productiondescytokinesIL2etIFNαaprèsstimulation
leucocytaireainsiquelaphagocytose(Edfors‐Liljaetal.,1994),
lestauxd’anticorpstotauxouspécifiques(Jolingetal.,1993;
Edfors‐Liljaetal.,1994;WilkieetMallard,2000;Henryonet
al.,2006)etdesprotéinesdelaphaseinflammatoireaiguë
(Henryonetal.,2006;Clappertonetal.,2008).
Unesélectiondivergentebaséesurunindexdequatre
paramètresaétéréaliséeavecsuccèssurhuitgénérations
chezleporc(Mallardetal.,1998;WilkieetMallard,1999)et
desQTLcontrôlantlenombretotaldeleucocytes,la
proliférationlymphocytaireinduitepardesagentsmitogènes,
laproductiond’IFNγetd’IL10ainsiqueletauxd’anticorps
spécifiquesontétéidentifiés(Edfors‐Liljaetal.,1994;
Wattrangetal.,2005;Luetal.,2010).
L’ensembledecesdonnéessuggèrequ’ilestenvisageablede
prendreencomptedesparamètresdelaRIdansdes
programmesdesélectionchezleporc.
Nousavonsdémarréunprogrammed’étudede
l’immunocompétencedeporcsLargeWhitesuiteàune
vaccination.L’objectifestdecouvrirunelargegammede
paramètresquicaractérisentlesRIinnéesetadaptativespour
unemêmepopulationd’animaux.Leprojetneciblepasun
agentpathogèneparticulieretassociedesapprochesde
génétiqueetdegénomiquefonctionnelle.L’approche
génétiquereposesuri)lephénotypagedesanimauxpourdes
paramètresvariésetcomplémentairesdelaRI,ii)une
estimationdel’héritabilitédesparamètresmesurésetiii)une
estimationdescorrélationsphénotypiquesetgénétiquesentre
lesparamètresimmunsetentrelesparamètresimmunsetdes
caractèresdeproduction.L’approchedegénomique
fonctionnellereposesurl’analysedutranscriptomedes
cellulesdusangtotalenutilisantunepuceàADNporcine
génériqueenrichieengènesdelaRI(Gaoetal.,2010).
Nospremiersrésultatssurl’estimationdeshéritabilitésetle
transcriptomesontprésentésici.
1. MATERIELETMETHODES
1.1. Animauxetprélèvements
Untotalde443mâlescastrésderaceLargeWhite,
appartenantàunelignéefemelleencontrôledeperformances
dansuneunitédetestageaétéinclusdansl’étude.
Lesanimauxsontnésdansdesélevagesdesélectionet
transférésdansl’unitédetestageà35jours,sansvaccination
préalable.Lelendemaindeleurarrivéeenstation,lesanimaux
ontétévaccinéscontreMycoplasmahyopneumoniae(vaccin
Stellamune,Pfizer,uneseuleinjection).L’arrivéedesanimaux
etlesprélèvementssesontdérouléssurunepériodede18
mois.Lapopulationestconstituéede307famillesnucléaires
issuesde106verrats.Touslesporcsétaientexemptsdesignes
cliniquesextérieursd’infection.Lesmesuresréaliséesen
routinesurlesanimauxencontrôledeperformancesontété
enregistréspourtouslesanimaux:paramètresdecroissance
etdecarcasse(n=19)etdequalitédeviande(n=6).
Deséchantillonsdesangontétéprélevéstroissemainesaprès
vaccinationparponctionauniveaudelaveinejugulaire
externeetcollectéssoitdansdestubessansanti‐coagulant,
soitdansdestubescontenantdel’EDTAoudel’héparinede
sodium.Dusangaégalementétéprélevédansdestubes
PAXgeneTM(PreAnalytiX).Lesprotocolesétaientenaccord
aveclaréglementationenvigueursurl’expérimentation
animale.
1.2. Paramètresimmunsmesurés
Lalistedesparamètresmesurésestprésentéedansletableau1.
LedétaildesprotocolesestdécritdansFlorietal.(soumis).
1.3. Analysesgénétiques
Desanalysesstatistiquespréliminairesontétéréaliséesà
l’aidedulangageR(R2.8.1,http://www.R‐project.org).
Lesmesuresdesparamètresontéténormaliséesparune
transformationboxcoxouln(1+x).Leseffetsdesvariablesâge
aumomentdelavaccination,jourdevaccination,élevagede
naissanceetjourdesprélèvementsontététrouvéssignificatifs
pourlaplupartdescaractèresmesurés(ANOVA,R2.8.1,
http://www.R‐project.org).Lescomposantesdelavariance,
lesparamètresgénétiquesetleurserreursstandardsontété
estimésenutilisantlaméthodologieREML(Pattersonet
Thompson,1971)appliquéeàunmodèlelinéairemixte.
2011. Journées Recherche Porcine, 43.
28

Tableau1‐Phénotypesmesurésdontl’estimationdel’héritabilitéestsignificative.
Typologiedes
mesuresdeRIProtocolesimplifiéParamètresmesurésh2(se)h2:Intervallede
confianceà95%
RIglobaleNumérationformulesanguineGlobulesblancs(GB)0,7(0,2)[0,3‐1]
Lymphocytes 0,7(0,2)[0,3‐1]
RIadaptativeà
médiationcellulaire
Stimulationinvitrodesangtotal
avecunmélangedephorbol
myristateacetate(PMA)et
ionomycine(PMAIONO)ou
lipopolysaccharide(LPS)ou
concanavalineA(CONA)pendant
48heures
IL4‐CONA0,5(0,1)[0,3‐0,6]
IL4‐PMAIONO0,6(0,2)[0,2‐0,9]
IL4‐LPS0,1(0,1)[0‐0,2]
IL10‐CONA0,5(0,2)[0,1‐0,8]
IL10‐PMAIONO0,9(0)[0,9‐0,9]
IL10‐LPS0,3(0,1)[0,1‐0,4]
IFNG‐CONA0,4(0,1)[0,2‐0,5]
IFNG‐PMAIONO0,5(0,2)[0,1‐0,8]
IFNG‐LPS0(0)[0‐0]
IL2‐CONA0,8(nd1)nd1
IL2‐PMAIONO0,7(0,1)[0,5‐0,8]
IL2‐LPS0,9(0)[0,9‐0,9]
Proliférationlymphocytairenon
spécifique:stimulationinvitrode
sangtotalpendant48heures
PROLIF‐CONA0,3(0,1)[0,1‐0,4]
PROLIF‐PMAIONO0,2(0,1)[0‐0,3]
PROLIF‐LPS0,3(0,1)[0,1‐0,4]
Cytométriedeflux
CD4‐CD8+(LymphocytesTαβ)0,4(0,2)[0‐0,7]
CD4+CD8+(LymphocytesTαβ)0,6(0,2)[0,2‐0,9]
CD4+CD8‐(LymphocytesTαβ)0,5(0,1)[0,3‐0,6]
RIadaptativeà
médiationhumorale
CytométriedefluxIgM+(lymphocytesB)0(0)[0‐0]
Anticorpstotaux
IgA 0,6(0,1)[0,4‐0,7]
IgG 0,6(0,2)[0,2‐0,9]
IgM 0,3(0,1)[0,1‐0,4]
AnticorpsspécifiquesIgGanti‐Mycoplasma(IgG‐Mh)0,1(0,1)[0‐0,2]
RIinnée
Numérationformulesanguine
Monocytes0,3(0,2)[0‐0,6]
Neutrophiles0,5(0,2)[0,1‐0,8]
Eosinophiles0,8(0,1)[0,6‐0,9]
Cytométriedeflux
CD16‐CD2+0,3(0,2)[0‐0,6]
CD16+CD2+0,8(0,1)[0,6‐0,9]
CD16+CD2‐0(0,1)[0‐0,1]
MHCII‐CD172+0,2(0,2)[0‐0,5]
MHCII+CD172+0,4(0,2)[0‐0,7]
MHCII+CD172‐0,3(0,2)[0‐0,6]
CD16‐CD172+0,3(0,2)[0‐0,6]
CD16+CD172+0,2(0,2)[0‐0,5]
CD16+CD172‐0,7(0,2)[0,3‐1]
CD16‐MHCII+0(0,1)[0‐0,1]
CD16+MHCII+0,7(0,1)[0,5‐0,8]
CD16+MHCII‐0,1(0,2)[0‐0,4]
TCRγδ+(LymphocytesTγδ)0,6(0,1)[0,4‐0,7]
Stimulationinvitrodesangtotal
avecunmélangedePMA,
ionomycineetLPSpendant24
heuresouavecunantigèneviral
pourl’interféronalpha(IFNA)
IL1B 0,1(0,1)[0,1‐0,2]
IL8 0(0)[0‐0]
TNF 0(0)[0‐0]
IL6 0,1(0,1)[0‐0,2]
IL120,5(0,2)[0,1‐0,8]
IFNA 0,6(0,2)[0,2‐0,9]
TestinvitrosursangtotalPhagocytose0,6(0,2)[0,2‐0,9]
Phaseinflammatoire
aiguëProtéinessériquesHaptoglobine0,5(0,1)[0,3‐0,6]
ProtéineCréactive0,1(0,1)[0‐0,2]
Autresparamètres
sanguinsNumérationformulesanguine
Tauxdeglobulesrouges0,4(0,1)[0,2–0,5]
Hématocrite0,5(0,1)[0,3–0,6]
Indicededéviationdesréticulocytes0,5(0,1)[0,3–0,6]
Plaquettes0,5(0,1)[0,3–0,6]
1ND:nondéterminé
2011. Journées Recherche Porcine, 43.
29

univariéàl’aidedulogicielASReml(Gilmouretal.,2004),en
prenantencomptecommeeffetsfixesl’âgeaumomentdela
vaccination,lejourdevaccination,l’élevagedenaissanceetle
jourdesprélèvementsetcommeeffetsaléatoireslabandede
contrôleetleseffetsgénétiquesdirects.Pourl’estimationdes
héritabilités,unintervalledeconfianceà95%aétécalculé.
1.4. ExtractiondesARNetanalysesdutranscriptome
DesARNtotauxontétéextraitsàpartirdesprélèvements
réalisésavecdestubesPAXgeneàl’aidedukitPAXgeneBlood
RNAKit(Qiagen,France).Lapucegénériqueporcineenrichie
engènesdelaRI(puceSLA‐RI/NRSP8‐13K)aétéutilisée(Gao
etal.,2010).Cinqmicrogrammesd’ARNsanguinoud’unARN
deréférenceissud’unmélangedetissusontété
respectivementmarquésparduCy3ouduCy5,directement
aucoursdelaréversetranscriptionaveclekitChipShotTM
DirectLabelingSystem(Promega,USA).Aprèspurification
(ChipShotTMMembraneClean‐UpSystem,Promega,USA),750
ngdesADNcciblesmarquéssoitenCy3soitenCy5ontété
mélangésetco‐hybridéssurlespuces.Lessignaux
d’hybridationontétécapturésavecunscannerAgilentDNA
MicroarraypuisquantifiésaveclelogicielGenePixTMPro
softwareV6.0(MDSInc.,Canada).Lesniveauxdifférentiels
d’expressionentreéchantillonsontétéétablisavecdestests
statistiquesdisponiblesdanslaversion2.12.0deLimma
(Bioconductor,R2.8.1,http://www.R‐project.org).
2. RESULTATSETDISCUSSION
2.1. PhénotypesdelaRIétudiés
Untotalde54paramètrescouvrantunlargeéventailde
mesuresreliéesauxRIinnéesetadaptativesaétéenregistré
(Tableau1).Lespopulationscellulairesdusangontété
quantifiéespardesnumérations‐formulessanguinesetdes
sous‐populationsleucocytairesontétécaractériséespar
cytométriedeflux.LescellulesassociéesàlaRIinnée
correspondentauxlymphocytesTγδ (LTγδ),monocytes,
cellulesnaturalkiller,neutrophilesetéosinophiles.Lescellules
impliquéesdanslaRIadaptativeincluentleslymphocytesTαβ
(LTαβ)CD4+CD8+,CD4+CD8‐,CD4‐CD8+etleslymphocytesB
(LB).LaRIinnéeestégalementcaractériséeparlacapacité
phagocytaire,laproductioninvitrodescytokinesIL1B,IL6,IL8,
TNF,IL12,IFNα.S’ajoutentledosagededeuxprotéinesdela
phaseinflammatoireaiguë,l’haptoglobineetlaprotéineC
réactive.LaRIadaptativeàmédiationhumoraleest
représentéeparlesmesuresd’anticorpstotaux(IgA,IgMet
IgG)etspécifiquesanti‐Mycoplasmahyopneumoniae(IgG‐Mh).
LaRIadaptativeàmédiationcellulaireinclut,d’unepart,le
dosagedecytokinesproduitesinvitroquispécifient
classiquementlesvoiesTh1(IL2etIFNγ)ouTh2(IL4etIL10)et,
d’autrepart,lamesuredelaproliférationnonspécifiquedes
cellulessanguinespardesmitogènesvariés(tableau1).
2.2. Estimationdeshéritabilités
Lesrésultatsdescalculsmontrentqueleshéritabilitéssont
estiméesmodéréesàélevées(h²≥0,2)pour42paramètresdes
54inclusdansl’analyse(tableau1).Deshéritabilités
significativesontétéobservéespourdesparamètresissusde
dosagesvariéstelsdesdosagessériques,descomptages
cellulaires,desmesuresdeprotéinesaprèsstimulationinvitro.
Celasuggèrequelesrésultatsnesontpasbiaisésversuntype
particulierdemesureetquedesparamètresassociésàdes
voletsdistinctsetcomplémentairesdelaRIsonthéritables.
Nosrésultatssontenaccordavecd’autresétudesaucours
desquellesdeshéritabilitésontétéestimées(Edfors‐Liljaetal.,
1994;WilkieetMallard,1999;Clappertonetal.,2008,2009)
etsontcohérentsavecl’identificationdeQTLspourdes
caractèrescommelenombredeleucocytes(Edfors‐Liljaetal.,
1998;Wattrangetal.,2005),laréponseanticorps(Wimmers
etal.,2008,2009)etplusrécemmentlesniveauxsériquesdes
cytokinesIL10etIFNγsuiteàunevaccinationcontrelevirusde
lagrippeporcine(Luetal.,2010).
IlestbienreconnuquelaRIestfortementdépendantede
l’environnement.Nosrésultats,associésàceuxd’autres
équipes,montrenttoutefoisquelapartgénétiquedelaRIse
confirme.Lenombrecroissantdedonnéessurlesestimations
d’héritabilitéetladétectiondeQTLsuggèrequ’unnombre
significatifdeparamètresàdespositionsdistinctesdela
complexecascadedelaRIsontcontrôlésgénétiquement.
2.3. Etudedutranscriptomedusangtotal
Pourchaqueparamètre,lesanimauxontétéclassésparniveau
devaleursmesurées.Lesanimauxlesplusextrêmespourles
valeursélevéesoubassesontétéchoisispourl’analysedu
transcriptomedescellulessanguines.Commeindiquédansla
partieMatérieletMéthodes,leséchantillonssanguinsétudiés
pourlesanalysesdutranscriptomeontétérécoltésenmême
tempsqueceuxdestinésauxmesuresphénotypiquesdelaRI,
afind’avoirunecorrespondancedirecteentrelesmesuressur
lesquellesdeshéritabilitéssontestiméesetlesanalysesdu
transcriptome.Unpremiergroupedeneufparamètresaété
analysé(Tableau2).
Tableau2‐BilandesanalysesdutranscriptomedescellulesdusangtotalpourneufparamètresdelaRI
Phénotypes
NombredeporcsNombredegènesdifférentiellementexprimés
Groupevaleurs
fortes
Groupevaleurs
bassesTotal
Groupevaleursbasses
>
groupevaleursélevées
Groupevaleursbasses
<
groupevaleursélevées
GB9933423599
PHAG911336125211
IL10‐PMAIONO1010756539217
IL2‐PMAIONO1010642312330
TNF77000
IFNG‐PMAIONO781009606403
IgG‐Mh1010000
TCRγδ+1010000
CD4‐CD8+4617311162
2011. Journées Recherche Porcine, 43.
30

Unnombresignificatifdegènesaététrouvé
différentiellementexpriméentrelesgroupesdevaleurs
élevéesetbassespourlesparamètresGB,PHAG,IL10‐
PMAIONO,IL2‐PMAIONOetLTCD4‐CD8+;lesanalysesen
coursindiquentunebonnepertinencebiologiquedesgènes
identifiés,avecunereprésentationsignificativedefonctions
liéesàl’immunitéetàl’hématopoïèse.Al’inverse,aucune
différenceentrelesdeuxgroupesn’aététrouvéepourles
paramètresTNF,IgG‐MhetTCRγδ+.Cesrésultatspréliminaires
montrentque,pourunepartiedesparamètresdelaRI,le
transcriptomedusangserainformatifetquedesprofils
d’expressiongéniquepourraientêtrecorrélésàdesniveauxde
mesures.
CONCLUSION
Anotreconnaissance,cetteétudeestlapremièreétudeà
grandeéchellequirassembledesdonnéessurautantde
paramètresdesRIinnéesetadaptativespourunemême
populationd’animauxetquicombinedesapprochesde
génétiqueetdegénomiquefonctionnelle.Nosrésultats
confirmentceuxd’autreslaboratoiresquantàuncontrôle
génétiquesignificatifd’unnombreimportantdeparamètres
immuns,bienquel’environnementsoitreconnucommeayant
uneinfluencetrèsfortesurlaRI.
Desdonnéespréliminairessurl’analysedutranscriptomedu
sangsuggèrentqueceparamètrepourraitêtreunphénotype
moléculaireprometteurpouraffinerlacaractérisationdelaRI
chezleporc,etquel’intégrationdesdonnéesde
transcriptomeetdegénétiquepourraitcontribueràidentifier
lesgènescandidatsimpliquésdanslecontrôledesparamètres
deRItrouvéshéritables.
L’analysedescorrélationsphénotypiquesetgénétiquesentre
caractèresimmunsetdeproductionestencours.Lespremiers
résultatsmettentenévidencedescorrélationspositiveset
négativesdontlasignificationbiologiqueestàexplorerplus
avant.L’étapesuivanteseral’étudedescorrélationsentre
immunocompétenceetrésistanceauxinfections.
REFERENCESBIBLIOGRAPHIQUES
• ClappertonM.,GlassE.J.,BishopS.C.,2008.Pigperipheralbloodmononuclearleucocytesubsetsareheritableandgeneticallycorrelatedwith
performanceAnimal,2,1575‐1584.
• ClappertonM.,DiackA.B.,MatikaO.,GlassE.J.,GladneyC.D.,MellencampM.A.,HosteA.,BishopS.C.,2009.Traitsassociatedwithinnateand
adaptiveimmunityinpigs:heritabilityandassociationswithperformanceunderdifferenthealthstatusconditions.GenetSelEvol,41,54.
• Edfors‐LiljaI.,WattrangE.,MagnussonU.,FossumC.,1994.Geneticvariationinparametersreflectingimmunecompetenceofswine.Vet
ImmunolImmunopathol,40,1‐16.
• Edfors‐LiljaI.,WattrangE.,MarklundL.,MollerM.,Andersson‐EklundL.,AnderssonL.,FossumC.,1998.Mappingquantitativetraitlocifor
immunecapacityinthepig.JImmunol,161,829‐835.
• GaoY.,FloriL.,LecardonnelJ.,EsquerreD.,HuZ.L.,TeillaudA.,LemonnierG.,LefevreF.,OswaldI.P.,Rogel‐GaillardC.,2010.Transcriptome
analysisofporcinePBMCsafterinvitrostimulationbyLPSorPMA/ionomycinusinganexpressionarraytargetingthepigimmuneresponse.
BMCGenomics,11,292.
• GilmourA.R.,CullisB.R.,WelhamS.J.,ThomsonR.,2004.ASRemlreferencemanual2ndedition,release2.0(draft1.62).In:NSWAgriculture
BiometricalBulletin3.
• HenryonM.,HeegaardP.M.H.,NielsenJ.,BergP.,Juul‐MadsenR.,2006.Immunologicaltraitshavethepotentialtoimproveselectionofpigs
forresistancetoclinicalanssubclinicaldisease.AnimalScience,82,597‐606.
• JolingP.,MokK.S.,deVriesReilinghG.,WeverP.J.,CornelisR.S.,OskamJ.P.,HenkenA.M.,1993.Anevaluationofimmunecompetencein
differentswinebreeds.VetQ,15,9‐15.
• LuX.,GongY.F.,LiuJ.F.,WangZ.P.,HuF.,QiuX.T.,LuoY.R.,ZhangQ.,2010.Mappingquantitativetraitlociforcytokinesinthepig.Anim
Genet.
• MallardB.A.,WilkieB.,KennedyB.,GibsonJ.,QuintonM.,1998.Immuneresponsivenessinswine:eightgenerationsofselectionforhighand
lowimmuneresponseinyorkshirepigs.Proc.Conference"6thWCGALP".pp.257‐264.
• PattersonH.D.,ThompsonR.,1971.Recoveryofinterblockinformationwhenblocksizesareunequal.Biometrika,58,545–554.
• WattrangE.,AlmqvistM.,JohanssonA.,FossumC.,WallgrenP.,PielbergG.,AnderssonL.,Edfors‐LiljaI.,2005.ConfirmationofQTLonporcine
chromosomes1and8influencingleukocytenumbers,haematologicalparametersandleukocytefunction.AnimGenet,36,337‐345.
• WilkieB.N.,MallardB.A.,1999.Selectionforhighimmuneresponse:analternativeapproachtoanimalhealthmaintenance?VetImmunol
Immunopathol,72,231‐235.
• WilkieB.N.,MallardB.A.,2000.Geneticaspectsofhealthanddiseaseresistanceinpigs.In:R.F.E.Axford,S.C.Bishop,F.W.Nicholas&O.J.B.
(Eds),Breedingfordiseaseresistanceinfarmanimals,2ndedition,379‐396.CABI.
• WimmersK.,MuraniE.,SchellanderK.,PonsuksiliS.,2009.QTLfortraitsrelatedtohumoralimmuneresponseestimatedfromdataofa
porcineF2resourcepopulation.IntJImmunogenet,36,141‐151.
• WimmersK.,JonasE.,SchreinemachersH.J.,TesfayeD.,PonsuksiliS.,TholenE.,JuengstH.,SchellanderK.,PhatsaraC.,2008.Verificationof
chromosomalregionsaffectingtheinnateimmunityinpigsusinglinkagemapping.DevBiol(Basel),132,279‐286.
2011. Journées Recherche Porcine, 43.
31
 6
6
1
/
6
100%