programme module transversal m2 « genomique » du 02/11/2010

PROGRAMME MODULE TRANSVERSAL M2 « GENOMIQUE » DU 02/11/2010 AU 05/11/2010
Locaux
le 2 et 3 novembre: Amphi HE1 (Hôpital Timone ENFANTS)
le 4 novembre: Amphi HE2 (Hôpital Timone ENFANTS)
le 5 novembre: un amphi sera réservé à la faculté de Pharmacie (détails en attente).
Responsable du Module : Martin Krahn martin.krahn@univmed.fr Tel : 04 91 78 68 94
Horaires
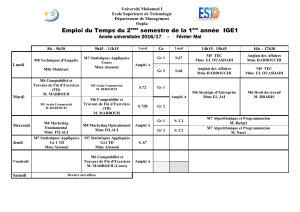
Mardi 02.11.2010
Mercredi 03.11.2010
Jeudi 04.11.2010
Vendredi 05.11.2010
9h-10h
Pas de cours
Pas de cours
Pas de cours
9h30. Pr J.L. MEGE
Présentation générale
du M2 Pathologie
Humaine et modalités
de candidature à
l’allocation de
recherche
10h-11h
Organisation du
génome humain.
J. Imbert
(1h) [GH]
Interactome protéique
M. Bartoli
(2h) [GH]
Modèles animaux
M. Aurrans-Lyons
(1h) [O]
11h-12h
Pas de cours
Les microARNs
P. Cau
(1h) [GH]
Création de Modèles
animaux
P. Roubertoux
(1h) [GH]
12h-13h : PAUSE

13h-14h
Analyses génomiques
en pathologie
humaine
M. Krahn
(2h) [GH]
ARN interférant
Dr. C. Pellegrino
(1h) [GH]
Séquençage des
génomes des
microorganismes
C. Robert
(1h) [MT]
Approches
thérapeutiques pour
les Maladies
Génétiques
M. Bartoli
(1h) [GH]
14h-15h
Transcriptome et
cancer
F. Bertucci
(2h) [O]
Analyse du génome
tumoral à grande
échelle: multi-FISH et
CGH array
M. Chaffanet
(1h) [O]
Thérapeutique
J.M. Rolain
(1h) [MT]
15h-16h
Bio-informatique du
transcriptome
D. Puthier
(2h) [O]
Prédispositions
héréditaires à la
carcinogenèse
S. Olschwang
(1h) [O]
Cibles thérapeutiques
et cancer
L. Xerri
(1h) [O]
16h-17h
Identification des
pathogènes
M. Drancourt
(1h) [MT]
Pas de cours
Thérapeutique anti-
EGFR
A. Goncalves
(1h) [O]
GH : Génétique Humaine
MT : Maladies transmissibles
O : Oncologie
1
/
2
100%