Conception d`un outil d`aide à la segmentation semi

Sujet de thèse : IGCNC – EA 7282 / UMR CNRS 6284 ISIT
Conception d'un outil d'aide à la segmentation semi-automatique supervisé
de structures cérébrales
Lieu : Clermont-Ferrand (63)
IGCNC, 2e HC – CHU Clermont-Ferrand
Encadrants : Pr Fabien Feschet (27e section) [75 %]
Pr Jean-Jacques Lemaire (PUPH Neurochirurgie) [25 %]
Sujet
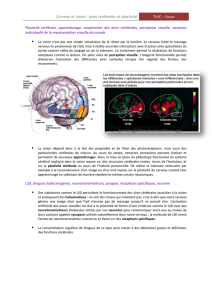
Dans le cadre du service de neurochirurgie du CHU Gabriel-Montpied de
Clermont-Ferrand, l'équipe IGCNC a acquis une expérience reconnue pour la segmentation
des structures cérébrales profondes. Elle a ainsi constitué une base de données des
structures cérébrales impliquées dans les différents traitements, de la maladie de
Parkinson notamment. Ces éléments combinés à la tractographie via une imagerie DTI sont
utilisés quotidiennement pour le planning et la réalisation des implantations (Deep Brain
Stimulation). Depuis 2005, la volumétrie des interventions a permis de déterminer la
segmentation manuelle de plus de 3700 structures cérébrales profondes. Cette
extraction a été rendu possible par la mise au point par les membres de l'équipe d'une
séquence d'acquisition IRM particulière, appelée WAIR, qui permet sur une IRM à 1,5 T de
déterminer plus aisément les contours des structures cérébrales profondes.
A partir de cette base de données, les travaux actuels de l'équipe IGCNC porte sur
la détermination de la statistique de forme et de positionnement des différentes
structures mises en jeu. L'objet de cette thèse est d'aider à la diffusion de l'utilisation de
cette base de données et de permettre sa mise en œuvre dans le cadre de la médecine
personnalisée, notamment lors d'une étude qui démarre prochainement sur la DBS
post-coma. La mise à disponibilité d'un outil d'aide à la segmentation est un point-clé et un
verrou dans l'exploitation efficace de la base de données IGCNC. L'objet de cette thèse
est donc de construire et mettre en œuvre un prototype logiciel permettant une telle
exploitation. Ce prototype pourra ensuite servir de démonstrateur pour une future
intégration dans les logiciels cliniques de neurochirurgie via le partenariat étroit avec la
société BrainLab.
La difficulté majeure de ce travail provient de la nécessaire gestion de la géométrie
théorique – définie sur l'acquisition d'un véritable cerveau sain - et de l'adaptation de cette
géométrie en tenant compte de la variabilité intrinsèque à chaque individu. Cette
problématique nécessite donc de mettre en œuvre du recalage géométrique et de l'analyse
d'images simultanément. Cette dernière étape devra également mettre en œuvre des
outils de fusion avec des données DTI permettant d'éclairer l'anatomie locale de chaque
patient. Qui plus est, l'objectif est l'utilisation du prototype dans des cas cliniques,

c'est-à-dire pour des patients qui ont subis des déformations ou des détériorations plus
ou moins grandes des structures cérébrales. La disparition complète de structures
cérébrales doit ainsi être prise en compte dès la conception de la méthode.
Le candidat devra dans un premier temps procéder à une analyse de l'état de l'art
dans le cadre d'une mise en correspondance rigide – la seule disponible dans la plupart
des outils cliniques – et de déterminer comment ce positionnement devra être modifié
dans le cadre d'un recalage élastique – prochainement disponible dans les logiciels
cliniques -. Une partie des structures cérébrales devra être considérée comme altérées ou
manquantes. Dans un second temps, le candidat devra procéder à une déformation locale
du recalage géométrique pour proposer des contours adaptés à l'imagerie individuelle de
chaque patient. La méthode devra prévoir de classifier les contours en fonction
d'estimation d'une probabilité de forme correcte. Comme dans toute méthode
semi-automatique, un soin particulier devra être apporté à l'interface-homme machine.
Cette dernière sera testée par les neurochirurgiens du CHU Gabriel-Montpied. L'objectif à
atteindre dans ce travail est la présentation du prototype sur des cas réels rencontrés
dans le service hospitalier.
Mots-clés : Segmentation semi-automatique supervisée ; structures cérébrales
profondes ; IRM structurelle ; DTI
1
/
2
100%