Bioinformatique : TD 5

Bioinformatique : TD 5
Analyse d’une protéine d’intérêt biotechnologique : la GFP
Introduction
La protéine fluorescente verte ou GFP (Green Fluorescent
Protein) est une protéine produite par une méduse (Aequorea
victoria)ayant la propriété d'émettre une fluorescence de
couleur verte. Son gène a été cloné et peut être fusionné in-
vitro au gène d'une protéine que l'on souhaite étudier. Le
gène recombinant réintroduit dans des cellules va alors
synthétiser la protéine de fusion, fluorescente. Ainsi, à l’aide
d’un microscope à fluorescence, il est possible d’effectuer
une analyse du profil d’expression de la protéine fusion. On
peut ainsi déterminer dans quels tissus est exprimée la
protéine d’intêret en recherchant la fluorescence. Une analyse
quantitative est également possible en dosant la
fluorescence émise à l’aide d’un fluorimètre. La découverte
et les applications de la GFP ont été couronnés par le prix
Nobel de chimie décerné le mois dernier à Osamu
Shimomura, Martin Chalfie et Roger Tsien (octobre 2008).
La GFP a été décrite pour la première fois en 1962. Elle est
constituée de 238 acides aminés. Le chromophore (centre
actif responsable de la fluorescence) est constitué par les
chaînes latérales d'une glycine, une tyrosine et une sérine.
Récemment différentes protéines mutantes de la GFP ont
été produites. Elles apportent des modifications d'ordre
quantitatif comme une augmentation de l'intensité de la fluorescence, ou qualitatif comme
l'apparition de formes fluorescentes jaunes, bleues ou rouges.
Ici nous étudierons la protéine GFPuv dont l'intensité de fluorescence est 18 fois celle de
la protéine GFP sauvage, lui conférant une sensibilité beaucoup plus importante. Nous
nous proposons donc de retrouver par analyse bioinformatique, les modifications qui
ont été apportées au gène sauvage.
La GFPuv a été réalisée par génie génétique par la société Clontech qui commercialise un
plasmide pGFPuv renfermant le gène muté.
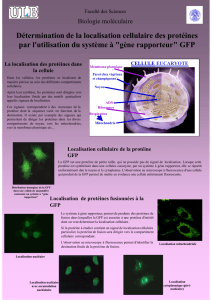
Figure 1 : Sructure tridimentionnelle de la GFP, et mise en évidence du profil d’expression
d’un gène, après réalisation d’une protéine fusion, avec expression préférentielle dans les
corps pédonculés du cerveau de drosophile, structure impliquée dans la mémoire.
I- Recherche des séquences et comparaison.
1.1- Récupération des séquences.
Sur le Site du NCBI :
- Retrouver la séquence de la GFP mutée (GFP-uv) clonée dans pGFP-uv.
- Copier la séquence du gène GFP-uv, en format FASTA, dans un document Word.
- La nommer : « CDS GFP-uv »
- Retrouver la séquence complète du plasmide pGFP (la séquence CDS, n’est pas
disponible)
Figure 1

- Copier la séquence du plasmide, en format BRUT, dans un document Word.
- La nommer : « séquence complète pGFP »
1.2- Comparaison des séquences nucléotidiques : BLASTN
Le logiciel BLAST permet de comparer deux séquences entre elles ou une séquence par
rapport à l’ensemble des séquences d’une base données (cf cours p.10)
Sur le Site du NCBI :
- Cliquer sur l’onglet BLAST, dans la barre horizontale de menu
- Choisir le programme « Nucléotide BLAST »
- Sélectionner la comparaison de deux séquences
- Dans la fenêtre « QUERY sequence » : coller la séquence CDS GFP-uv
- Dans la fenêtre « SUBJECT sequence » : coller la séquence complète pGFP
- Cliquer sur « BLAST »
- Copier l’alignement de séquence (fenêtre « aligment ») et coller dans votre document
word. Nommer le document : BLAST N : Query : GFPuv / Subject pGFP
- Grâce à l’alignement réalisé, identifier la position du gène GFP-wt dans pGFP et
enregistrer la séquence correspondante dans le document word sous le nom : « CDS
GFP-wt »
1.3- Comparaison des séquences protéiques : TBLASTX
Le logiciel TBLASX permet de comparer deux séquences protéiques entre elles (ou une
séquence par rapport à l’ensemble des séquences d’une base données) à partir de séquences
nucléotidiques. Le logiciel recherche des ORF à partir des différents cadres de lecture et
recherche des alignements possibles.
Sur le Site du NCBI :
- Revenir à la page d’accueil de BLAST, en cliquant sur « home » dans la barre
horizontale de menu
- Choisir le programme « TBLASTX »
- Sélectionner la comparaison de deux séquences
- Dans la fenêtre « QUERY séquence » : coller la séquence CDS GFP-uv
- Dans la fenêtre « SUBJECT sequence » : coller la séquence complète pGFP
- Cliquer sur « BLAST »
Copier l’alignement de séquence le plus pertinent (généralement le premier proposé et coller
dans votre document word. Nommer le document : TBLASTX : Query : GFPuv / Subject pGFP
NB : Organiser le doc word, de façon à ce que chaque séquence ou comparaison ne soit pas
présentée sur deux pages, et que la césure des lignes soit respectée. Surligner les titres des
différents documents. Imprimer l’ensemble.
II- Analyse des séquences.
1.1- Analyse préliminaire
A partir du document papier de la comparaison nucléotidique :
- Faire apparaître, en les surlignant proprement en fluo, toutes les différences entre les
deux séquences (« mismaches »)
A partir du document papier de la comparaison protéique :

- Sachant que la GFP est une protéine de 238 acides aminés, vérifier que l’intégralité de la
séquence est présente sur l’alignement (nombre d’AA, codon START, codon STOP…)
- Faire apparaître, en les surlignant proprement en fluo, toutes les différences entre les
deux séquences
Conclure sur le type de mutations mises en évidence en comparant les deux alignements.
1.2- Recherche des mutations impliquées dans l’augmentation de la fluorescence de la
protéine GFPuv.
- Combien de mutations d’acides aminés semblent être impliquées dans l’augmentation
de fluorescence ?
- Répertorier les mutations selon la nomenclature suivante :
A56L (XY) : 167
Avec : A : acide aminé d’origine (wt) en code une lettre
56 : position de l’acide aminé dans la protéine (1 = Methionine)
X : nucléotide d’origine (wt)
Y : nucléotide dans la séuquence mutée (uv)
167 : position du nucléotide muté (= 56*3 +/- 2)
1.3- Analyse des autres mutations
Afin de poursuivre l’étude la protéine GFPuv, on peut se demander pourquoi la société
Clontech a réalisé dans cette protéine mutée, d’autres mutations (……………………..) qui ne
semblent pas impliquées dans l’augmentation de la fluorescence.
Deux hypothèses majeures peuvent être émises :
1- Ces mutations permettent de créer des sites de restriction dans la protéine
2- Ces mutations permettent une optimisation des codons
1.3.1- Réalisation de cartes de restriction
Réaliser les cartes de restriction des deux séquences nucléotidiques et effectuer une
comparaison, selon la méthode suivante :
A partir de l’application « WATCUT » hébergée par l’université de Waterloo
(http://watcut.uwaterloo.ca/watcut/watcut/template.php) :
- Coller tour à tour les deux séquences nucléotidiques (CDS GFP-wt et CDS GFP-uv)
dans la fenêtre prévue à cet effet.
- Dans le menu « select enzymes », sélectionner uniquement les enzymes dont les sites
de reconnaissance sont à 6 pb, et exclure les sites dégénérés et non-palindromiques.
- Lancer la recherche des sites.
- Dans la présentation du résultat, sélectionner les enzymes qui ne coupent qu’une fois,
mettre à jour (« Update Display ») et demander une version imprimable (Printer
Friendly Version).
- Copier et coller dans votre doc word, en mettant un titre pour chaque analyse.
- Comparer les résultats. Combien de nouveaux sites sont apparus dans la séquence
GFP-uv ?
- Localiser ces sites (associés aux mutations) sur l’alignement de séquences
nucléotidiques (BLAST N : Query : GFPuv / Subject pGFP)

1.3.2- Recherche d’une optimisation de codons
A partir du document : BLAST N : Query : GFPuv / Subject pGFP :
- Parmi les mutations qui restent et dont la fonction demeure inconnue, rechercher des
motifs de trois nucléotides que l’on retrouve d’une mutation à l’autre.
- Ces motifs de trois nucléotides ou codons, sont-ils dans la phase codante ?
- Si oui retrouver, pour la protéine sauvage, et pour la protéine mutée, l’acide aminé
codé par ces différents codons.
- Conclure.
III- Synthèse.
Faire apparaitre sur le document BLAST N : Query : GFPuv / Subject pGFP , toutes les
mutations, avec le code couleur suivant :
- Rouge : augmentation de la fluorescence
- Bleu : nouveaux sites (les nommer)
- Vert : optimisation de codons
- Noir : Eventuelles mutations à rôle non déterminé
1
/
4
100%