1. Le xénope : un organisme de choix pour l`étude du

Introduction
1
1. Le xénope : un organisme de choix pour l’étude du
développement embryonnaire
1.1. Généralités
L’étude du développement embryonnaire nécessite l’utilisation d’organismes adaptés aux
manipulations expérimentales. Au cours du siècle précédent, différents organismes
« modèles » ont été étudié par des approches classiques d’embryologie expérimentale et de
génétique. Récemment, ces organismes « modèles » ont suscité un regain d’intérêt grâce à
l’adaptation des techniques les plus modernes de la biologie moléculaire et cellulaire à l’étude
de ces organismes. Actuellement, l’étude des mécanismes moléculaires contrôlant le
développement embryonnaire des animaux s’appuie sur des organismes tels que le poisson
zèbre, le xénope, le poulet et la souris chez les vertébrés ainsi que la drosophile,
Caenorhabditis elegans (un vers nématode) et l’ascidie chez les invertébrés. Chaque
organisme « modèle » comporte une série d’avantages et d’inconvénients qui permettent une
certaine complémentarité de ces différents modèles. L’embryon d’amphibien Xenopus laevis
présente de nombreux avantages : les adultes s’élèvent aisément en laboratoire, les œufs sont
disponibles en grandes quantités et ce tout au long de l’année, leur développement est rapide
et externe donnant ainsi accès à l’ensemble des stades de développement.
De nombreuses techniques et outils sont disponibles pour étudier la fonction des gènes
au cours du développement embryonnaire de ces organismes. Des expériences d’hybridation
in situ ou d’immunochimie sur embryons permettent de visualiser l’expression spatio-
temporelle des gènes au cours du développement. Chez le xénope, des expériences de
surexpression des gènes via la micro-injection d’ARNm ou d’ADN permettent une analyse
rapide de la fonction des gènes. Des expériences de sous-expression peuvent également être
réalisées par la micro-injection d’oligonucléotides de type morpholino bloquant la traduction
d’un ARNm cible ou son épissage ou par la surproduction de formes tronquées de protéines
fonctionnant comme «dominant négatif». De plus, ces techniques de sur- et sous-expression
permettent d’étudier les relations hiérarchiques entre les gènes ou d’interférer avec le
fonctionnement normal de plusieurs gènes simultanément, ce qui est par exemple beaucoup
plus difficile et long à réaliser chez d’autres organismes modèles tels que la drosophile ou la
souris. Plusieurs techniques rapides de transgenèse ont également été développées chez le
xénope (Kroll and Amaya, 1996; Pan et al., 2006). La transgenèse permet d’étudier la
régulation transcriptionnelle des gènes in vivo ainsi que de surexprimer un gène d’intérêt

Introduction
2
codant pour des formes sauvages ou mutantes de sa protéine tout en contrôlant son expression
spatio-temporelle. L’utilisation de la transgenèse permet également de la mutagenèse aléatoire
par insertion dans le génome (Bronchain et al., 1999). Enfin, la transgenèse permet la création
de lignées d’animaux où cetains tissus ou organes expriment spécifiquement la GFP, ce qui
facilite l’analyse des phenotypes provoqués par des expériences de sur- et sous-expression.
Un désavantage majeur de l’amphibien Xenopus laevis est qu’il ne se prête pas aux études
génétiques à cause de la tétraploïdie de son génome et de son temps de génération
relativement long (un an). Pour palier à ce désavantage, différents laboratoires se sont
intéressés depuis quelques années au développement de Xenopus tropicalis, une espèce très
proche de X.laevis, plus fragile, mais diploïde et mature sexuellement en quatre mois (Hirsch
et al., 2002). De plus, le génome de cette espèce a été entièrement séquencé. L’émergence de
ce nouveau modèle devrait permettre de complémenter par des méthodes génétiques l’étude
de la fonction des gènes chez l’embryon d’amphibien.
1.2. Le développement embryonnaire du xénope
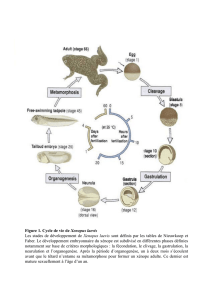
Le développement embryonnaire des vertébrés est subdivisé en différentes phases définies
notamment sur base de critères morphologiques : la fécondation, le clivage, la gastrulation, la
neurulation et l’organogenèse (Fig. 1). Chez le xénope, les différents stades de développement
sont désignés par l’usage des tables de Nieuwkoop et Faber (Nieuwkoop and Faber, 1967).
Après la fécondation, le zygote subit une série de divisions mitotiques sans croissance
cellulaire. Les cellules dérivant de ces divisions sont appelées blastomères. Ces clivages
continus donnent naissance à des blastomères de plus en plus petits. L’apparition d’une cavité
au sein de l’embryon, le blastocoele, définit le stade blastula (stades 8-9). Aux stades blastula,
les différents feuillets primordiaux : ectoderme, mésoderme et endoderme ainsi que la polarité
dorso-ventrale sont spécifiés par des déterminants maternels. Les cellules du pôle animal
forment l’ectoderme qui donnera naissance à l’épiderme et au système nerveux. Les cellules
situées dans la région équatoriale correspondent au mésoderme d’où dériveront les muscles, le
tissu conjonctif, le système urogénital et le système vasculaire. Les cellules du pôle végétal
donnent naissance à l’endoderme qui est à l’origine des systèmes respiratoire et digestif.
La gastrulation est la période durant laquelle les feuillets primordiaux se mettent en
place à leur position correcte en relation avec le plan du corps de l’animal. Cette étape du
développement implique de vastes mouvements et réarrangements cellulaires. Chez le
xénope, la gastrulation commence au stade 10. Les cellules du mésoderme et de l’endoderme

Introduction
3
s’invaginent au niveau du blastopore et s’étendent le long de l’axe antéro-postérieur sous
l’ectoderme par des mouvements de convergence et extension. En même temps, les cellules
de l’ectoderme recouvrent l’entièreté de l’embryon par un processus appelé épibolie. Une
nouvelle cavité remplace le blastocoele : l’archentéron qui correspond au tube digestif
primitif. Durant la gastrulation, le mésoderme dorsal forme la notochorde et les somites.
Lorsque la neurulation succède à la gastrulation au stade 13, le tube neural, précurseur
du système nerveux central embryonnaire, se forme. Les premiers signes du développement
neural sont l’épaississement de l’ectoderme dorsal qui formera la plaque neurale ainsi que
l’apparition des bourrelets neuraux aux bords de celle-ci. Ces bourrelets se soulèvent et se
rejoignent pour former le tube neural pendant que la notochorde et les somites continuent de
se développer selon l’axe antéro-postérieur. A la fin de la neurulation (stades 20-21), les
principales structures reconnaissables sont le tube neural, la notochorde, les somites, le
mésoderme latéral et le tube digestif.
Après la neurulation, la phase d’organogenèse commence. La partie antérieure du tube
neural se régionalise en trois compartiments : le prosencéphale, le mésencéphale et le
rhombencéphale tandis que la partie du tube neural au dessus de la notochorde forme la
moelle épinière. Ensuite, les vésicules optiques et otiques, les arcs branchiaux, le cœur, le rein
embryonnaire, le tube digestif et ses annexes ainsi que le système hematopoïétique primaire
apparaissent. Lorsque l’organogenèse est terminée, le têtard commence à nager et à se nourrir.
Deux mois après la fécondation, la queue régresse, les membres se forment et le têtard se
métamorphose en grenouille adulte.
2. Le développement de la crête neurale
2.1. La crête neurale : une structure spécifique des vertébrés
Les vertébrés sont distingués des autres deutérostomiens par un cerveau plus élaboré entouré
d’un squelette cranio-facial et d’organes sensoriels complexes tels que le nez, les yeux et les
oreilles. Beaucoup de ces innovations de la tête des vertébrés proviennent de seulement deux
tissus embryonnaires : la crête neurale (CN) et les placodes (Schlosser, 2006; Meulemans and
Bronner-Fraser, 2004). La CN a été décrite pour la première fois dans des embryons de poulet
par Wilhem His en 1868 comme « la corde entre » (Zwischenstrang), donnant naissance aux
ganglions spinaux et craniaux. La CN est une population de cellules mutlipotentes présente de
manière transitoire uniquement chez les embryons de vertébrés. Cette population de cellules

Introduction
4
naît à la jonction de l’ectoderme non-neural et de la plaque neurale, connue également sous le
nom de bourrelet neural. Durant la fermeture du tube neural, les cellules de la CN subissent
alors une transition épithélio-mésenchyme leur permettant de se délaminer du
neuroépithélium et de migrer dans l’embryon selon des chemins bien définis (Fig. 2). Les
cellules de la CN contribuent à l’élaboration de nombreux organes et de structures complexes
et se différencient en de nombreux types cellulaires tels que des mélanocytes, du cartilage, des
cellules musculaires lisses ou encore de nombreux types de neurones et cellules gliales du
système nerveux périphérique (Huang and Saint-Jeannet, 2004).
2.2. Mécanismes moléculaires impliqués dans le développement de la crête
neurale
2.2.1. La crête neurale : une structure d’origine ectodermique
Comme énoncé précédemment, la CN se forme à partir de l’ectoderme. Chez le xénope, les
trois feuillets embryonnaires, l’ectoderme, le mésoderme et l’endoderme, sont spécifiés au
stade blastula le long d’un axe animal-végétal. L’ectoderme est à l’origine de la plaque
neurale, de la CN, des placodes et de l’ectoderme non-neural qui donneront naissance
principalement au système nerveux central, périphérique et à l’épiderme. Des ARNm de
facteurs maternels sont stockés durant l’oogenèse et localisés au pôle animal ou au pôle
végétal. Après la fécondation, ces facteurs maternels orchestrent la mise en place des feuillets
embryonnaires. La polarité dorso-ventrale apparaît à la fécondation suite à un mouvement de
rotation du cytoplasme cortical vers le point d’entrée du sperme. Ce mouvement de rotation
induit une accumulation de la β-catenin, une protéine essentielle de la voie de signalisation
Wnt (Wingless Int-1), dans la partie dorsale de l’embryon (Heasman, 2006). La combinaison
de VegT (Zhang et al., 1998) et de β-catenin au niveau des cellules dorsales végétales donne
naissance au centre de Nieuwkoop. Lorsque la transcription des gènes zygotiques commence
après la transition mid-blastula, les cellules du centre de Nieuwkoop expriment les facteurs
Xnrs (Xenopus nodal related factors; Xnr1, 2, 4, 5, 6) de la famille des TGF-β (Transforming
Growth Factor β) (Agius et al., 2000). Les Xnrs, sécrétés selon un gradient dorso-ventral, vont
induire du mésoderme dans les cellules adjacentes au niveau de la zone équatoriale de la
blastula. Une forte activité des Xnrs va induire la formation d’un deuxième centre, appelé
organisateur de Spemann au niveau du futur mésoderme dorsal. Des facteurs maternels
localisés au niveau du pôle animal, Sox3 (Zhang et al., 2004) et Zic2 (Houston and Wylie,
2005), limitent l’activité des Xnrs dans cette zone. De même, Ectodermin, une ubiquitine

Introduction
5
ligase, contrôle la réponse des cellules aux TGF-β en régulant Smad4, un de ses médiateur
intracellulaire (Dupont et al., 2005), spécifiant ainsi l’ectoderme.
Après la spécification des trois feuillets embryonnaires, l’ectoderme va être régionalisé
d’abord en neuroectoderme et ectoderme non-neural. Les mécanismes moléculaires du
processus d’induction neurale sont relativement bien connus chez le xénope. Au stade
blastula, la β-catenin induit l’expression d’antagonistes de la voie de signalisation BMP (Bone
Morphogenetic Protein) comme Chordin et Noggin au niveau de la partie dorsale du pôle
animal, puis dans la région équatoriale dorsale (organisateur de Spemann). Ces inhibiteurs de
la voie BMP vont établir un gradient dorso-ventral d’activité des BMPs au niveau de
l’ectoderme. Cette partie dorsale du pôle animal exprimant les antagonistes des BMPs,
nommée BCNE (« Blastula Chordin Noggin Expression center »), contribuera à la formation
de la plus grande partie du tissu neural (la plaque neurale) et de la notochorde (De Robertis
and Kuroda, 2004). La partie ventrale de l’ectoderme, où le niveau d’activité des BMPs est
élevé, donnera le futur épiderme (Fig. 3).
2.2.2. Spécification de la crête neurale au sein de l’ectoderme
2.2.2.1. Les signaux extracellulaires
Ces dernières années, les bases moléculaires de l’induction de la CN ont été peu à peu
décryptées. Des expériences de dissections ont montré que la CN est induite au début de la
gastrulation chez le xénope (Mayor et al., 1995). De plus, des travaux chez le xénope et le
poulet ont suggéré que des interactions entre la plaque neurale et l’épiderme sont suffisantes
pour induire de la CN lorsque ces tissus sont juxtaposés (Seleck and Bronner-Fraser, 1995;
Mancilla and Mayor, 1996). Le rôle de plusieurs signaux extracellulaires, provenant des tissus
avoisinants, dans l’induction de la CN est bien démontré (Fig. 4). Il est aujourd’hui
communément admis que l’induction de la CN entre la plaque neurale et l’ectoderme non-
neural se déroule en deux étapes. Chez le xénope et le poisson zèbre, de nombreux travaux
indiquent qu’un gradient dorso-ventral de BMPs, lié au processus d’induction neurale, est
requis pour la formation de la CN au début de la gastrulation (LaBonne and Bronner-Fraser,
1998; Nguyen et al., 1998). Une dose intermédiaire de signaux BMPs établi une zone de
compétence en bordure de la plaque neurale, alors qu’une faible dose induit la formation de la
plaque neurale et une forte dose de l’épiderme. L’importance de ce gradient de BMPs était,
jusqu’il y a peu, cependant beaucoup moins claire chez le poulet. En effet, l’addition de
BMPs au niveau de la plaque neurale induit de la CN, suggérant qu’un haut niveau de BMPs
serait requis pour la formation de la CN (Liem et al., 1995). Enfin, un haut niveau
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
1
/
29
100%