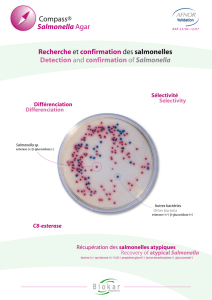
Salmonelloses

Manuel terrestre de l’OIE 2008 1387
CHAPITRE 2.9.9.
SALMONELLOSES*
RÉSUMÉ
La salmonellose est une maladie infectieuse de l'homme et de l'animal dont sont responsables les
organismes de deux espèces de Salmonella (Salmonella enterica et S. bongori). Bien que la
bactérie soit avant tout d'origine intestinale, les salmonelles diffusent largement dans
l'environnement et sont habituellement retrouvées dans les effluents d'élevage, les eaux usées
d’origine humaine et au niveau de matériels pouvant être sujet à une contamination fécale. Les
salmonelles sont l'agent étiologique d’infections diarrhéique et systémique chez l'homme, le plus
souvent en tant que contaminant secondaire d'aliment d’origine animale ou agricole ; en général la
maladie chez l’homme fait suite à une infection sub-clinique des animaux entraînant une
contamination de la viande, des œufs, et du lait ou encore une contamination secondaire des fruits
et des légumes qui ont été fertilisés ou irrigués avec des eaux usées. La salmonellose humaine est
l’une des plus fréquentes et importantes, pour ses conséquences économiques, maladies
zoonotiques. Les salmonelles peuvent aussi être trouvées dans l'alimentation animale, entraînant
un portage gastro-intestinal asymptomatique ou des maladies infectieuses chez l'animal,
particulièrement chez les volailles et les porcs. La salmonellose a été identifiée dans de nombreux
pays, mais semble être plus fréquente dans les zones d'élevage intensif, particulièrement de porcs
et de vaches reproductrices et dans certains élevages de volaille en confinement. Les reptiles sont
aussi habituellement porteurs asymptomatiques de salmonelles. Plusieurs sérovars sont
spécifiques de leur hôte (par ex. S. Abortusovis chez les moutons ou S. Typhi chez les humains) ou
adaptés à un hôte (par ex. S. Choleraesuis, S. Dublin).
La maladie peut atteindre toutes les espèces d'animaux domestiques ; les animaux jeunes et en
gestation ou lactation sont les plus sensibles. Les maladies entériques sont la manifestation la plus
fréquente, mais on peut observer une large variété de signes cliniques, tels qu’une septicémie
aiguë, un avortement, de l'arthrite et des signes respiratoires. Beaucoup d'animaux, en particulier,
le porc et la volaille peuvent être infectés mais sans montrer de signes cliniques. Ces animaux
jouent un rôle important dans la diffusion de l'infection entre les différents élevages et également en
tant que source de contamination des aliments et à l'origine de l'infection humaine.
La typhose et pullorose à S. pullorum, maladies des volailles causées par les salmonelles, sont
mentionnées dans le Chapitre 2.3.11. de ce Manuel terrestre.
Identification de l'agent pathogène : le diagnostic est basé sur l'isolement dans l'organisme à
partir soit de tissus prélevés aseptiquement lors de l’autopsie, soit de fèces ou d’échantillons
prélevés par écouvillonnage au niveau rectal, soit encore à partir de prélèvement réalisés sur le
terrain, de produits alimentaires ou destinés a l’alimentation animale ; une infection préalable ou
concomitante par certains sérovars peut aussi être diagnostiquée par la sérologie. En cas
d'infection des organes génitaux, lors d’avortement ou au cours de la mise bas, il est nécessaire de
mettre en culture le contenu stomacal du fœtus, et des frottis placentaires et vaginaux. Dans le cas
de volaille, il convient de mettre en culture des œufs embryonnés.
Les salmonelles peuvent être isolées en utilisant des techniques variées, qui peuvent inclure un
pré-enrichissement pour revivifier les salmonelles moribondes, des milieux d'enrichissement
contenant des substances inhibitrices pour éliminer la flore compétitive, et des géloses pour
l’isolement sélectif afin de différencier les salmonelles des autres entérobactéries.
Divers tests biochimiques, sérologiques et moléculaires peuvent être appliqués sur une culture
pure pour obtenir une confirmation définitive de la souche isolée. Les salmonelles possèdent des
antigènes de type somatique (O), flagellaire (H) et de virulence (Vi), qui peuvent être mis en

Chapitre 2.9.9. — Salmonelloses
1388 Manuel terrestre de l’OIE 2008
évidence par des sérums de typage, et le sérovar peut être déterminé par référence à une formule
antigénique du schéma de Kauffmann-White. De nombreux laboratoires envoient les isolats à un
laboratoire de référence pour confirmer l'identité sérologique complète et réaliser la détermination
du lysotype et du génotype de la souche lorsque c'est possible.
Épreuves sérologiques : des épreuves sérologiques peuvent être réalisées sur un échantillon
statistiquement représentatif de la population, mais sont de valeur limitée si la vaccination est
pratiquée. Chez les volailles, le test sur sang total est utilisé dans les élevages pour un diagnostic
rapide de S. Pullorum/Gallinarum, ce test étant relativement fiable dans certaines circonstances. Au
laboratoire, le test d'agglutination en tube est la méthode de choix pour les échantillons de toutes
espèces animales à des fins de diagnostic et d'exportation. Des épreuves immuno-enzymatiques
sont disponibles pour certains sérovars et peuvent être utilisées pour le diagnostic sérologique et la
surveillance, en particulier pour les élevages de volaille et de porc. La vaccination peut
compromettre la valeur diagnostique des épreuves sérologiques.
Spécifications applicables aux vaccins et aux produits biologiques à usage diagnostique :
plusieurs vaccins à bactéries inactivés sont utilisés contre la salmonellose et des vaccins à
bactéries vivantes sont commercialement disponibles. Du fait de l'efficacité plutôt faible des vaccins
à bactéries inactivées, des adjuvants huileux ou contenant des alhydrogels sont utilisés pour
améliorer leurs propriété immunogènes. Les données de terrain sur l'efficacité sont souvent
manquantes, bien que les tests de laboratoire puissent fournir des informations utiles. Des tests
d'innocuité sont réalisés en animalerie et, dans le cas des vaccins à bactéries inactivées, des tests
de stérilité utilisant des milieux de culture d'enrichissement sont réalisés. D’autres précautions, tels
que l'impact environnemental et la stabilité, sont nécessaires pour la production de vaccins
préparés par manipulation génétique. L'exclusion compétitive peut être utilisée pour réduire
l'infection salmonellique chez les volailles et dans d'autres espèces animales.
A. INTRODUCTION
Selon la dernière nomenclature, qui reflète les avancées récentes en taxonomie (42), le genre Salmonella
comprend seulement deux espèces : S. enterica and S. bongori. Une troisième espèce potentielle,
S. subterranea, a aussi été proposée à la suite de l’isolement unique à partir d’une souche inhabituelle trouvée
dans l’environnement (24, 27, 50, 54). Salmonella enterica est divisée en 6 sous-espèces, qui se distinguent par
certains caractères biochimiques et certains d'entre eux correspondent aux anciens sous-genres. Ces sous-
espèces sont :
Sous-genre d’origine Nomenclature actuelle
• Sous-espèces I = Sous-espèces enterica
• Sous-espèces II = Sous-espèces salamae
• Sous-espèces IIIa = Sous-espèces arizonae
• Sous-espèces IIIb = Sous-espèces diarizonae
• Sous-espèces IV = Sous-espèces houtenae
• Sous-espèces VI = Sous-espèces indica
Pour le sérovar S. bongori, le symbole V a été retenu pour éviter toute confusion avec le nom de sérovar
S. enterica subsp. enterica. Les souches de Salmonella sont classées en sérovars sur la base de la diversité des
antigènes (O) des lipopolysaccharidiques (LPS) et des protéines flagellaires (H) en accord avec le schéma de
Kauffmann-White ; actuellement environ 2 500 sérovars sont reconnus (42). Ce nombre est en croissance
constante. Les sérovars les plus fréquemment impliqués dans les infections humaine et animale appartiennent à
la sous-espèce enterica. Les sérovars des autres sous-espèces sont plus aisément retrouvés chez les animaux à
sang froid et dans l'environnement, mais sont parfois associés à une maladie humaine. Certains sérovars des
sous-espèces arizonae and diarizonae ont été associés à des infections chez la dinde et le mouton et d’autres
pourraient infecter des reptiles et des amphibiens en captivité ou à l’état sauvage.
Des noms ont été retenus seulement pour les sérovars appartenant à la sous-espèce enterica. Ces noms ne
doivent plus être écrits en italique. La première lettre est en capitale. Il n'est pas nécessaire d'indiquer le nom de
sous-espèce en pratique clinique, mais seulement le nom de sérovar de la sous-espèce enterica, par exemple,
Typhimurium, London ou Montevideo sont des sérovars de la sous-espèce enterica. Le genre Salmonella suivi du
nom du sérotype peut être utilisé en pratique (par ex. Salmonella Typhimurium). La plupart des sérovars des
autres sous-espèces sont désignés par une formule antigénique, qui comprend le nom de la sous-espèce sous
forme d’un chiffre romain (par ex. Salmonella IV 48:g.z51).

Chapitre 2.9.9. — Salmonelloses
Manuel terrestre de l’OIE 2008 1389
Dans ce chapitre, les abréviations des nouvelles conventions sont respectées, par exemple S. Typhimurium
plutôt que la nomenclature complète S. enterica, subsp. enterica sérovar Typhimurium. Par ailleurs, des
changements surviennent régulièrement à chaque fois que de nouveaux arguments sur la parenté génétique sont
disponibles, par ex. S. Pullorum est maintenant classé comme S. Gallinarum biovar Pullorum (42).
Les salmonelloses sont des maladies infectieuses humaines ou animales dont deux espèces de Salmonella sont
responsables (Salmonella enterica et S. bongori). Bien que la bactérie soit avant tout d'origine intestinale, les
salmonelles diffusent largement dans l'environnement et sont habituellement retrouvées dans les effluents
d'élevage, les eaux usées d’origine humaine et au niveau de matériels pouvant être sujet à une contamination
fécale. La salmonellose a été identifiée dans de nombreux pays, mais semble être plus fréquente dans les zones
d'élevage intensives, particulièrement dans les élevages de volaille et de porc.
La maladie peut atteindre toutes les espèces d'animaux domestiques ; les animaux jeunes et les femelles en
gestation sont les plus sensibles. La maladie entérique présentant souvent une diarrhée profuse aqueuse ou
sanglante avec hyperthermie est la manifestation clinique la plus fréquente, mais il est possible d'observer une
large variété de signes cliniques tels qu’une septicémie aiguë, des avortements, des arthrites, une nécrose des
extrémités et des signes respiratoires. Les signes et les lésions ne sont pas pathognomoniques. Beaucoup
d'animaux, particulièrement les volailles et les porcs peuvent aussi être infectés sans présenter de signes
cliniques (65). Ces animaux jouent un rôle important dans la diffusion de l'infection entre les différents élevages et
également en tant que source de toxi-infection alimentaire. Ces dernières apparaîtront lorsque ces animaux
entreront dans la chaîne alimentaire conduisant à des produits alimentaires contaminés (64, 65).
L'évolution de l'infection, les signes cliniques, les examens post mortem et les profils épidémiologiques varient
selon le sérovar et l'espèce animale atteinte. Certains sérovars n'affectent que certains hôtes, par exemple
S. Gallinarum pour les volailles ou S. Choleraesuis chez le porc, bien que la plupart des sérovars puissent être
responsables d'infections chez une grande variété d'espèces animales (51). Beaucoup de sérovars (incluant ceux
adaptés à l'hôte comme S. Choleraesuis et S. Dublin) ont été à l'origine d’une maladie chez l'homme ; les
vétérinaires et les personnels en abattoir peuvent être infectés directement au cours de leur travail, de même que
le personnel de laboratoire.
La maladie est couramment référencée comme une salmonellose, bien que le terme paratyphoïde puisse être
employé, comme par exemple, la paratyphoïde porcine. En volaille, les termes de pullorose ou diarrhée blanche
bacillaire et de typhose sont souvent utilisés pour décrire l'infection causée par S. Pullorum et S. Gallinarum,
respectivement (51). La typhose et la pullorose sont traitées en détails dans le Chapitre 2.3.11. de ce Manuel
terrestre.
Lors d’enquêtes épidémiologiques approfondies, l'identification de la souche est nécessaire et de telles enquêtes
sont classiquement basées sur les méthodes biochimiques et sérologiques, la lysotypie de certains sérovars et
l'antibiogramme. L'analyse génotypique de l'agent pathogène par des techniques d'amplification en chaîne par
polymérase en temps réel et d’empreintes moléculaire de l'ADN a été utilisée avec de bons résultats ces
dernières années. L'analyse des profils plasmidiques est une méthode rapide et relativement facile pour typer les
souches, et a été utilisée à la fois en médecine humaine et vétérinaire pour étudier la diffusion des Salmonella.
Cette technique a ses limites car toutes les souches de Salmonella ne portent pas de plasmides, et les plasmides
peuvent être facilement acquis ou être de taille identique bien que génétiquement différents. La technique s’est
cependant révélée utile lors d’enquêtes sur des foyers en complément d’autres techniques. D’autres méthodes
génétiques, telles que l'électrophorèse en champ pulsé, l’analyse du polymorphisme de longueur de fragments
amplifiés (AFLP pour amplified fragment length polymorphism), l’étude du nombre variable des tandems répétés
(VNTR pour variable number tandem repeat), l’analyse du polymorphisme d’un nucléotide simple (SNP pour
single nucleotide polymorphism) et le ribotypage automatisé sont de plus en plus utilisées (4, 32, 53, 56). Le
génotypage est un domaine qui s'est rapidement étendu et de nombreuses nouvelles techniques ont été
développées ces dernières années. Il importe de savoir qu'une seule méthode ne fonctionne pas pour tous les
isolats et qu'il peut être nécessaire d'évaluer un certain nombre de techniques différentes pour trouver une
méthode ou une combinaison de méthodes satisfaisantes et capables de différencier les clones d'un sérovar ou
d’un lysotype donné (45, 55). Les méthodes moléculaires sont souvent plus discriminantes et plus rapides que les
techniques phénotypiques, comme par ex. le sérotypage et la lysotypie, et sont en train de les supplanter dans
les recherches épidémiologiques. Cependant, ces techniques moléculaires peuvent ne pas être disponibles dans
tous les laboratoires ; elles ne sont pas non plus nécessairement normalisées en vue de donner des résultats
reproductibles dans des laboratoires différents. Il est donc possible que les isolats doivent être envoyés dans un
Laboratoire de référence pratiquant ce type d’épreuve.
Des techniques génétiques comme les analyses par micro-puces ou PCR multiplex visant autant à l’identification
des sérotypes spécifiques qu’à apporter des informations complémentaires sur le contenu des gènes, sont en
cours de développement (18, 19, 44).

Chapitre 2.9.9. — Salmonelloses
1390 Manuel terrestre de l’OIE 2008
L'isolement et l'identification des salmonelles ne dépendent pas seulement de la qualité de l'échantillon, mais
aussi des milieux de culture et des caractéristiques de croissance du sérovar, en particulier pour ceux adaptés à
une espèce d'hôte. Une étude complète des infections à Salmonella chez l'animal domestique a été récemment
publiée (65).
Des mesures nationales ont été mises en place dans de nombreux pays de façon à contrôler les infections à
Salmonella chez l'animal afin de protéger le consommateur. La directive Zoonoses 2003/99/CEE de l'Union
Européenne recommande la surveillance des élevages de plus de 250 animaux et des couvoirs pour
S. Enteritidis et S. Typhimurium. Dans une réglementation ultérieure, S. Virchow, S. Infantis et S. Hadar seront
aussi soumis à des contrôles spéciaux (10). Les cultures à partir des litières des boîtes de transport des poulets
et des poussins morts ou réformés sont réalisées le jour de l'arrivée. Après 4 semaines et 2 semaines avant la
ponte, un mélange de fèces (jusqu'à 60 échantillons selon la taille de l'élevage) est mis en culture. Par la suite,
les animaux adultes sont prélevés toutes les 2 semaines. Au niveau des couvoirs, le meconium ou les embryons
morts sont mis en culture toutes les 2 semaines, bien qu’il soit question de remplacer cette pratique par un suivi
dans les fermes ou au niveau des litières des boîtes de transport. Une nouvelle réglementation de l’UE pour le
contrôle des salmonelles dans les élevages de poules pondeuses, de poulets de chair, de dindes et de porcs est
actuellement mise en œuvre. Le suivi sérologique est autorisé mais comme une mesure complémentaire et ne
peut en aucun cas remplacer le suivi bactériologique dans les élevages de volaille. Au Danemark, la surveillance
sérologique pour Salmonella est utilisée pour les élevages de porc et les élevages commerciaux de ponte.
Plusieurs autres pays ont mis en place des programmes de suivis sérologiques dans les élevages
d’engraissement des porcs qui utilisent comme échantillons du « jus de viande » ou des sérums récoltés à
l’abattoir.
Les infections à Salmonella des animaux de rente jouent un rôle important en santé publique et particulièrement
en ce qui concerne la sécurité des aliments, car les produits alimentaires sont considérés comme la principale
source des infections humaines à Salmonella (64). Des programmes spéciaux ont été mis en œuvre pour la
surveillance des volailles, des porcs et des bovins incluant la surveillance des animaux sains porteurs
asymptomatiques de la bactérie Salmonella. Les contaminations croisées au cours de la transformation dans le
secteur agro-alimentaire sont également surveillées du fait qu’il peut exister une contamination à partir des
manipulateurs sains (65).
La contamination de l’alimentation animale a été la cause la plus fréquente d’introduction de nouvelles souches
de Salmonella dans les filières de production ; les salmonelles se sont répandues alors grâce aux déplacements
des animaux ou par d’autres moyens. Le commerce national ou le commerce international du bétail ou d’autres
animaux peuvent se révéler la principale menace de diffusion. Les aliments pour animaux peuvent aussi contenir
de petites quantités de sérovars pathogènes provenant de « l’environnement », qui sont peu susceptibles
d’entraîner des maladies ou des cycles d’infection chez les animaux. Étant donné que la contamination des
aliments pour animaux peut être due à des sérovars impliqués en santé publique, les produits pour l‘alimentation
animale devraient être analysés pour la recherche de salmonelles (65). Comme les aliments pour animaux sont
stockés et mélangés avec des ingrédients d’origine variée, il est possible de trouver des salmonelles
« exotiques » dans ces aliments. Une fois installé dans une ferme, la transmission entre animaux et la
contamination de l’environnement prennent le dessus pour assurer la pérennité de l’infection et sa dissémination.
Les échantillons d’alimentation humaine ou animale analysés pour la recherche de Salmonella devraient être
vraiment représentatifs. Des étapes appropriées devraient être mises en œuvre pour prévenir toute contamination
survenant durant le transport ou le stockage (20, 21). En raison de la grande variété de produits alimentaires pour
l’homme et l’animal, il n’existe pas de méthode d’échantillonnage appropriée pour tous les produits. Cependant
différentes méthodes spécifiques au produit doivent être utilisées (11, 22).
L’Organisation mondiale de la santé (OMS) apporte des informations sur la mise en œuvre de mesures
appropriées pour la prévention et la maîtrise des maladies transmises par les aliments, incluant les infections à
Salmonella chez l’homme. Les œufs et les ovoproduits, la viande de volaille et la viande des autres animaux de
rente, ainsi que les produits carnés sont le plus fréquemment impliqués dans la transmission de l’infection. En
outre, des salades et des épices contaminés ont été impliqués dans de nombreux foyers. Salmonella Enteritidis
et S. Typhimurium sont les sérovars les plus largement identifiés dans de nombreux pays européens (bien que
les salmonelles soient rares dans les productions animales, certains pays de l’UE possèdent des programmes
stricts de contrôle), alors que S. Typhimurium est le sérovar dominant dans l’Amérique du Nord (64).
Une maîtrise réglementaire de Salmonella dans un but de santé publique devrait couvrir toutes les étapes de « la
fourche à la fourchette » ou de « l’étable à la table ». Cela inclut l’obligation de déclarer tous les foyers
épidémiques de la maladie (13), et de tester les aliments, les animaux et les aliments pour animaux (65). La
surveillance de l’alimentation animale comprend les composants de l’alimentation et les autres matières qui, sont
distribuées non transformées ainsi que l’échantillonnage durant la fabrication des produits destinés à
l’alimentation animale. Des enquêtes épidémiologiques à l’échelle mondiale devraient être faites pour surveiller la
transmission des Salmonella et encourager les contrôles réglementaires de Salmonella.

Chapitre 2.9.9. — Salmonelloses
Manuel terrestre de l’OIE 2008 1391
Les contrôles sanitaires et d’hygiène à l’abattoir sont essentiels et des précautions spéciales devraient être
appliquées quand l’abattage concerne des troupeaux infectés. Des mesures de décontamination devraient être
mises en place tout au long de l’opération. Les vaccins étant de plus en plus fréquemment utilisés dans la lutte
contre les salmonelles dans les élevages de volailles, il est important de pouvoir distinguer les souches
vaccinales des souches sauvages et de vérifier que les souches vaccinales ne se répandent pas au-delà des lots
d’animaux vaccinés (60).
Un autre élément essentiel dans la prophylaxie de la salmonellose humaine est l’éducation des détaillants et des
consommateurs, en particulier l’attention portée à la manipulation et au stockage des aliments, l’hygiène des
cuisines et une cuisson appropriée pour limiter le risque d’infection.
B. TECHNIQUES DE DIAGNOSTIC
1. Identification de l’agent pathogène
La fréquence d’échantillonnage et le type d’échantillons obtenus dépendront largement des objectifs des
programmes d’investigations (y compris les impératifs réglementaires), des données cliniques, du niveau de
détection demandé ou de la précision requise sur les estimations de la prévalence, du coût des prélèvements et
de la disponibilité des matériels, et enfin des équipements du laboratoire.
Des échantillons individuels sont collectés le plus aseptiquement possible pour des tests bactériologiques et, en
cas de maladie clinique ou lors de suivi de routine, les échantillons doivent être collectés avant que tout
traitement antibiotique ait été commencé. Les échantillons cliniques sont collectés préférablement durant la
phase aiguë de la maladie ou le plus tôt possible après le décès. Dans le cas d’élevage intensif de volaille ou
d’autres espèces d’oiseaux, des échantillons d’environnement prélevés sur le sol tels que des mélanges de
fèces, la litière, la poussière ou des prélèvements réalisés sur les bottes à l’aide d’un écouvillon (5, 25), peuvent
être le meilleur moyen efficace d’identifier les troupeaux infectés. Des précautions doivent être prises pour éviter
les contaminations croisées des échantillons au cours de la collecte puis durant leur transport et au laboratoire.
Les emballages doivent être gardés au froid et accompagnés d’informations adéquates. Pour les espèces
animales plus petites, il est préférable de transmettre un nombre représentatif de malades ou d’animaux
récemment décédés au laboratoire si c’est possible (63). Des sérovars adaptés à l’hôte sont en règle générale
plus difficiles à isoler à partir de fèces, et si ces derniers sont suspectés, des tissus infectés devront être mis en
culture dans la mesure où cela est possible.
Une attention particulière doit être portée à l’isolement de salmonelles à partir d’animaux présentant une infection
sub-clinique, du fait que ceux-ci peuvent excréter la bactérie de façon intermittente et en faible nombre.
L’augmentation de la taille de l’échantillon, du nombre d’échantillons représentant plus d’individus associée
parfois au poolage des échantillons et à un échantillonnage répété peuvent augmenter la sensibilité du
diagnostic. Dans de telles situations, les méthodes bactériologiques ou sérologiques devraient être utilisées pour
identifier un troupeau infecté plutôt que pour identifier un animal infecté individuellement.
• Culture
De nombreuses méthodes sont largement utilisées à travers le monde pour l’isolement de Salmonella (9, 14, 17,
29, 46, 63). Celles qui sont les plus communes sont décrites ci-dessous. Les techniques de culture et les milieux
qui sont les plus performants dans une situation donnée dépendent de plusieurs facteurs, y compris le type de
Salmonella, la source et le type de prélèvements, l’espèce animale dont proviennent les prélèvements,
l’expérience du microbiologiste et la présence de milieux d’enrichissement et de milieux sélectifs.
Tous les milieux de culture préparés doivent subir un contrôle de qualité et doivent permettre la croissance du
micro-organisme recherché à partir d’un inoculum faible. La culture systématique d’une souche de référence en
parallèle avec des échantillons de routine peut entraîner une contamination croisée des échantillons si les
techniques sont réalisées sans précaution ; il convient donc d’utiliser un sérovar rare ayant des caractéristiques
de croissance connues, semblables à celles des souches cibles considérées comme prioritaires.
L’usage croissant de programmes d’assurance qualité a conduit à l’emploi de plus en plus généralisé de
méthodes internationales normalisées telle que l’ISO 6579:2002 ; même si celle-ci n’a pas été validée pour les
échantillons de fèces et de l’environnement et qu’elle n’a été conçue que pour les aliments (pour l’homme et
l’animal). Au cours des dernières années, une méthode normalisée pour la détection de Salmonella à partir des
animaux de production a été mise au point et évaluée, et une méthode ISO est en cours d’adoption (35).
L’essentiel de la méthode normalisée consiste en un pré-enrichissement dans de l’eau peptonée, un
enrichissement sur le milieu semi-solide modifié de rappaport-Vassiliadis (MSRV) et un isolement sur du milieu
xylose-lysine-désoxycholate (XLD) et sur gélose supplémentaire au choix.
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
1
/
19
100%