BioGen19 6 dias Fichier - Moodle

BioGen 19 -2016 -DC Gillan -UMons 1
1. La membrane plasmique (Euc et Proc)
2. Le cytosquelette (Euc)
3. La paroi des bactéries
4. Les mitochondries et les chloroplastes (Euc)
5. Le réseau intracellulaire de membranes (Euc)
6. Les constituants extracellulaires (Euc)
7. La communication cellulaire (Euc)
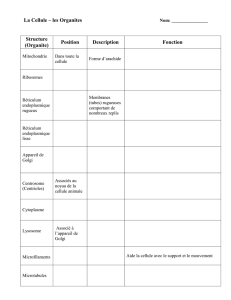
5. Le réseau intracellulaire de membranes (Eucaryotes)
-Enveloppe nucléaire (noyau)
-Réticulum endoplasmique (RE)
-Appareil de Golgi
-Lysosomes
-Peroxysomes
La cellule eucaryote est compartimentée
cytosol
Volumes relatifs des différents compartiments
Ex : hépatocyte
%"volume"
cellulaire"total
Nombre"par"
cellule
!"#$%$& 54 '
()#$*+$,-.)/ 22 '011
234 9'
2356764$&8) 9 '
:$";< 9 '
=/.$>)%$?/% '@11
5"%$%$?/% 'A11
3,-$%$?/% 'B11
Dépendance du type cellulaire
Hepatocyte Cellule"exocrine"
du"pancréas
(/?C.;,/6
D&;%?)E</
B F
234 35 60
235 16 G'
4$&8) 010
()#$*+$,-.)/6
H?/?C.I6/>#IJ
0 @
()#$*+$,-.)/
H?/?C.I6),#IJ
32 17
(/?C.I6),#I6,$";< 1KB 1K0
LM%)*<&/%6
%M*.M#)$,
,I-I A
5"%$%$?/% 1K@ ,I-I
=/.$>)%$?/% 1K@ ,I-I
3,-$%$?/% 1K@ ,I-I
Surfaces membranaires
Ribosomes
Cytosol
Mitochondries
Noyau
Peroxysomes
REG
Golgi
Lysosomes
Surface cellulaire
Vésicules de
sécrétion
rétention rétention
rétention
Branches du traffic protéique
Transport en fonction
dune séquence signal
Chloroplastes
RE(G) = Reticulum Endoplasmique (Granuleux)
Peu importe la destination du polypeptide, sa synthèse
débute toujours dans le cytoplasme.
Pfs, apparition dune séquence signal d
aa (extrémité N).
Exemple :
Polypeptides destinés au REG, ou à lextérieur de la cellule,
comportent une séquence signal de 20 aa.
La séquence signal est reconnue par un complexe protéique
appelé particule de reconnaissance du signal : SRP, Signal
Recognition Particle; contient des protéines et un pRNA).

BioGen 19 -2016 -DC Gillan -UMons 2
SRP
complexe de
translocation
séquence
signal
membrane du RE
lumière du RE
cytoplasme
mRNA
ribosome
Transport co-traductionnel
Le complexe de translocation comporte :
-un canal (passage du polypeptide en formation)
-une enzyme de clivage de la séquence signal
séquence signal
clivée
Transport co-traductionnel :
Limport des protéines vers la cible se fait en même tremps
que la traduction. Pour le RE.
Transport post-traductionnel :
Limport des protéines vers la cible se fait après la traduction.
Pour : chloroplastes, mitochondries, peroxysomes, noyau.
SRP = Signal recognition particle
ribonucléoprotéine :
6 polypeptides + 1 pRNA (7SL RNA : 300 nucléotides)
LARN 7SL
Le complexe de translocation comporte un récepteur
au SRP, un canal, BiP, et hydrolyse du GTP :
BiP : « binding protein » : reconnaît les protéines mal repliées
et aide au reploiement correct : "protéine chaperon".
Les protéines BiP sont retenues dans le RE grâce à une
séquence signal de 4 aa à lextrémité C-terminale.
Peptides signal
-15-60 résidus (aa)
-Souvent éliminés en cours de processus
-Souvent à lextrémité N-terminale
-Peuvent consister en un arrangement tridimentionnel de résidus
(après reploiement de la protéine)
Import dans REG H3N-Met-Met-Ser-Phe-Val -Ser
-Leu-Leu-Leu-Val-Gly-Ile-Leu
-Phe-Trp-Ala-Thr-Glu-Ala-Glu
Gln-Leu-Thr-Lys-Cys-Glu-Val-
bloc de résidus hydrophobes

BioGen 19 -2016 -DC Gillan -UMons 3
Rétention REG -Lys-Asp-Glu-Leu-COO–
Import vers mitochondrie H3N-Met-Leu-Ser-Leu-Arg-Gln
-Ser-Ile-Arg-Phe-Phe-Lys-Pro
-Ala-Thr-Arg-Thr-Leu-Cys-Ser
-Ser-Arg-Tyr-Leu-Leu
Vo i r d é t a i l s p l u s l o i n
Import vers le noyau -Pro-Pro-Lys-Lys-Lys-Arg-Lys-Va l -
Attachement à la membrane H3N-Gly-Ser-Ser-Lys-Ser-Lys-Pro-Lys-
(acide myristique
à lextrémité N-terminale : Gly)
Les propriétés physico-chimiques (charge, hydrophobicité,
espacement) sont plus importantes que la séquence exacte.
P K K K R KV
Compartiment cytosolique
Certaines modifications post-traductionnelles des protéines sont
effectuées dans le cytosol :
Exemples
-Fixation dun résidu N-acétylglucosamine à une sérine
-Phosphorylation
-Fixation dun co-enzyme (biotine, acide lipoïque, pyridoxal
phosphate)
-Fixation dun acide gras surface cytosolique membrane
Ces modifications servent à activer ou à adresser une protéine
Généralement pas de séquence signal
Co-enzyme :
La plupart des enzymes sont des hétéroenzymes et donc
constituées de deux éléments :
• Une apoenzyme, de nature protéique (codé par le génome)
• Un coenzyme, molécule organique de petite taille de nature
non protéique.
Coenzyme + apoenzyme = hétéroenzyme
-Les coenzymes favorisent l'activité de lheteroenzyme
-Bcp sont souvent indispensables (vitamines)
-Bcp sont apportés par lalimentation (pfs synthèse bactéries
du tube digestif).
Quelques exemples de co-enzymes :
• Porphyrine des cytochromes
• NAD+
• ATP, CTP, GTP, TTP, UTP
• Biotine (vit. B8)
• Pyridoxal phosphate
Fixation d
un acide gras
Les enzymes catalysant la réaction dans le cytosol détectent
une séquence signal
Un acide myristique (C14:0) est ajouté à une Glycine N-terminale
seulement dans un contexte particulier.
Un acide palmitique (C16:0) est àjouté à une Cystéine localisée à 4
résidus de lextrémité C-terminale (dans contexte particulier)
Beaucoup denzymes transmembranaires sont fixés par
de lacide palmitique au niveau de la face cytosolique

BioGen 19 -2016 -DC Gillan -UMons 4
Acide myristique : C14:0
Acide palmitique : C16:0
Ancres lipidiques
protéine attachée
à la membrane
S-CoA
C=O
H2N
HN
C=O
CoA-SH
Myristyl CoA
lien amide
Cytosol
protéine src
protéine attachée
à la membrane
S-CoA
C=O
S-Cys
C=O
CoA-SH
Palmitoyl CoA
lien thioester
Cytosol
protéine ras
HS-Cys COOH
COOH
protéine attachée
à la membrane
S-CoA
C=O
S-Cys
C=O
CoA-SH
Palmitoyl CoA
lien thioester
Cytosol
HS-Cys
La protéine attachée peut être transmembranaire :
Stabilité variable des protéines dans le cytosol
• Importance du premier acide aminé
Acides aminés stabilisants, à lextrémité N-terminale
Met, Ser, Thr, Ala, Val, Cys, Gly, Pro.
Les 12 autres sont déstabilisants (protéases)
• La dégradation sélective des protéines dans le cytosol
se fait par la voie de lubiquitine.
Ubiquitine = petite protéine de 76 aa.
Si plusieurs ubiquitines sont ajoutées à une protéine (au moins
4), celle-ci sera dégradée par le protéasome.
Les protéines dénaturées, mal repliées, ou comportant des
aa anormaux, sont des cibles pour lubiquitination.

BioGen 19 -2016 -DC Gillan -UMons 5
Le noyau et l
enveloppe nucléaire
-Diamètre moyen du noyau : 5 μm
-Deux bicouches lipidiques séparées de 10-50 nm
-Ribosomes sur face cytoplasmique
-Milliers de pores nucléaires, comportant un complexe protéique
Le complexe du pore nucléaire (100 nm ø total)
= Nuclear Pore Complexes (NPCs)
Composé de nucléoporines (50-100 protéines ≠ chez Euc.)
Diamètre effectif : 10 nm
Passage de molécules et macromolécules
-Lamina nucléaire : filaments intermédiaires (IF) à la face interne
du noyau (soutient mécanique de la forme du noyau).
-Matrice nucléaire comportant un réseau de fibres et la chromatine.
-Nucléole : région nucléaire ou les rRNA sassemblent
nucléole
La membrane externe est continue
avec le REG
REG
ribosomes
nucléole espace
périnucléaire
(continu avec REG)
Phosphorylation de la lamina pendant la mitose : désassemblage
Lamine
-Protéines de type Filaments intermédiaires (FI)
-Existence de Lamine A et Lamine B (Vertébrés)
-Forment un réseau 2D sous lenveloppe nucléaire
-Le noyau se désaggrège lorsquelles sont phosphorylées
(mitose, méiose)
• Lamine A : non attachée à la membrane nucléaire
(la pré-Lamine A est attachée par prénylation, puis est
détachée pour former la Lamine A)
• Lamine B : attachée à la membrane nucl. par prénylation
Prénylation : adjonction groupement farnesyl ou geranylgeranyl.
Farnesol :
15 carbones
Ajout sur cystéine :
Cystéine terminale méthylée
Cystéine terminale
méthylée
Enzyme : FTase (farnesyl transférase)
Importance de la lamina nucléaire (Lamine A) : progeria
Mutation ponctuelle position 1824 gène LMNA (C remplacé par T)
-Dominant
-Mutation
de novo
-Très rare
(1 / 4.106naiss.)
80 cas
répertoriés
dans le monde
La pré-lamine A mutée (progérine) reste attachée à la
membrane dans la progéria et déforme le noyau.
ancre
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
1
/
21
100%