Outils de Biologie Cellulaire : Techniques et Lexique
Telechargé par
massilia catseyes

QUELQUES OUTILS DE LA BIOLOGIE CELLULAIRE
1) Immunocytochimie
Après une fixation sur lamelles, la perméabilisation des cellules a lieu avec un détergent. On incube alors les cellules
avec un premier anticorps spécifique de la protéine étudiée, puis on révèle les sites où le premier anticorps s’était
fixé avec un second anticorps (dirigé contre les parties constantes des anticorps spécifiques) qui peut être couplé à un
fluorochrome. Le résultat est alors observé en microscopie à fluorescence.
Cette technique permet détecter et de localiser des protéines dans des cellules et tissus, in situ.
2) Immunoprécipitation (IP)
La présence d’une protéine donnée (A) dans un extrait cellulaire peut être mise en évidence à l’aide d’un anticorps
dirigé contre cette protéine (Anticorps-antiA) ; cette technique permet aussi de mettre en évidence les protéines
associées directement (B) ou indirectement (C) à la protéine A.
Il en résulte la formation d’un complexe Anticorps-AntiA/complexe A-B-C. Ce complexe après avoir été isolé (par
centrifugation) peut être analysé en électrophorèse. Pour ce faire, le complexe doit être dissocié en vue de libérer
toutes les protéines afin qu’elles puissent migrer dans un gel soumis à champ électrique (les complexes trop
volumineux ne rentrent pas dans les mailles du gel et l’analyse ne peut se faire).
3) Electrophorèse en SDS- PAGE (technique du Western)
a) Les protéines dénaturées se chargent négativement et uniformément par le SDS (Dodecyl sulfate de sodium) ; dans
ces conditions, elles migrent vers le pôle positif en fonction de leur masse moléculaire (MM). Pour estimer la taille
des protéines on fait migrer en parallèle des protéines dont la MM est connue.
b) Transfert des protéines sur une membrane de nitrocellulose qui lie fortement les protéines ; on récupère alors une
image fidèle du gel sur le filtre.
c) Repérage des protéines en traitant la membrane avec des anticorps spécifiques (immunoblot). Les anticorps se
fixent spécifiquement sur la protéine, c’est à dire au niveau de la bande ; la visualisation s’effectue avec l’utilisation
d’un deuxième anticorps dirigé contre les parties constantes des anticorps spécifiques. Ces anticorps ont été
préalablement marqués (par couplage à un enzyme par exemple). La réaction enzymatique se fait en présence du
substrat qui donnera un produit coloré ; le produit coloré sera trouvé dans la région où se trouve l’enzyme c’est à dire
au niveau de la protéine recherchée.
Cette technique est très sensible et permet la détection de fiables quantités de protéines. Elle peut être rendue
quantitative.
4) Northern Blot
Il permet de mesurer un taux d’ARNm (c’est à dire un équilibre entre synthèse et dégradation). Les ARNs sont
extraits. Ils sont séparés selon leur taille sur un gel d’agarose, transférés sur une membrane (support hydrophobe
solide). La membrane est ensuite hybridée avec la sonde spécifique de l’ARNm étudié (marquée au
32
P) et
autoradiographiée. On hybride, en même temps ou après, cette membrane avec une sonde de référence (par exemple
spécifique d’un ARN ribosomal) afin de tenir compte de l’efficacité des techniques d’extraction et de l’importance
de la dégradation des ARNs (molécules fragiles) au cours de ce processus.
5) Run on
Il s’agit de mesurer la synthèse d’ARNm, c’est à dire le taux d’initiation de la transcription. Les noyaux, c’est à dire
la machinerie transcriptionnelle, sont extraits des cellules puis incubés avec des ribonucléotides dont un
(généralement l’UTP) est radiomarqué (
32
P). On dépose sur une membrane des ADNc de référence et l’ADNc du
gène étudié. On hybride la membrane avec les ARN préparés à l’opération précédente. La membrane est
autoradiographiée. Un effecteur agit au niveau transcriptionnel si cette expérience montre une synthèse augmentée
c’est-à-dire une hybridation plus intense par rapport au contrôle.
LEXIQUE
Transcription : synthèse de RNA par une RNA polymérase à partir d’une matrice d’ADN
Trans (facteurs agissant en) : facteur diffusible capable de moduler l’activité (en + ou en -) s’un ou de plusieurs
gènes en interagissant avec leur(s) séquence(s) régulatrice(s).
Cis (regulation en) : action régulatrice des séquences DNA non codantes s’exerçant sur un ou plusieurs promoteurs
situés à proximité sur le même chromosome.

Promoteur : région d’ADN en amont des gènes comportant le site de fixation de la RNA polymérase ainsi que les
sites de fixation de protéines régulatrices de la transciption (facteurs trans)
Enhancer (séquence stimulatrice) : séquence d’ADN stimulant en cis la transcription de certains gènes eucarytotes
quelles que soient son orientation, sa localisation en amont ou en aval du site d’inititation, et, dans une certaine
limite, la distance qui le sépare du promoteur. Certains facteurs protéiques interagissant avec eux peuvent leur
conférer une spécificité tissulaire. Dans des constructions in vitro ils conservent leur pouvoir stimulateur sur des
promoteurs hétérologues.
Silencer : séquence d’ADN ayant, au contraire d’un enhancer, un effet cis-inhibiteur sur la transcription d’un gène
Consensus (séquence) : courte séquence nucléotidique, d’ADN ou d’ARN, impliqué dans un mécns
ADNc (cDNA) : séquence d’ADN complémentaire d’un ARNmessager obtenue par transcription inverse
(transcriptase inverse). Elle correspond à une version du gène débarassé de ses introns.
Vecteur : Séquence nucléotidique capable de s’autorépliquer, utilisé pour la recombinaison in vitro du DNA et son
alplification extra-chromosomique
Plasmides : fragments de DNA extra-chromosomique (épisomal) circulaire présent dans les bactéries, susceptibles
de se répliquer de façon autonome. Ils peuvent porter des gènes de résistances aux antibiotiques. Expérimentalement
ils sont utilisés comme vecteurs. Ils peuvent contenir des régions cis-régulatrices, un cDNA…
Transfection : technique expérimentale consistant à faire pénétrer un fragment de DNA (vecteur…) dans une cellule
eucaryote
Cotransfection : transfection simultanée de plusieurs séquences d’ADN dont un gène marqueur le plus souvent
sélectionnable (gène de résistance à la néomycine etc…)
Vecteur d’expression : insertion d’un ADNc d’intérêt en aval d’un promoteur fort (promoteur viral en général
inductible ou non). Après transfection, la séquence d’ADNc est transcrite puis traduite. On peut ainsi tester l’activité
de protéines sauvages ou mutées dans des modèles cellulaire.
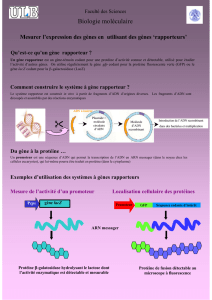
Gène rapporteur (encore appelé reporter) : Se dit d’un gène indicateur introduit dans des vecteurs d’expression
pour servir de témoin de l’effet régulateur d’une séquence donnée.
Technique du gène rapporteur : différents fragments de promoteur sont insérés dans un plasmide en amont d’un
gène rapporteur (le plus souvent CAT ou luciférase). Ce gène code une enzyme bactérienne qui n’est pas présente
dans les cellules eucaryotes. Les cellules eucaryotes sont transfectées par ces plasmides. Les transfections peuvent
être transitoires si le plasmide ne contient pas de gène de sélection ni de dispositif d’intégration dans le génome
eucaryotes (LTR viraux) ou permanentes si il contient un gène permettant d’effectuer la sélection des cellules
transfectées (1 % des cellules, résistance à la néomycine par exemple). Les procédés de transfection sont multiples.
La mesure de l’activité CAT ou luciférase permet d’évaluer l’activité de transactivation des éléments du promoteur
insérés en amont
Sonde : séquence d’acide nucléique, d’au moins 15 nucléotides, homologue à une séquence d’ADN ou d’ARN, avec
laquelle elle s’hybride de façon stable et spécifique par réassociation entre bases complémentaires.
Amorces (primer) : courte séquence d’ADN ou d’ARN complémentaire du début d’un matrice servant de point de
départ à son recopiage par une polymérase.
Retard sur gel : technique permettant d’objectiver la présence et la spécificité d’une interaction entre une protéine et
une séquence d’ADN.
1
/
2
100%