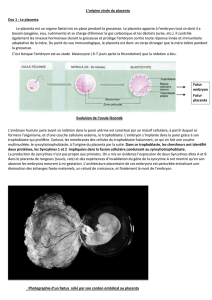
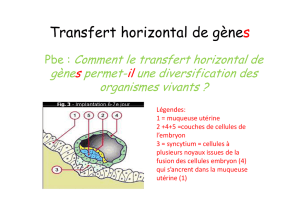
Approche populationnelle des interactions entre contrôle des

Stage M2
Approche populationnelle des interactions entre
contrôle des rétrovirus endogènes et résistance aux pathogènes.
Laboratoire d'accueil :
UMR CNRS 5558
Laboratoire de Biométrie Biologie Évolutive
43 bv 11 novembre 1918
69622 Villeurbanne Cedex
Équipe d'accueil :
Éléments transposables, Évolution, Populations
Direction : Cristina Vieira
Encadrant scientifique :
Marie Fablet
04 72 43 29 16
Les rétrovirus endogènes sont des séquences virales intégrées au génome de leur hôte, vestiges
d'infections passées. Leur intégration dans le génome engendre des mutations, qui sont le plus
souvent neutres ou délétères. Différents systèmes de régulation par le génome hôte contrôlent les
effets délétères de ces séquences, au niveau transcriptionnel ou post-transcriptionnel. En particulier,
les travaux de recherche des dernières années ont mis en évidence un mécanisme d'interférence
ARN impliquant de petits ARN appelés « piwi interacting RNAs » (piRNA), responsables de la
dégradation des transcrits des éléments transposables et des rétrovirus endogènes (Siomi et al.,
2011).
Nous savons depuis longtemps que la proportion du génome occupée par les rétrovirus endogènes
ainsi que l'activité de ces séquences sont très variables d'une espèce à l'autre, et même entre
populations d'une même espèce. Ainsi, le rétrovirus endogène tirant chez Drosophila simulans est
totalement inactif dans certaines populations, tandis qu'il est fortement transcrit dans certaines
lignées dépourvues des piRNA correspondants (Akkouche et al., 2012). Différents modèles sont
proposés pour expliquer cette variabilité, dont une variabilité des systèmes de régulation mis en
place par les génomes hôte. Nous avons en effet récemment montré que l'activité des gènes de
biogenèse des piRNA est très variable entre lignées de D. simulans (Fablet et al., 2014).
Ce mécanisme de régulation des rétrovirus endogènes par piRNA s'apparente à un processus
immunitaire, dont le champ d'action serait le génome. D'autres voies de régulation par ARN
interférence ont été décrites, comme la voie des siRNA, impliquée dans la défense contre les
infections virales. Des éléments de plus en plus nombreux suggèrent l'existence d'interactions entre
ces différentes voies d'ARN interférence. Ainsi, nous souhaitons tester l'hypothèse d'interactions
entre l'immunité au sens large et l'immunité génomique dirigée contre les rétrovirus endogènes.
L'étudiant/e aura à sa disposition différentes lignées naturelles de deux espèces de drosophile, pour
lesquelles nous avons mis en évidence une grande variabilité dans l'activité des rétrovirus
endogènes et dans la voie de biosynthèse des piRNA qui les régulent. Il/elle les soumettra à des
infections par différentes souches de levure du genre Candida, afin de caractériser la variabilité
naturelle dans la réponse à l'infection. Dans les différentes conditions, l'étudiant/e mesurera par RT-
qPCR l'activité de gènes impliqués dans l'immunité (au sens large et génomique) et des rétrovirus
endogènes, dans le but d'identifier des interactions entre les différentes voies de l'immunité.

Références
Akkouche et al., 2012. tirant, a newly discovered active endogenous retrovirus in Drosophila
simulans. J Virol. 86 : 3675-81.
Fablet et al., 2014. Variable expression levels detected in the Drosophila effectors of piRNA
biogenesis. Gene 537 : 149-53.
Siomi et al., 2011. PIWI-interacting small RNAs the vanguard of genome defence. Nat Rev Mol
Cell Biol. 12 : 246-58.
1
/
2
100%