master 2 - spécialité "biophysique"

Université PARIS 6 et Université PARIS 7
MASTER 2 - SPÉCIALITÉ
"BIOPHYSIQUE"
Proposition de Stage - Année 2012 – 2013
http://www.master.phys.upmc.fr/S_biophysique/
master.phys.biophys@upmc.fr
Sujet du stage (sous forme de titre court) :
La réplication d'ADN chez les eucaryotes
Laboratoire
Nom du Responsable : Marie-Claude MARSOLIER-KERGOAT
Affiliation administrative (CNRS, INSERM...) et n° l'Unité : CEA
Adresse précise du Laboratoire :
Laboratoire du métabolisme de l'ADN et réponses aux
génotoxiques
CEA/DSV/iBiTec-S/SBIGeM, bâtiment 144
91191
Équipe d'accueil des Doctorants
Nom de l'équipe: Mécanismes de Surveillance de l'ADN
Nom du Responsable : Marie-Claude Marsolier-Kergoat
École Doctorale de rattachement : n°426 : GENES, GENOMES, CELLULES
Responsable du Stage
Nom : Arach Goldar
Numéro de téléphone : 01 69 08 64 86
Numéro de télécopie :
Adresse électronique : Arach.g[email protected]
Profil de l'étudiant(e) souhaité : Physique et/ou biophysique
Renseignements complémentaires
Perspectives de poursuite de thèse :
X
oui
Non
Avec une bourse spécifique : oui
X
Non
si oui précisez :
Laboratoire d'accueil (Unité CNRS, INSERM, etc..) :
Nombre de chercheurs : 4
(dont 2 dans l'équipe d'accueil)
Nombre d'enseignants- chercheurs : 0
(dont 0
dans l'équipe d'accueil)
Nombre de "HDR" : 3
(dont 1 dans l'équipe d'accueil)
Nombre d'ITA : 3
(dont 0 dans l'équipe d'accueil)
Nombre de "post-docs" : 1
(dont 0 dans l'équipe d'accueil)
Nombre de visiteurs étrangers : 0
(dont
0
dans l'équipe d'accueil)

Sujet de stage (et principales techniques) :
L’ensemble des informations né
cessaires pour le bon fonctionnement d’une cellule
vivante est encodé sous forme d’un alphabet à 4 lettres dans un polymère appelé
acide désoxyribonucléique (ADN). L’ADN est confiné
dans un compartiment
particulier de la cellule soit par un processus de sé
paration de phase (chez les
procaryotes) soit par un barrière physique : le membrane nuclé
aire (chez les
eucaryotes). L’ADN génomique est associé à tout instant à des proté
ines. Ces
protéines sont en compétition pour interagir avec des séquences ré
gulatrices. Chez
les eucaryotes le complexe protéique le plus abondamment pré
sent le long de l’ADN
est appelé le nucléosome. Les nucléosomes confèrent à l’ADN u
ne structure
compacte tridimensionnelle appelée la chromatine. Pour pouvoir prolifé
rer les
cellules doivent se diviser de faç
on identique. Pour cela une copie de l’ADN de la
cellule mère est transmise à
la cellule fille. Ce processus de division (le cycle
cellulaire) chez les eucaryotes comprend 4 phases : 1) la préparation de l’ADN à
la
duplication (phase G1), 2) la réplication complè
te d’ADN dans un temps fini (phase
S), 3) le contrôle de l’ADN dupliqué et la préparation à
la division cellulaire (phase
G2) et 4) la division cellulaire (phase M).
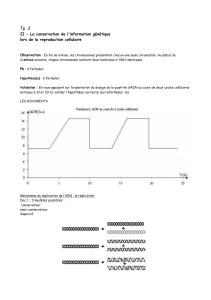
Nous é
tudions la phase S du cycle cellulaire chez les eucaryotes, nous utilisons
comme système modèle la levure de boulanger, Saccharomyces cerevisiae
. Ce
dernier possède 16 chromosomes. La réplication de l’ADN commence de faç
on
asynchrone à plusieurs loci chromosomiques appelés les « origines de réplication »
.
Le déclenchement d’une origine crée deux fourches de ré
plication qui progressent
dans des sens opposé
s avec une vitesse constante. Quand deux fourches de
réplication se rencontrent, elles fusionnent. La phase S est terminé
e une fois que
l’ADN est entièrement dupliqué. Les connaissances actuelles permettent de dé
crire
de façon détaillée les protéines intervenant durant la ré
plication de l’ADN.
Cependant, les mécanismes qui régulent la ré
plication de l’ADN sont mal
connus, par exemple : 1) comment l’ADN compacté
sous forme de chromatine peut
ê
tre dupliquer? 2) quels sont les facteurs qui influent sur le positionnement des
origines de réplication et leur temps de dé
clenchement? 3) comment la cellule
régule la ré
plication de son ADN en fonction de son environnement pour que le
temps de la phase S soit fini?
Nous proposons d’é
tudier les relations entre la structure de la chromatine et la
réplication. Des travaux récents ont permis d’établir la probabilité
spatiale de
présence des nucléosomes le long du génome de Saccharomyces cerevisiae
. Nous
utiliserons la technique de peignage moléculaire pour dé
tecter les origines de
réplication et analyser leur répartition spatia
le et temporelle. En, faisant une analogie
entre la réplication de l’ADN et un phénomène de nucléation et de croissance à
1
dimension, nous utiliserons des concepts issues de la physique statistique à
l’équilibre et hors équilibre pour explorer l'existe
nce d'une relation entre la
distribution spatio-temporelle des origines de ré
plication et la structure de la
chromatine obtenu à partir des probabilité de présence des nuclé
osomes. Nous
utiliserons des simulations numériques (Mont Carlo dynamique) pour vér
ifier la
validité des relations obtenues en reproduisant la répartition spatio-
temporelle
mesurée des origines de réplication.
Techniques mises en œuvre :
Culture cellulaire,
Peignage moléculaire,
Analyse du signal
Physique statistique
Simulation numérique
1
/
2
100%