BILAN D’ACTIVITE DES PLATEFORMES HOSPITALIERES DE GENETIQUE MOLECULAIRE Coordonnateur

Plateforme hos
p
italière de
g
énéti
q
ue moléculair
e
Coordonnateur
CHU de Bordeaux et Institut Bergonié Pr JP MERLIO
BILAN D’ACTIVITE DES PLATEFORMES HOSPITALIERES DE GENETIQUE MOLECULAIRE
DES CANCERS POUR L’ANNEE 2010
Merci de faire parvenir ces données avant le 11 mai 2011 aux coordonnées suivantes :
Frédérique Nowak

I- Données d’activité 2010 : le document cible l’activité ayant un impact sur la prise en charge médicale.
Niveau interrégional
Nombre de
patients avec
mutation ou autre
anomalie
Nombre de patients
sans mutation ou
autre anomalie
Motif le plus
fréquent ayant
conduit aux
résultats non
interprétables
CH Ets privés
Activité pour des
établissements
dépendant d'une
autre plateforme
leucémies (LAL/LAM/LMC) caryotype conventionnel 448 (dont 42 congelations non
étudiées
)
363 395 38 15 0
RT-PCR multiplex
q
ualitative 219 219 129 90 73 48 29 69
PCR simplex de
confirmation ou
suivi 131 130 130 0 0 20 18 15 78
LMC - suivi maladie résiduelle quantification BCR-ABL RT-PCR
q
uantitativ
e
1766 589 1223 524 1191 151 93 331
LMC/LAL mutations ABL (RT)-PCR +
sé
q
uen
ç
a
g
e 52 48 11 31 CDNA degrade 12 5 2 33
LMC - diagnostic et suivi anomalies de structure hors BCR-ABL FISH 11 1
RT-PCR multiplex
q
ualitative 37 37 4 33 33 2 2 0
PCR simplex de
confirmation ou
suivi 4440 0 2110
détection divers transcrits de fusion
MLL-AF4 RT-PCR qualitative 35 35 1 33 1 QI 33 0 2 0
E2A-PBX1 RT-PCR qualitative 35 35 1 33 1 QI 33 0 2 0
TEL-AML1 RT-PCR qualitative 28 28 6 22 0 22 0 6 0
SIL-TAL RT-PCR qualitative 3 3 1 2 0 3 0 0 0
PML-RARA RT-PCR qualitative 11 11 9 2 0 10 1 0 0
AML1-ETO RT-PCR qualitative 9 9 5 4 0 9 0 0 0
CBFbêta-MYH11 RT-PCR qualitative 1 1 1 0 0 1 0 0 0
NUP214-ABL1 RT-PCR qualitative 5 5
autres anomalies de nombre et structure FISH 157 154 138 15 4 0
mutations FLT3 PCR et analyse de
fra
g
ment 85 85 21 64 81 4 0
mutations NPM PCR , analyse de
fragment et
sé
q
uen
ç
a
g
e 67 67 26 41 63 4 0
mutation CEPBA séquençage 23 23 2 21 20 3 0
mutations autres gènes
quantification BCR-ABL RT-PCR
q
uantitativ
e
65 18 22 41 60 5 0
quantification WT1
quantification divers transcrits de fusion
quantification d'un allèle muté ou gène
hyperexprimé
anomalies de nombre et structure FISH 13 13 13 0 0 0
LAL - diagnostic clonalité B et/ou T
nb IGHDHJH =
nb IGHVHDHJH =
nb IGK =
nb IGL =
nb TCRB =
nb TCRD =
nombre de patients
pour qui au moins une
de ces analyses a été
réalisée:
LAL - suivi maladie résiduelle quantification IgH - TCR
caryotype conventionnel 86 86 52 19 15 0
anomalies de nombre et structure FISH 286 153 83 55 15
mutations somatiques IgVH
mutation p53
caryotype 11 11 911 0
clonalité B et/ou T sur moelle et/ou sang
n
b
IGHDHJH
=
nb IGHVHDHJH =
nb IGK =
nb IGL =
nb TCRB =
nb TCRD =
nb TCRG =
nombre de patients
pour qui au moins une
de ces analyses a été
réalisée:
LLC
0
2
LAL/LAM- suivi maladie résiduelle
Technique
utilisée1
Pathologie Test : Gène/marqueur
Onco-hématologie
0
LAL/LAM -diagnostic
10
0
0
Nombre de patients avec un résultat non
interprétable
0
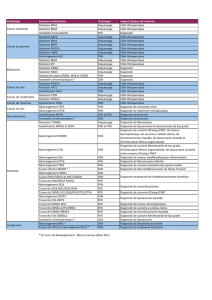
Résultats des tests (il n'est pas nécessaire de compléter les cases grisées)
Nombre de
prescriptions
établissements
de la plateforme
Nombre d’examens
effectués en 2010 Nombre de
patients
Nombre de prescriptions établissements hors
plateforme
Niveau régional
LMC - diagnostic détection BCR-ABL
19
détection BCR-ABL

clonalité B et/ou T sur tissus PCR acrylamide/
Protocole Biomed2
Clonalité B
PCR classique
nb IGH FR3/JH = 861
Biomed2
nb IGHVHDHJH =486
nb IGK =152
Clonalité T
PCR/DGGE
nb TCRG =1129
Biomed2
nb TCRB =11
nb TCRD =5
Clonalité B
PCR classique
nb IGH FR3/JH =
755
Biomed2
nb IGHVHDHJH
=173
nb IGK =152
Clonalité T
PCR/DGGE
nb TCRG =926
Biomed2
nb TCRB =11
nb TCRD =5
nb TCRG =3
Clonalité B
PCR classique
nb IGH FR3/JH = 208
Biomed2
nb IGHVHDHJH =65
nb IGK =46
Clonalité T
PCR/DGGE
nb TCRG =475
Biomed2
nb TCRB =3
nb TCRD =3
nb TCRG =2
Clonalité B
PCR classique
nb IGH FR3/JH = 470
Biomed2
nb IGHVHDHJH =74
nb IGK =82
Clonalité T
PCR/DGGE
nb TCRG =397
Biomed2
nb TCRB =6
nb TCRD =1
nb TCRG =1
Clonalité B
PCR classique
nb IGH FR3/JH
= 77
Biomed2
nb IGHVHDHJH
=34
nb IGK =24
Clonalité T
PCR/DGGE
nb TCRG =54
Biomed2
nb TCRB =2
nb TCRD =1
nb TCRG =0
Absence
d'amplification par
PCR = ADN dégradé
du à des problèmes
de fixateurs
Clonalité B
PCR classique
nb IGH FR3/JH =
463
Biomed2
nb IGHVHDHJH
=60
nb IGK =55
Clonalité T
PCR/DGGE
nb TCRG =578
Biomed2
nb TCRB =3
nb TCRD =2
nb TCRG =1
Cl
ona
li
t
é
B
PCR
classique
nb IGH
FR3/JH = 85
Biomed2
nb
IGHVHDHJH
=33
nb IGK =31
Clonalité T
PCR/DGGE
nb TCRG
=191
Biomed2
nb TCRB =4
nb TCRD =3
nb TCRG
=
1
Clonalité B
PCR classique
nb IGH FR3/JH
= 207
Biomed2
nb IGHVHDHJH
=79
nb IGK =66
Clonalité T
PCR/DGGE
nb TCRG =157
Biomed2
nb TCRB =4
nb TCRD =0
nb TCRG =1
nb IGHVHDHJH =1
mutations somatiques IgVH
anomalies de nombre et structure FISH 805 535 162 65 162 148
détection remaniements spécifiques (BCL1-
JH, BCL2-JH, autres…) FISH - PCR t(14;18) 133 110 62 14 40 6
détection cycline D1 RT-PCR
com
p
étitive 3302 210
quantification BCL1 ou2-JH pour suivi
mutation JAK2 V617F PCR quantitative
allèle s
p
écifi
q
ue 900 878 304 596 268
quantification JAK2V617F
autres mutations (MPL) HRM + Sequençage 10 1 9 10
mutations exon 12 JAK2 HRM 23 23 3 20 12 7 2 2
mutations exon 14 JAK2 HRM 23 23 3 20 12 7 2 2
TEL-PDGFRb PCR qual 9 10 2 8 19
caryotype conventionnel 36 34 23 5 8
anomalies de nombre et structure FISH 9 8 702
caryotype conventionnel 286 278 186 63 37
anomalies de nombre et structure FISH 29 29 20 7 2
mutations diverses
caryotype conventionnel
anomalies de nombre et structure FISH
chimérisme post greffe - mise au point
chimérisme post greffe - suivi
CGH array
mutations K-RAS
séquençage -
HRM puis
séquençage si
anormal
1191 1169 448 681
nb patients avec
un ADN non
amplifiable:
38
nb de patients
pour lesquels le
bloc est épuisé :
nb de patients avec
un % de cellules
tumorales en dessous
du seuil de sensibilité
du laboratoire et ne
permettant pas de
conclure en cas
d'absence de mutation
identifiée: 22
173 283 656 79
mutations BRAF
PCR AS -
HRM puis
séquençage si
anormal
701 693 96 576 102 160 421 18
mutations EGFR Séquençage direct 1292 1284 161 1058
nb patients avec
un ADN non
amplifiable au
moins sur les
exons 19 et 21:
58
nb de patients
pour lesquels le
bloc est épuisé :
nb de patients avec
un % de cellules
tumorales en dessous
du seuil de sensibilité
du laboratoire et ne
permettant pas de
conclure en cas
d'absence de mutation
identifiée: 118
problème de
fixateurs + faible
cellularité 471 240 567 9
mutations K-RAS
séquençage -
HRM puis
séquençage si
anormal
41 41 17 21 14 8 18 1
translocation EML4-ALK FISH 54 53 7 36 10 problème de
fixateurs + faible
cellularité 27 6 15 6
amplification HER2 FISH 578 549 74 350 256 76 206 40
HER2NEU SISH 25 24 1 23 24 1
cancer de l'estomac amplification HER2 FISH 26 26 4 17 4 4 17 1
gliome LOH 1p et 19q
méthylation de MGMT methyl-specific
PC
R
128 128 53 75 125 1 2
FISH 1
p
19
q
FISH 12 12 1 6 5
p
aucellularité 12 0 0
cancer du col utérin détection HPV de haut risque ( hors
indications ACSUS
)
neuroblastome amplification de Nmyc
test MSI 254 250 42 206 102 29 105 15
mutations BRAF PCR AS 38 38 6 31 8 6 21 3
cancer du sein
lymphome
cancer du poumon
SMP non LMC
tumeurs du spectre HNPCC
Leucémies
syndromes myélodysplasiques
cancer colorectal
Tumeurs solides
myélomes et autres syndromes
lymphoprolifératifs
5
1
3
21
1
106
0
ADN extrait de tissus
fixé de mauvaise
qualité = problème
de fixateurs
glioblastome
2

méthylation MLH1
mutations c-KIT 230 226 180 32 40 14 33 143
mutations PDGFRA 230 226 180 32 40 14 33 143
PCR 181 176 64 95 17 28 8 14 131
FISH 331 329 165 97 45 11 36 239
q-PCR 32 30 8 17 5 921 20
FISH 365 357 146 163 56 8 38 263
autres anomalies : mutations2 - Gène
CTNNB1 - tuemur desmoïd
e
séquençage 148 147 94 49 20 4 12 112
désé
q
uilibres
g
énomi
q
ues Chr 6 et Chr 1
1
mutation BRAF HRM puis
séquençage si
anormal 18 17 2 14 549
mutations cKIT HRM puis
séquençage si
anormal 27 26 1 24 1 932 13
FISH FISH 130 126 39 81 10 fixateur (Excell, AFA) 12 14 27 73
autres anomalies : am
p
lifications/délétions2
autres anomalies : translocations2
CGH array3 - Sarcomes 27 27 12 0 15 16 0 0 11
Pharmacogénétique
constitutionnelle mutations DPYD, UGT1A1, TPMT…
mutation BRAF
PCR AS -
HRM puis
séquençage si
anormal
33 29 11 14 11 4 13 4
rérran
g
ement RET/PT
C
RT-PC
R
14 12 2 8 2 228 2
rérran
g
ement RET/PT
C
FISH 33 111 0
mutation PIK3CA
BRAF
PCR AS -
HRM puis
séquençage si
anormal
8825221 1331
mutation PIK3C
A
mutation
p
53
p
53 : test fonctionnel en levure
mutation
p
53
p
53 : test fonctionnel en levure
mutation
p
53
p
53 : test fonctionnel en levure
ORL détection HPV de haut ris
q
u
e
corticosurrénalomes LOH 17
p
cancer de la vessi
e
mutation FGFR3
g
lioblastome recherche de mutation IDH1 et IDH2
Cancer du foi
e
recherche de mutation beta caténine
tumeur de la granulos
a
mutation Foxl2: intérêt diagnostiqu
e
séquençag
e
18 17 11 6 0 406 8
sarcome du stroma endometrial réarrangement de JazF1: intérêt diagnostique FISH 18 18 4 10 4 3 213
sarcome Amplification Cmyc FISH 2 2 1 1 2
mastocytose mutation cKIT séquençage 14 10 2 4 5 12 1 1
2 Préciser les localisations tumorales et le biomarqueur (créer autant de lignes que nécessaire).
3 Préciser les localisations tumorales.
4 Préciser l'intérêt de la détection de ce marqueur dans la prise en charge des patients.
Cancer colorectal
GIST
1 Si deux techniques distinctes sont utilisées au sein de la plateforme, merci de préciser le nombre de tests effectués par type de technique (créer autant de lignes que nécessaire).
A titre indicatif, autres tests en cours de validation4
cancer de la thyröide
sarcomes
cancer du sein
67
3
cancer du poumon
sarcomes
4
5
44
translocations diverses
14
14
Mélanomes
amplification MDM2/CDK4

Profil du poste (technicien,
ingénieur, secrétaire…) Nombre d’ETP (Equivalent Temps
Plein) Laboratoires où sont employés les personnes recrutées sur la dotation
allouée (nom du laboratoire, établissement, nom du responsable)
In
g
énieur 1 Service de Biolo
g
ie des Tumeurs - CHU de Bordeaux - Pr MERLIO JP
Technicien 1 Service de Biolo
g
ie des Tumeurs - CHU de Bordeaux - Pr MERLIO JP
Secrétaire 0,5 Service de Biolo
g
ie des Tumeurs - CHU de Bordeaux - Pr MERLIO JP
V
acation médicale 0,4 Service de Biolo
g
ie des Tumeurs - CHU de Bordeaux - Pr MERLIO JP
In
g
énieur 1 CDI Patholo
g
ie moléculaire - Institut Ber
g
onié - Dr SOUBEYRAN I
Technicien 0,5 CDI + 1 CDD (dotation KRAS) Patholo
g
ie moléculaire - Institut Ber
g
onié - Dr SOUBEYRAN I
II- Personnel recruté sur la dotation allouée
1
/
5
100%