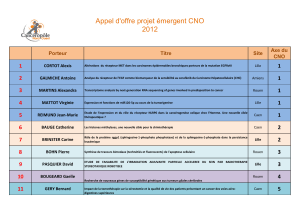
Analyse bioinformatique à visée diagnostique Bioinformatique, LBCO-CLCC François Baclesse, Plateforme SéSAME, Caen 1

Bioinformatique, LBCO-CLCC François Baclesse, Plateforme SéSAME, Caen 1
Analyse bioinformatique à visée
diagnostique

2
Objectifs
Séminaire d’Animation
de l’axe Cancéropole
Nord – Ouest
7 Décembre 2012
Rouen
Bioinformatique, LBCO-CLCC François Baclesse, Plateforme SéSAME, Caen
• Utilisation du séquençage Haut débit
– Diagnostic sur BRCA 1 et BRCA 2
• Un rendu des résultats plus rapide
• Rendu d'un Cas index : max 3 mois
– Recherche mutation ponctuelle et réarrangement de grande taille
• Etude sur les variants dans les autres gènes
• Objectifs du laboratoire
1 Run par mois
1 Run = 80 patients + 8 témoins
Nombre de patients traités annuellement : 960
Automatisation de l’analyse bioinformatique

3
Le set de capture CASOv3
Séminaire d’Animation
de l’axe Cancéropole
Nord – Ouest
7 Décembre 2012
Rouen
Bioinformatique, LBCO-CLCC François Baclesse, Plateforme SéSAME, Caen
CASO :
– Version 1 :
• 21 gènes
– Version 2 :
• 16 gènes
– Version 3 :
• 28 gènes
• Amélioration de la capture sur certaines zones
Gènes utilisés pour le rendu de diagnostique
– BRCA 1 : NM_007294.3
– BRCA 2 : NM_000059.3

Séminaire d’Animation
de l’axe Cancéropole
Nord – Ouest
7 Décembre 2012
Rouen
Etape 1 Etape 2 Etape 3
Mai Juin Sept Nov
Manuelle Semi-automatique Automatique

5
Étape 1
Séminaire d’Animation
de l’axe Cancéropole
Nord – Ouest
7 Décembre 2012
Rouen
Bioinformatique, LBCO-CLCC François Baclesse, Plateforme SéSAME, Caen
Fin du séquençage
o Présence du bioinformaticien
Début de l’analyse bioinformatique
Analyse
CASAVA : Démultiplexage + Alignement + Variant Calling
NextGENe : Alignement + Variant Calling
CNV-seq : réarrangement de grande taille
Création manuelle des rapports
CNV-seq
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
 46
46
 47
47
 48
48
 49
49
 50
50
 51
51
1
/
51
100%