D1-UE7-Doray-Diagnostic_moleculaire-27.03.17

Page 1 sur 16
UE7 – Génétique
Date : 27/03/16 Plage horaire : 10h45 – 12h45
Promo : DFGSM3 2016-2017 Enseignant : Dr. DORAY Bérénice
Ronéistes :
SCHOTT Zoé
HOARAU Christophe
Diagnostic moléculaire
I. Introduction
a. Champs d’application de la génétique moléculaire
b. Intérêts et conditions du diagnostic génétique
c. Extraction d’ADN
d. Diagnostic direct vs. indirect
II. Techniques diagnostiques classiques
a. La technique de PCR
b. Stratégie classique de détection des mutations uniques
c. Séquençage - technique PCR séquence Sanger
1. Principe et exemple
2. Avantages et limites de la méthode
d. Exemples de techniques alternatives au Sanger
1. Recherche non ciblée de variant
2. Recherche ciblée de variants
3. Recherche de grands réarrangements
4. Analyse quantitative –recherche d’anomalies de dosage
III. Approche pangénomique - le NGS (« next generation
sequencing »)
a. Principe et technique
b. Applications du NGS
---------------------------------------------------------------
2015 : précisions de la prof avant de commencer le cours, elle ne nous « embêtera pas » sur les différentes
techniques de biologie moléculaire. Le but étant de comprendre comment tout cela fonctionne et leur intérêt
en médecine.(sous entendu ce qui ne change pas en 2017!!!)

Page 2 sur 16
I. Introduction
• Les avancées révolutionnaires pour la génétique moléculaire :
1970 : endonucléases de restriction
1972-1973 : ligases
1975 : séquençage (Sanger et Coulon, Maxan et Gilbert, Hood)
1985 : PCR → fondamental dans l'étude de n'importe quelle maladie
1998 : puces à ADN ("microarrays"). D'abord utilisées en recherche (technique de CGH). Ne sont utilisées
en pratique courante que depuis 5-6ans.
=> on développe de plus en plus de technique de séquençage de haute définition (ex :NGS)
• Champ d'application de la génétique moléculaire en génétique humaine: (a quoi ça sert?)
→ établir le diagnostic d'une maladie héréditaire (elle donne un exemple d'un cas de drépanocytose. Un
enfant atteint de drépanocytose aura surtout un diagnostic clinic → observation simple de la forme de ses
GR + électrophorèse identifiant l'HbS. Mais dans le cas d'une grossesse à risque pour cette maladie, le
diagnostic ne pourra pas se faire sur les GR du fœtus. Il faudra donc ici utiliser la génétique moléculaire
pour vérifier si oui ou non le fœtus est atteint) (autre exemple/cas : pathologies pour lesquelles on a
BESOIN du test génétique pour confirmer le diagnostic)
→ établir le dépistage des hétérozygotes (porteurs sain de la mutation → problème pour hérédité); le
diagnostic prénatal; le diagnostic pré-implantatoire (dépistage même fondamental); le conseil génétique
aux couples et aux familles,…
• Le diagnostic génétique :
- permet de déterminer les individus porteurs d'une mutation
- permet d'identifier ceux qui ne la portent pas (diagnostic prédictif. Ex: patient dont le père est mort
maladie de Huntington et qui cherche à se rassurer à ce niveau)
- nécessite un consentement éclairé
- ne peut se faire que dans un laboratoire autorisé par un praticien ayant reçu un agrément
• L'extraction de l'ADN peut se faire à partir de différents tissus :
- Sang périphérique +++ (extraction de l'ADN des leucocytes)
- Salive ou Frottis buccaux (études de population, 1 ou 2 tests) → représentent au mieux l'état des cellules
du cerveau car ont pour origine les cellules ectodermiques (proche cerveau)
- Liquide amniotique et Biopsie de trophoblaste → diagnostic prénatal
- Cellule embryonnaire (stade 8) → diagnostic pré-implantatoire
- Cheveux, Sperme (médecine légale)
- Spécimens pathologiques archivés (individus décédés)
- Carte de Guthrie (buvard, goutte de sang talon nouveau né à J3) pour dépistage de Phénylcétonurie,
Hypothyroïdie congénitale, Hyperplasie surrénale, Mucoviscidose et Drépanocytose si population à risque

Page 3 sur 16
• Différents diagnostics
DIRECT
INDIRECT
La mutation est CONNUE
On cherche donc cette mutation chez l'individu
atteint. Ce qui nous permettra de confirmer le
diagnostic de façon très fiable.
Mutation non connue dans la famille
On cherche ici les liaisons génétiques avec la
mutation en cause.
C'est un suivi indirect du gène délétère à l'aide de
marqueurs polymorphes nécessitant l'analyse de
marqueurs génétiques (polymorphisme).
Cette recherche se fait sur plusieurs membres de la
famille et donne des résultats plutôt probabilistes
avec des risques de recombinaison.
Dans le cas de diagnostic indirect elle donne un exemple:
Un couple ayant eu un premier enfant diagnostiqué positif pour la Myopathie de Duchenne, cherche à
savoir si le deuxième enfant désiré le sera aussi. Mais sachant que le gène de Duchenne fait 80 exons, et que
ce n'est pas UNE mutation spécifique qui provoque la myopathie de Duchenne, la mutation responsable de
la maladie du premier enfant n'est pas connue/identifiée.
Le diagnostic indirect nous permettra de voir si le deuxième enfant aura hérité ou non du même
chromosome à risque.
Dans le cas de la myopathie de Duchenne, on sait que c'est sur le chromosome X et je sais aussi à quel
endroit la mutation doit se trouver pour provoquer cette maladie.
Donc au niveau de cette région là, on utilisera les marqueurs polymorphes au niveau du gène de
Duchenne. Le but n'est pas d'essayer de déterminer la mutation (parce que l'on ne la connait pas) mais de
savoir si la configuration génétique du futur enfant sera identique ou non du premier enfant atteint.
→ on observe alors des combinaisons d'allèles : haplotype
Complément de 2015 pr mieux assimiler:
Diagnostic direct : la mutation est connue a priori
• Recherche de cette mutation chez l’individu atteint, est-ce qu’elle est là ?
• Permet de confirmer le diagnostic.
• Fiable.
• C’est ce qui se fait le plus aujourd’hui.
Diagnostic indirect : la mutation n’est pas connue
• Souvent fait en parallèle du direct notamment dans le diagnostic pré-natal.
• Il y a plusieurs années, quand les recherches géniques commençaient à peine à se développer, les gènes en
cause dans des maladies étaient peu connus, c’était donc essentiellement du diagnostic indirect qui était
effectué.
• Le gène n’est pas connu, on ne connait pas précisément la mutations, la séquence d’introns et d’exons etc.
mais on a testé des individus atteints dans la famille et identifié quel chromosome est malade grâce à des
marqueurs micro-satellites : on regarde comment les micro-satellites se fixent sur le chromosome malade
(dans la famille, un chromosome qui fixe les micro-satellites d’une certaine manière est souvent associé à
la maladie). Si par exempl, dans un dépistage pré-natal, on voit qu’un chromosome fixe les micro-satellites
comme les individus malades dans la famille, on peut craindre qu’il possède la mutation. Ce n’est pas une
technique fiable à 100% car peu précise, et il y a le phénomène de crossing-over pendant la méiose qui
rend la technique encore plus probabiliste.

Page 4 sur 16
• Recherche de liaisons génétiques avec la mutation en cause.
• Suivi indirect du gène délétère à l’aide de marqueurs polymorphes.
• Combinaison d’allèles : haplotypes.
• Nécessite l’analyse de marqueurs génétiques (polymorphismes).
• Chez plusieurs membres de la famille.
• Résultat probabiliste, risque de recombinaison.
• A La Réunion on utilise cette technique de diagnostic indirect en complément du diagnostic direct pour
diagnostiquer le syndrome RAVINE.
• Comme on identifie la région du chromosome où est la mutation, la recherche précise de la mutation peut
se faire dans un second temps en recherche.
Exemple d’arbre généalogique avec manifestement une maladie autosomique dominante mais dont on ne connaît pas
la mutation. On a juste la certitude de la maladie et on sait que le gène est localisé entre m1 et m2 illustrés sur la diapo.
On ne peut pas séquencer tout l’ADN et tous les exons ni les introns, on donc fait le diagnostic indirect. On regarde
comment co-ségrège la maladie avec des marqueurs micro-satellites (connus et recensés dans les bases de données) et
on va pouvoir suivre l’allèle normale ou l’allèle muté. Ces marqueurs sont très nombreux (on n’en a représenté que 2
ici). Ici une très grande famille (idéal), on va tester la marqueur m1 et m2 et on voit que c’est toujours la combinaison
de marqueurs (ou haplotype) [ba] qui co-ségrègent avec la maladie. Donc l’enfant pour lequel on effectue cette
démarche de diagnostic, s’il a l’haplotype [ba] va surement être malade.
Même si l’on ne connaît pas la mutation on peut faire le diagnostic : ça marche très bien dans les grandes familles avec
des individus malades et d’autres sains. Ça marche moins bien quand les familles sont petites, qu’ils se connaissent
mal, que les personnes ne se savent pas encore malades, ou quand le gène est situé dans une région où il y a peu de
marqueurs micro-satellites proches, avec le risque de résultat altéré à cause du crossing-over dans les gamètes entre
ces marqueurs éloignés.

Page 5 sur 16
II. Les techniques diagnostiques classiques
a. La technique de PCR
La première étape est bien sûr de prélever l’ADN. Puis il va falloir l’amplifier pour pouvoir l’analyser.
La PCR est une technique fondamentale permettant l’amplification sélective de façon exponentielle d’une région
donnée grâce à choix de primers spécifiques. Le but étant d’avoir suffisamment d’ADN pour analyse ultérieure.
On mélange l’ADN, l’ADN polymérase et les primers dans des tubes et on les met dans la machine à PCR.
On va d’abord dénaturer l’ADN double brin à 95°C. Une fois les brins séparés par le jeu de complémentarité des bases,
on va avoir les primers en rouge (petits fragments d’ADN sélectionnés pour encadrer la région qu’on veut étudier,
exemple : exon 22 du gène myopathie de Duchenne; on prend les primers qui encadrent cet exon) qui vont s’hybrider
sur la région d’intérêt et cela se fait à 65-66°C.
A 70°C on va avoir l’élongation des primers et donc l’amplification de la région encadrée par ces primers. Ces 3 étapes
constituent 1 cycle et on va laisser la réaction se dérouler sur plusieurs cycles pour amplifier cette région d’intérêt de
l’ADN.
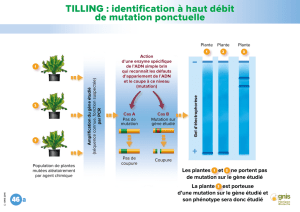
b.Stratégie classique de détection des mutations uniques
Exemple de la drépanocytose
• Avant on utilisait une technique « compliquée » :
o Substitution nucléotidique A>T abolissant un site de l’enzyme de restriction BsuI,
o Réalisation d’une PCR de la région contenant ce site de restriction,
o Incubation des produits de PCR avec l’enzyme BsuI,
o Migration sur gel d’électrophorèse.
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
1
/
16
100%