Télécharger le fichier - Fichier

. De l’œil au cerveau : quelques aspects
De la vision
thème 1
19
Thème 6 – ChapiTre 1 19
chapitre 1
UNITÉ
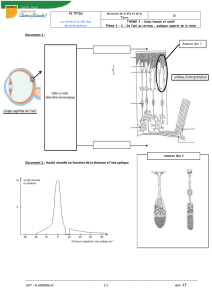
Le trajet de la lumière dans l’œil [pp. 292-293 du manuel de l’élève]
1
Connaissances du programme Capacités et attitudes mises en œuvre dans l’unité
• Le cristallin est l’un des systèmes transparents de l’œil humain.
Il est formé de cellules vivantes qui renouvellent en permanence leur
contenu. Les modalités de ce renouvellement sont indispensables
à sa transparence. Des anomalies de forme du cristallin expliquent
certains défauts de vision. Avec l’âge, sa transparence et sa souplesse
peuvent être altérées.
• Recenser, extraire et organiser des informations et/ou manipuler
(dissection, maquette et/ou recherche documentaire) pour :
– localiser et comprendre l’organisation et le fonctionnement du cristallin
(doc. 1 à 6) ;
– comprendre certains défauts de vision (doc. 4 et 6, exercices p. 304).
Exploitation des documents par les activités
➊ Doc. 1 et 2 (Manipuler, extraire des informations). La structure
dessinée en vert sur la modélisation du doc. 2 doit être transpa-
rente pour laisser passer la lumière et doit faire converger cette
lumière sur la rétine pour permettre la formation d’une image. Elle
doit donc posséder les caractéristiques d’une lentille convergente.
➋ Doc. 2 À 4 (Extraire et organiser des informations à partir de
différents documents). La dissection de l’œil (doc. 1) et le schéma
de coupe sagittale de l’œil (doc. 2) montrent qu’il existe dans
l’œil plusieurs milieux transparents traversés par la lumière : la
cornée, l’humeur aqueuse, le cristallin, l’humeur vitrée. Le cristallin
a une forme de lentille et, placé sur un texte, a l’effet d’une loupe
(doc. 3) : il se comporte donc comme une lentille convergente.
Une anomalie de forme du cristallin (doc. 4) entraîne un défaut
de vision. Le cristallin est donc très vraisemblablement la structure
représentée en vert sur la modélisation du doc. 2.
➌ Doc. 4 a 6 (S’informer à partir d’un tableau et de sa légende).
Plusieurs caractéristiques du cristallin contribuent à sa transparence :
– il est constitué de cellules particulières, vivantes mais dépourvues
de noyau et d’organites ;
– ces cellules sont remplies de cristallines, protéines solubles pré-
cisément arrangées.
La courbure du cristallin peut être modifiée grâce aux muscles
ciliaires, dont l’état de contraction modifie la convergence des
rayons lumineux qui le traversent : c’est l’accommodation, qui
permet de former une image nette sur la rétine lorsque l’objet
observé est proche. Le cristallin est déformable car il présente une
certaine élasticité, mais conserve sa structure grâce à l’étroite asso-
ciation des fibres cristalliniennes qui le constituent.
Au cours du vieillissement (doc. 4 et 6), le cristallin perd de son
élasticité, provoquant des difficultés d’accommodation et une
Conseils et suggestions
– La page d’ouverture du chapitre peut être utilisée pour aborder
la notion d’organe sensoriel et présenter d’œil comme un organe
sensoriel spécialisé dans la réception de stimuli lumineux.
– L’objectif de ce chapitre est de comprendre comment l’œil produit
un message nerveux visuel à partir d’un stimulus lumineux, en
envisageant des implications en matière de santé et en associant à
cette étude une approche évolutive permettant de placer l’Homme
parmi les primates.
– Lors de cette première unité, l’élève est amené à comprendre les
phénomènes optiques par lesquels une image est focalisée sur la
rétine ainsi qu’à découvrir le rôle et la structure du cristallin. Il est
à noter que celui-ci n’est pas la seule structure oculaire à dévier
les rayons lumineux : la cornée présente également un pouvoir de
réfraction élevé mais n’est pas abordée ici faute d’être mentionnée
dans le programme.
– La dissection proposée (doc. 1) a été réalisée sur un œil de veau.
Elle peut être mise en œuvre sur des yeux de lapin (à se procurer
auprès d’un boucher). Cette analyse peut être complétée avanta-
geusement par l’utilisation du logiciel L’œil et la vision de Pierre
Perez (voir l’atelier informatique p. 326), téléchargeable gratuite-
ment sur le site de l’Académie de Toulouse.
– De nombreuses informations complémentaires sur la vision et
le cerveau sont disponibles sur le site Internet Le cerveau à tous
les niveaux, de l’Université McGill de Montréal : http://lecerveau.
mcgill.ca
– La compréhension du trajet de la lumière dans l’œil s’appuie sur
des notions du programme de physique : optique géométrique,
fonctionnement optique de l’œil.
De la lumière au message nerveux :
le rôle de l’œil
. De l’œil au cerveau : quelques aspects
De la vision
thème 6
SVT 1reS © Éditions Belin 2011

Thème 6 – ChapiTre 1
20
baisse de la vision de près, constitutives de la presbytie. Le cristallin
peut également perdre sa transparence si l’arrangement des cristal-
lines dans les fibres cristalliniennes est modifié : c’est la cataracte,
caractérisée par une baisse de l’acuité visuelle.
➍ en conclusion (Communiquer en rédigeant une synthèse).
La lumière émise par un objet traverse les milieux transparents de
l’œil et converge sur la rétine grâce au cristallin. Celui-ci joue le rôle
d’une lentille convergente à focale variable : lorsque l’objet observé
est proche, l’accommodation modifie la courbure du cristallin et
permet de former une image nette sur la rétine. Lorsque l’objet est
éloigné, l’accommodation n’est pas nécessaire.
UNITÉ
La rétine et le rôle des photorécepteurs [pp. 294-295 du manuel de l’élève]
2
Connaissances du programme Capacités et attitudes mises en œuvre dans l’unité
« La rétine est une structure complexe qui comprend les récepteurs
sensoriels de la vision appelés photorécepteurs. Celle de l’Homme
contient les cônes permettant la vision des couleurs […] et les bâtonnets
sensibles à l’intensité lumineuse.
[…]
Le message nerveux issu de l’œil est acheminé au cerveau par le nerf
optique. »
• Extraire et exploiter des informations (maquette, logiciel et/ou recherche
documentaire et/ou observations microscopiques) pour :
– comprendre l’organisation de la rétine (doc. 1 et 3 à 6) ;
– déterminer le rôle des photorécepteurs (doc. 2 à 6, exercices 5 p. 305
et 7 p. 306).
nerveuses repérables par leur noyau et organisées en couches.
La lumière traverse ces couches pour parvenir à celle constituée
de cellules photoréceptrices appelées cônes et bâtonnets. Les
différents types de cellules nerveuses rétiniennes sont en contact
par des prolongements cellulaires. Les cellules ganglionnaires, qui
constituent la première couche de cellules ont des prolongements
cellulaires qui gagnent le nerf optique. Cette organisation de la
rétine suggère que la lumière reçue par les cônes et les bâtonnets
serait à l’origine d’un message transmis par les différents types
cellulaires au nerf optique.
Doc. 2 La réalisation d’un électrorétinogramme met en évidence
une activité électrique de la rétine en réponse à une stimulation
lumineuse. La rétine produit donc un message nerveux visuel
lorsqu’elle reçoit de la lumière. Dans le même temps, des élec-
trodes placées sur le cuir chevelu enregistrent une activité élec-
trique, signifiant que le message produit par la rétine est transmis
au cerveau. La dissection de l’œil a permis d’observer le nerf
optique qui met en relation anatomiquement l’œil et le cerveau :
il conduit donc vraisemblablement le message nerveux visuel au
cerveau.
Doc. 3 Les images obtenues grâce au microscope électronique à
balayage mettent en évidence les différences d’organisation des
cônes et des bâtonnets : leur nom respectif est dû à la forme de leur
segment externe. Ces cellules contiennent des pigments, protéines
capables d’absorber les rayonnements lumineux, et génèrent un
signal électrique lorsqu’elles sont stimulées par la lumière. Les
cônes et bâtonnets se distinguent également par leur sensibilité :
un seul photon suffit à déclencher une réponse des bâtonnets alors
qu’il en faut une centaine pour un cône.
Doc. 4 et 5 Le graphique met en relation la densité des cônes et
bâtonnets selon leur localisation par rapport à l’axe optique de l’œil
Conseils et suggestions
– Cette unité vise à montrer que la rétine, structure complexe,
contient des photorécepteurs qui convertissent un stimulus lumi-
neux en un message nerveux (doc. 1 à 3). Les caractéristiques dis-
tinguant les deux grands types de photorécepteurs sont dégagées
(doc. 3 à 6).
– Cette unité s’appuie sur des observations microscopiques, qui
pourront être confrontées aux images de microscopie électronique
pour mieux comprendre l’organisation de la rétine et des photo-
récepteurs. Des résultats cliniques (électrorétinogrammes), des
données de publications scientifiques (doc. 4 et 5) et une inter-
view d’un chercheur complètent l’approche pour en comprendre
les aspects fonctionnels. La diversité des ressources utilisées est
l’occasion de proposer de résoudre le problème posé sous la
forme d’une tâche complexe : il s’agit pour l’élève de mobiliser
des connaissances, de mettre en œuvre des capacités et attitudes
afin de répondre à une problématique globale. L’élève construit sa
propre démarche, en utilisant les ressources dont il dispose. S’il le
souhaite, il peut s’aider des conseils proposés qui constituent une
aide procédurale.
– L’atelier Sciences actualité (p. 326) constitue un travail com-
plémentaire à cette unité pouvant être associé à une recherche
documentaire (B2i).
Exploitation des documents par les activités
(Réaliser une observation microscopique, extraire des informations
à partir d’observations et de documents, organiser ces informa-
tions, communiquer par écrit)
Doc. 1 La rétine, fine « membrane » qui tapisse le fond de l’œil, est
une structure complexe constituée de plusieurs types de cellules
SVT 1reS © Éditions Belin 2011

(où convergent les rayons lumineux lorsqu’on fixe l’objet observé).
La répartition des photorécepteurs n’est pas homogène. Ils sont
absents de la zone de départ du nerf optique. Les cônes sont très
nombreux à proximité de l’axe optique de l’œil, dans une zone
appelée fovéa, où il y a peu de bâtonnets. En revanche, à mesure
que l’on s’éloigne de la fovéa, la densité des cônes diminue bruta-
lement tandis que celle des bâtonnets augmente (doc. 4). Ces don-
nées sont corroborées par des observations de la surface rétinienne
au MEB (doc. 5). Les cônes prédominent donc largement au niveau
de la fovéa alors que les bâtonnets représentent l’essentiel des
photorécepteurs dans la rétine périphérique. On constate donc que
les photorécepteurs les moins sensibles – les cônes – sont concen-
trés dans la zone où convergent les rayons lumineux. Cela suggère
une relation entre répartition et fonction des photorécepteurs.
Doc. 6 La vision centrale, qui met en jeu la fovéa, permet une
meilleure acuité visuelle ainsi qu’une meilleure vision des couleurs,
mais est moins efficace la nuit : elle nécessite une luminosité plus
importante. On peut en déduire que les cônes, peu sensibles et
localisés dans la fovéa, sont mis en jeu dans la vision des cou-
leurs, lorsque la luminosité est suffisante (vision photopique). En
revanche, la vision périphérique permet de mieux détecter un objet
peu lumineux – une étoile par exemple – la nuit : les bâtonnets, très
sensibles et essentiellement localisés dans la rétine périphérique,
sont mis en jeu lorsque la luminosité est faible (vision scotopique)
mais ne permettent pas de distinguer les couleurs.
en conclusion La rétine contient différents types de cellules : des
photorécepteurs qui produisent un message nerveux en réponse à
une simulation lumineuse, et des cellules nerveuses qui participent
à l’élaboration et à la transmission d’un message nerveux visuel qui
sera acheminé au cerveau par le nerf optique. Les photorécepteurs
sont de deux types : les bâtonnets et les cônes. Les bâtonnets, sur-
tout localisés dans la rétine périphérique, sont très sensibles et mis
en jeu dans des conditions de faible luminosité. Les cônes, présents
dans la fovéa, sont moins sensibles et interviennent si la luminosité
est suffisante. Ils permettent de voir les couleurs.
Thème 6 – ChapiTre 1 21
UNITÉ
Les pigments rétiniens et la vision des couleurs [pp. 296-297 du manuel de l’élève]
3
Connaissances du programme Capacités et attitudes mises en œuvre dans l’unité
« La rétine de l’Homme contient trois types de cônes respectivement
sensibles au bleu, au vert et au rouge.
Des anomalies des pigments rétiniens se traduisent par des perturbations
de la vision des couleurs. »
• Extraire et exploiter des informations (maquette, logiciel et/ou recherche
documentaire et/ou observations microscopiques) pour :
– comprendre l’origine de troubles de la vision colorée (doc. 1 à 5
et doc. 8, exercice 6 p. 305) ;
– comprendre le rôle des cônes dans la vision des couleurs (doc. 4 à 7).
– Pour aller plus loin dans la compréhension du daltonisme, il
est possible de comparer la proportion d’hommes et de femmes
concernés (doc. 3) et de faire la relation avec la localisation chro-
mosomique des gènes codant pour les opsines (doc. 8).
– La compréhension de la vision des couleurs s’appuie sur des notions
abordées dans le cadre du programme de physique : lumière, couleur.
Exploitation des documents par les activités
➊ Doc. 1 À 5 (Extraire et mettre en relation des informations
pour formuler une hypothèse explicative). La simulation de la
vision d’un individu daltonien (doc. 1) met en évidence l’impos-
sibilité de distinguer le rouge du vert. Le daltonisme (doc. 3) cor-
respond à plusieurs formes d’anomalies de vision des couleurs. Le
plus souvent, il s’agit d’une difficulté à distinguer le rouge du vert.
Le spectre de la lumière visible (doc. 2) correspond à un ensemble
de radiations lumineuses de couleurs différentes selon leur lon-
gueur d’onde, dans un intervalle compris entre 400 et 700 nm envi-
ron. Un individu qui voit normalement les couleurs peut distinguer
l’ensemble de ce spectre.
Conseils et suggestions
– Dans l’unité précédente, l’existence et le rôle des deux types
de photorécepteurs ont été montrés. Il s’agit ici de comprendre
comment les cônes peuvent permettre de distinguer les couleurs,
en utilisant comme point de départ une anomalie de la vision des
couleurs : le daltonisme.
– Le Dossier Pour La Science sur le thème de la couleur (dossier
hors-série, avril 2000) rassemble de nombreuses informations sur
la vision des couleurs et ces troubles.
– Le daltonisme est l’une des pistes évoquée par le programme :
l’exercice 6 (p. 305) permet d’étudier sa transmission dans une
famille. De nombreuses données sur ce sujet sont disponibles
sur Internet : le site www.daltoniens.fr ou celui du Syndicat des
Ophtalmologistes de France www.snof.org/vue/couleurs1.html,
par exemple.
– Le logiciel en ligne de traitement d’images Vischeck (en anglais)
simule la vision correspondant aux différents types de daltonisme
(www.vischeck.com). Il permet d’obtenir des images telles que
celle présentée dans le doc. 1.
SVT 1reS © Éditions Belin 2011

Thème 6 – ChapiTre 1
22
Toute couleur peut être obtenue en mélangeant trois couleurs dites
primaires (doc. 4). On peut formuler l’hypothèse que la perception
des couleurs pourrait reposer sur un système de combinaison de
trois « canaux » plutôt que sur de très nombreuses catégories de
photorécepteurs sensibles chacune à une longueur d’onde donnée.
Lorsqu’un daltonien croit reproduire une lumière jaune orangé en
utilisant seulement la lumière verte, ou seulement la lumière rouge
(doc. 5), on peut penser qu’il lui manque un de ces « canaux ».
L’hypothèse d’une vision des couleurs reposant sur trois « canaux »
– donc sur trois types de cônes – de sensibilité différente selon la
longueur d’onde se trouve confortée.
➋ Doc. 6 et 7 (S’informer à partir d’un graphique et d’un
schéma). Les cônes présentent une absorption différente selon
la longueur d’onde du rayonnement reçu. Cette absorption est
maximale pour une longueur d’onde donnée et décroît à mesure
que l’on s’éloigne de cette longueur d’onde : c’est le spectre d’ab-
sorption. On peut distinguer trois types de cônes selon leur spectre
d’absorption : certains présentent une absorption maximale dans le
bleu (430 nm), d’autres dans le vert (530 nm), les derniers dans
le rouge (560 nm). Leur spectre d’absorption diffère car ils contien-
nent des pigments différents : opsine S, opsine M ou opsine L. La
comparaison des séquences peptidiques de ces opsines (doc. 7)
révèle des différences, surtout entre les opsines M et S. On peut
supposer que ces différences dans la structure moléculaire des
opsines expliquent les différences de leur spectre d’absorption.
L’hypothèse de trois « canaux » mis en jeu dans la vision des cou-
leurs est confortée par l’existence de ces trois types de cônes dont
les spectres d’absorption sont variables. La combinaison de l’activité
des trois types de cônes permet la perception de l’ensemble des
couleurs du spectre visible.
➌ Doc. 8 (Mettre en relation des informations et raisonner).
Les opsines sont des protéines, codées donc par des gènes. Le gène
codant l’opsine S est localisé sur le chromosome 7, ceux codant les
opsines M et L sur le chromosome X. Dans l’exemple présenté, l’in-
dividu daltonien ne possède pas de gène codant pour l’opsine M.
On peut en déduire que les couleurs qu’il percevra dépendront
seulement de l’activité des cônes S et L. Cela peut expliquer des dif-
ficultés à faire la différence, par exemple, entre des rayonnements
lumineux de 540 nm (vert) et de 590 nm (rouge) : dans les deux
cas, seuls les cônes L absorbent ces rayonnements, avec le même
taux d’absorption.
➍ en conclusion (Communiquer en rédigeant une synthèse).
L’Homme perçoit les couleurs grâce à trois types de cônes sensibles
au bleu, au vert et au rouge car ils contiennent respectivement une
opsine S, M ou L. On dit que l’Homme a une vision trichromate car
c’est la combinaison de l’activité de ces trois types de cônes qui
lui permet de distinguer l’ensemble des couleurs du spectre de la
lumière visible.
UNITÉ
La vision des couleurs chez les primates [pp. 298-299 du manuel de l’élève]
4
Connaissances du programme Capacités et attitudes mises en œuvre dans l’unité
« Les gènes des pigments rétiniens constituent une famille multigénique
(issue de duplications) dont l’étude permet de placer l’Homme parmi
les primates. »
• Extraire et exploiter des informations (maquette, logiciel et/ou recherche
documentaire et/ou observations microscopiques) pour :
– s’informer sur la vision des couleurs chez les primates (doc. 1 et 2) ;
– faire le lien entre la vision des couleurs et l’évolution (doc. 3 à 5,
exercice 8 p. 306) ;
– Des informations issues de l’unité précédente peuvent être uti-
lisées pour conduire au questionnement concernant l’origine des
gènes codant les opsines : la comparaison entre opsines montre
des différences, très peu nombreuses entre opsine L et M. La
nature protéique de ces pigments est établie, et les gènes qui
les codent sont localisés (voir docs. 7 et 8, p. 297). Cela amène
naturellement à une comparaison des séquences nucléotidiques de
ces gènes, pouvant être mise en œuvre par l’utilisation du logiciel
Anagène (doc. 3).
– La comparaison des séquences peptidiques de l’opsine S de
différentes espèces de primates peut être également réalisée
par l’élève en utilisant le logiciel Phylogène : on proposera ainsi
un arbre de parenté à partir de la matrice des différences entre
séquences comparées deux à deux (doc. 5).
Conseils et suggestions
– Dans l’unité précédente, on a montré que la vision trichromate de
l’Homme dépend de l’existence de trois types de cônes, différents
par le pigment, de nature protéique, qu’ils contiennent. D’autre
part, l’appartenance de l’Homme aux primates est rappelée en
ouverture de cette unité. La démarche proposée ici vise d’abord à
s’interroger sur les mécanismes évolutifs ayant abouti à la vision
trichromate chez l’Homme puis à étudier les relations de parenté
entre primates sur la base de la vision des couleurs. Si des argu-
ments anatomiques ont déjà été utilisés pour l’établissement des
parentés, et notamment en seconde générale (voir le manuel de
SVT Seconde 2010, Thème 1, chapitre 6, pp. 77-88), les argu-
ments moléculaires sont abordés ici pour la première fois dans le
cursus.
SVT 1reS © Éditions Belin 2011

– L’utilisation des pigments rétiniens pour étudier les relations de
parenté chez les vertébrés est également abordée dans l’exercice 8
(p. 306), qui permet de réinvestir la démarche mise en œuvre ici.
– Pour aller plus loin, il est possible de construire d’autres matrices
de dissimilitudes à partir de séquences peptidiques de pigments
rétiniens pour diverses espèces : ces séquences sont disponibles
sur le site GenBank (en anglais) : www.ncbi.nlm.nih.gov/protein.
L’alignement des séquences peptidiques et leur comparaison peu-
vent être effectués en ligne avec le logiciel ClustalW2 (en anglais) :
www.ebi.ac.uk/Tools/msa/clustalw2. Ces deux sources proposent
également de travailler sur des séquences nucléotidiques.
Exploitation des documents par les activités
➊ Doc. 1 (Comparer des images et des graphiques pour extraire
des informations et raisonner). La simulation de la vision d’une
même image par un chimpanzé et un saïmiri montre que la vision
des couleurs du premier, trichromate, est comparable à celle de
l’Homme alors que le saïmiri semble incapable de distinguer le vert
du rouge. Le spectre d’absorption des différentes longueurs d’onde
de la lumière visible par les cônes rétiniens montre l’existence de
trois types de cônes chez le chimpanzé (dont les propriétés sont
comparables à ce qui est observé chez l’Homme, voir le doc. 6,
p. 297) et seulement deux chez le saïmiri. Ceci confirme la dichro-
matie de ce dernier, incapable de distinguer le vert du rouge.
➋ Doc. 2 (S’informer à partir d’un tableau, mettre en relation des
informations pour proposer une explication). Le chimpanzé et les
autres primates de l’Ancien monde possèdent, comme l’Homme,
un gène codant l’opsine S sur le chromosome 7 et deux gènes
codant les opsines L et M sur le chromosome X : cela explique
qu’ils disposent de trois types de cônes permettant une vision
trichromate, comme l’Homme. En revanche, le saïmiri et les autres
primates du Nouveau monde ne possèdent que deux gènes codant
des opsines : un gène sur le chromosome 7 code pour l’opsine S et
un gène sur le chromosome X code une opsine M/L (selon l’allèle
dont dispose l’individu, l’opsine fabriquée présente un maximum
d’absorption proche de celui de l’opsine M ou de l’opsine L de
l’Homme). En conséquence, le saïmiri ne possède que deux types
de cônes, ce qui explique sa vision dichromate et son incapacité à
distinguer certaines couleurs.
NB : Le cas des primates du Nouveau Monde a été simplifié pour
faciliter l’approche par les élèves : puisqu’il existe trois allèles dif-
férents pour le gène de l’opsine M/L, les femelles (qui possèdent
deux chromosomes X) peuvent être hétérozygotes pour ce gène :
elles disposent alors de trois types de cônes différents et sont dans
ce cas trichromates. L’article « L’évolution de la vision de la couleur
chez les primates » de G. Jacobs et J. Nathans (Pour La Science
n° 389, mars 2010, pp. 34-41) fait le point sur ce sujet.
➌ Doc. 3 et 4 (Extraire des informations). Les séquences nucléo-
tidiques des gènes des opsines L et M sont comparées à celle du
gène de l’opsine S. On constate qu’un certain nombre de nucléo-
tides sont identiques dans les trois séquences et qu’il existe peu de
différences (3 sur la centaine de nucléotides présentée) entre les
séquences des gènes de l’opsine M et de l’opsine L (doc. 3). Les
ressemblances entre séquences de gènes suggèrent une origine
commune : ces gènes différents dériveraient d’un même gène
ancestral, qui aurait été dupliqué, chaque copie accumulant de
manière indépendante des mutations expliquant les différences
constatées aujourd’hui (doc. 4). En raison de leur origine com-
mune, ces gènes forment une famille multigénique.
➍ Doc. 5 (Extraire et mettre en relation des informations).
Comme dit précédemment, les ressemblances entre les séquences
des gènes des opsines des primates traduisent l’origine commune
de ces gènes. La comparaison des séquences protéiques de l’op-
sine S, qui reflètent les séquences nucléotidiques des gènes qui les
codent, permet donc d’établir des parentés : puisque les mutations
s’accumulent au cours du temps, plus il y a de différences entre
deux espèces, plus leur dernier ancêtre commun ayant transmis le
gène codant l’opsine S est éloigné. À l’inverse, plus les séquences
sont semblables, plus le lien de parenté entre espèces est étroit. La
matrice des différences permet d’identifier deux groupes d’espèces
qui présentent moins de 14 acides aminés différents entre les
séquences d’une espèce à l’autre : cebus, alouate et saïmiri d’une
part, Homme, gorille, bonobo, chimpanzé et macaque d’autre part.
Deux espèces appartenant chacune à l’un de ces deux groupes pré-
sentent au moins 23 différences dans la séquence protéique étu-
diée : le lien de parenté est donc plus étroit au sein de chacun de
ces deux groupes, qu’entre espèces de groupes différents. L’analyse
de cette matrice permet alors de construire l’arbre de parenté pro-
posé, où l’on constate la séparation de ces deux groupes au cours
de l’évolution. D’après le doc. 2, les espèces du groupe cebus/
saïmiri/alouate (primates du Nouveau monde) possèdent un gène
codant pour une opsine M/L sur le chromosome X, les espèces du
groupe Homme/bonobo/chimpanzé/gorille/macaque (primates
de l’Ancien monde) possèdent deux gènes codant les opsines M
et L sur le chromosome X, traduisant une duplication génétique
chez ces derniers. Celle-ci s’est donc probablement produite après
la séparation de ces deux groupes.
➎ en conclusion (Communiquer en rédigeant une synthèse).
La comparaison des gènes d’opsine (nombre de gènes d’opsines,
séquences d’un même gène) montre que le lien de parenté est plus
étroit entre l’Homme et les autres primates de l’Ancien monde,
qu’entre l’Homme et les primates du Nouveau monde. Au sein de
ce groupe, la comparaison des séquences du gène de l’opsine S
montre en outre que l’Homme est plus étroitement apparenté au
bonobo et au chimpanzé qu’au gorille ou au macaque.
Thème 6 – ChapiTre 1 23
SVT 1reS © Éditions Belin 2011
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
1
/
13
100%