Je révise sur chap3 et chap4

Je révise sur le chap3 + chap4 – C.MARECAUX – 2
nde
THEME 1 –chap3 + chap4 : QUELQUES EXERCICES D’ENTRAINEMENT en SVT
POUR S’ENTRAINER A UTILISER SES CONNAISSANCES :
ENONCE 1 p.83 doc7
EBAUCHE DE
CORRECTION
1. On sait que la molécule d’ADN est constituée de 2 brins complémentaires de nucléotides. Le
doc 2 nous permet de le vérifier puisque quel que soit l’organisme étudié, les % de nucléotides
A et T sont très proches (27,2% ou 27,3% , pour l’exemple du blé) et les % de nucléotides C et
G aussi (22,8% et 22,7%, pour l’exemple du blé).
On vérifie donc bien que A est le nucléotide complémentaire de T dans la molécule d’ADN, et que C
est le complémentaire de G dans la molécule d’ADN.
Aussi , la séquence du 2d brin complémentaire à celui du doc1 est :
TCCAAGCGGATTCGAGCAT.
2.
% de nucléotides
Cellules de
A/T C/G (A+T)/(C+G)
Homme (30.9/29.4)= 1
(19.8/19.9)~1
(30.9+29.4)/(19.8+19.9)= 1.5
Levure de bière 1 1 1.2
Blé 1 1 1.2
Poule 1 1 1.3
On observe que quel que soit l’être vivant observé, le rapport A/T ou C/G est toujours égal à 1 .
On en déduit donc qu’il y a autant de A que de T, et autant de C que de G : A et T sont donc des paires
de nucléotides, et C et G sont une autre paire.
En revanche, le rapport (A+T)/(C+G) peut varier en fonction des êtres vivants (1.2, 1.3, 1.5), car c’est
justement la séquence de nucléotides qui caractérise un être vivant.
3. On observe que quel que soit l’organisme étudié, les % de nucléotides A et T sont similaires
(27,2% ou 27,3% , pour l’exemple du blé) et les % de nucléotides C et G aussi (22,8% et 22,7%,
pour l’exemple du blé).
On sait que la molécule d’ADN est constitué de paires de nucléotides, et ce tableau nous permet
de le vérifier : A est le nucléotide complémentaire de T dans la molécule d’ADN, et que C est le
complémentaire de G dans la molécule d’ADN. Cette structure de la molécule d’ADN est la même
quel que soit l’être vivant étudié (Homme, levure de bière, blé, poule)
POUR S’ENTRAINER A EXTRAIRE DES INFORMATIONS DANS DES DOCUMENTS POUR CONSTRUIRE SA
REPONSE :
ENONCE 2

Je révise sur le chap3 + chap4 – C.MARECAUX – 2
nde
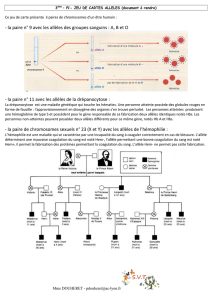
Q1 : utiliser le doc5 ci-dessus pour montrer des différences génétiques entre les 2 populations de lions,
et montrer que la diversité génétique est moindre au sein de la population de lions Ngorongoro.
Q2 : à partir de l’ensemble des documents fournis, proposer une explication aux différences constatées
dans la Q1
EBAUCHE DE
CORRECTION
Q1 : on observe que les gènes 1, 2, 3 et 4 existent dans les 2 populations de lions étudiées.
Toutefois, le nombre et la fréquence de chaque allèle de chaque gène varie.
Variation du nombre d’allèles par gène : au sein de la population de lions du Serengeti, le gène 1
possède 3 allèles (a, b, c), alors qu’il n’existe qu’en 2 versions dans la population de lions Ngorongoro (a
et b).De même, pour les gènes 3 et 4, les lions de Ngorongoro ne possèdent qu’un allèle
(respectivement r et y), alors que les gènes 3 et 4 possèdent 2 allèles chacun dans la population de
Serengeti.
Variation de la fréquence des allèles : regardons le gène 2, qui existe sous 2 versions (allèles m et n)
dans les 2 populations de lions. On remarque que la fréquence de ces allèles est différente : l’allèle m
est très majoritaire (94%) par rapport à l’allèle n (6%), alors qu’au sein de la population de lions de
Serengeti, les fréquences de ces allèles sont de respectivement 74% et 26%. Autrement dit, la
population de lions Ngorongoto est moins diversifiée génétiquement que la population de lions
Serengeti.

Je révise sur le chap3 + chap4 – C.MARECAUX – 2
nde
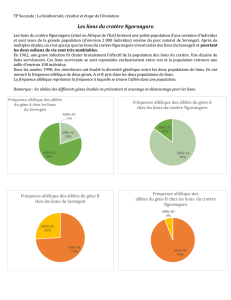
Q2 : On observe qu’au départ, la fréquence des allèles 1, 2 et 3 est la même dans la population de faible
effectif (10individus) ou de fort effectif (500 individus) .
Au cours des générations successives, du fait de la ‘loterie de la reproduction sexuée’ (doc3), on
observe dans le doc4 qu’une population de faible effectif (10 individus) enregistre l’extinction de
l’allèle 2 dès la 12
ème
génération, et l’extinction de l’allèle 1 dès la 25
ème
génération. Au contraire, une
population de grand effectif n’enregistre aucune extinction d’allèle, puisque les allèles 1, 2 et 3 sont
présents en permanence au cours des générations, avec des fréquences qui varient très peu.
La dérive génétique est d’autant plus importante que l’effectif de la population est faible.
J’en conclue donc que la baisse de la diversité génétique observée dans la population de lions
Ngorongoro (voir Q1) s’explique de 2 façons :
- l’infection de 1962 a fait très nettement chuter l’effectif de la population, donc certains allèles
ont du disparaître.
- l’effectif réduit de cette population a amplifié le mécanisme de dérive génétique, qui baisse la
diversité allélique au sein de la population de lions Ngorontoro, et accentue les différences
génétiques entre les 2 populations de lions.
1
/
3
100%