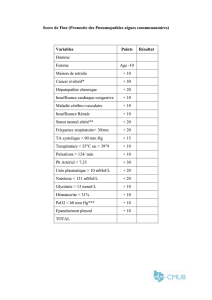
Document n°1. Mesures de vitesses initiales de réactions pyruvate

AT – 0412 Documents page 1/4
Document n°1. Mesures de vitesses initiales de réactions pyruvate kinase (PK)
Pour suivre les cinétiques pyruvate kinase on couple la réaction pyruvate kinase à la réduction du
pyruvate en lactate catalysée par la lactate déshydrogénase (LDH). Le NADH, qui absorbe fortement à
340 nm, est oxydé en NAD+ qui n'absorbe pas à cette longueur d'onde. L'enregistrement de l'absorbance
du système à 340 nm permet le suivi cinétique.
phosphoenolpyruvate (PEP) + ADP = pyruvate + ATP (réaction principale catalysée par la PK)
pyruvate + NADH + H3O+ = lactate + NAD+ + H2O (réaction indicatrice catalysée par la LDH)
Dans le système des deux réactions couplées, la réaction principale, en état de vitesse initiale, devra être
limitante de la réaction indicatrice.
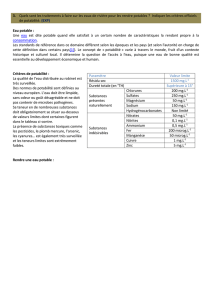
Les milieux réactionnels ont la composition suivante :
tampon réactionnel Tris-HCl 50 mmol.L-1, 0,1 mol.L-1 KCl, 5 mmol.L-1 MgSO4 , pH 7,5 ;
substrats PEP et ADP aux concentrations souhaitées ;
NADH 0,20 mmol.L-1 ;
Lactate déshydrogénase (LDH) selon une quantité conduisant à une vitesse maximale de la
réaction indicatrice dans les conditions du milieu réactionnel de 20 µmol.mL-1.min-1 ;
volume réactionnel total de 1 mL, 37°C, démarrage de la réaction avec 20 µL de préparation
pyruvate kinase à mesurer ajoutés à 980 µL de mélange réactionnel, suivi cinétique en continu à
340 nm.
Document n°2. Données caractérisant le comportement cinétique de la lactate
deshydrogénase (LDH)
La lactate deshydrogénase (LDH) utilisée pour la réaction indicatrice est une LDH de muscle
squelettique de lapin. Le comportement est Michaélien selon un mécanisme bi-bi ordonné avec fixation
première du NADH puis fixation du pyruvate.
Le coefficient de Michaelis pour le pyruvate est Kp = 0,100 mmol.L-1
Le coefficient de Michaelis pour le NADH est KN = 0,010 mmol.L-1
Le coefficient de dissociation du couple LDH-NADH est KsN = 0,002 mmol.L-1
L'équation générale de cinétique est :
vi=Vmax
[
NADH
]
[
pyr
]
[NADH ][ pyr]+ [NADH ]Kp+ [ pyr]KN+KsN Kp
qui peut s'écrire :
(1+ ) (1 )
(1 )
max
iNsN
pN
pyr
V
v = K K
NADH NADH
K + pyr
K
NADH
équation dite équation « doc2 »

AT – 0412 Documents page 2/4
Document n°3. Cinétiques pyruvate kinase (PK) de E. Coli, PEP substrat variable, ADP
paramétrique
Pour l’ensemble des graphes :
- V désigne la vitesse initiale ;
- FBP désigne l'effecteur fructose-1,6-
biphosphate ;
- [PEP] désigne la concentration en PEP dans le
milieu réactionnel ;
- [ADP] la concentration fixe d'ADP dans le
milieu réactionnel (2 mmol.L-1).
Document n°4. Pyruvates kinases (PK) chez le rat
Les mammifères proposent plusieurs isoenzymes. Les PK sont des homotétramères. C'est
l'épissage alternatif exclusif de l'ARN pré-messager du gène M qui conduit aux 2 isoenzymes M1 et M2.
M1 et M2 diffèrent ainsi de 21 résidus aminoacyls sur le segment protéique allant du résidu 388 au résidu
432. Ce segment forme 2 hélices alpha engagées dans l'association des sous-unités en homotétramère.
Dans le document ci-dessous M2.WT désigne l'enzyme M2 de type sauvage et M2.C423L un mutant de
M2 pour lequel la cystéine en position 423 a été remplacée par une leucine.
Pour l’ensemble des graphe :
- en abscisse la concentration en
phosphoénolpyruvate (PEP) en
µmol.L-1 ;
- en ordonnée le rapport vitesse
initiale sur vitesse maximale
(v/Vmax) ;
- la concentration en ADP est fixe
à 2 mmol.L-1 ;
- chaque enzyme est étudiée en
présence ou non de l'effecteur
fructose-1,6-biphosphate (FBP) à
la concentration de 0,5 mmol.L-1.
Note : Les données du document n°2 sont adaptées -afin d'obtenir des calculs simples- de http://biochemifa.kikkoman.co.jp/products/rinsyou/pdf/61170_ldh-
p.pdf et R. A. Stinson and H. Gutfreund Transient-kinetic studies of pig muscle lactate dehydrogenase, Biochem J. 1971 January 121(2): 235–240 (dernière
page). Les données des documents n°1, 3 et 4 sont adaptées de Valentini, Chiarelli ...,The Allosteric Regulation of Pyruvate Kinase, The Journal of Biological
Chemistry, 2000 Vol. 275, No. 24 :18145–18152 et Ikeda, Noguchi, Allosteric Regulation of Pyruvate Kinase M2 Isozyme Involves a Cysteine Residue in the
Intersubunit Contact, The Journal of Biological Chemistry, 1998 Vol. 273 : 12227-12233.

AT – 0412 Documents page 3/4
Document n°5. Carte fonctionnelle du vecteur pLIC-NHis
Légende :
Ori : origine de réplication
Kan : gène de résistance à la kanamycine (phosphotransférase)
lacI : gène codant LacI
T7 : promoteur transcrit par l’ARN polymérase du phage T7
RBS : site de fixation du ribosome
ATG (M) = séquence codant la méthionine initiatrice
TGA = (*) = séquence d’un codon STOP.
Code à une lettre des acides aminés : H= histidine , M=méthionine, V=valine, D= acide aspartique,
E=acide glutamique, N= glutamine
D’après JOURNAL OF BIOMOLECULAR TECHNIQUES, VOLUME 20, ISSUE 5, DECEMBER 2009
Document n°6. Site de reconnaissante et de coupure de l'enzyme PmlI
5' CAC GTG 3'
3' GTG CAC 5'

AT – 0412 Documents page 4/4
Document n°7 : Préparation du vecteur LIC
Double traitement du vecteur pLIC-NHis par pmlI (1.) et l'ADN polymérase (2.) de T4 en
présence de dGTP.
Dans la figure ci-dessous, seule la partie du plasmide concernée par le traitement est représentée.
PmlI
M H H H H H H G
5’CATATGCACCATCATCATCATCACGTGGAAGTGGATAACTGAGATCC 3’
3’GTATACGTGGTAGTAGTAGTAGTGCACCTTCACCTATTGACTCTAGG 5’
1. PmlI
2. T4 DNA polymérase + dGTP
5’CATATG 3’ 5’ GTGGAAGTGGATAACTGAGATCC 3’
3’GTATACGTGGTAGTAGTAGTAGTG 5’ 3’ GACTCTAGG 5’
Document n°8 : ADN à insérer prêt pour le clonage
gène cible
H H H H H H G
5’CAC CAT CAT CAT CAT CAC GGT ATG NNN NNN C 3’
3’CCA TAC NNN NNN G CAC CTT CAC CTA TT 5’
Masse molaire de l'histidine : 155 g.mol-1.
Masse molaire de la glycine : 75 g.mol-1.
1
/
4
100%