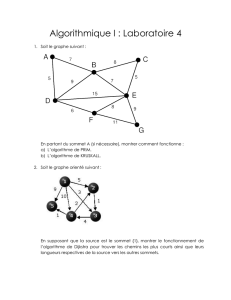
Un graphe génératif pour la classification semi

Un graphe génératif pour la classification
semi-supervisée
Pierre Gaillard *—Michaël Aupetit ** —Gérard Govaert ***
*CEA, DAM, DIF, F-91297 Arpajon, France
pierre.gaillar[email protected]
** CEA, LIST, F-91191 Gif-sur-Yvette, France.
** UTC, U.M.R. C.N.R.S. 6599 Heudiasyc, 60205 Compiègne Cedex, France
gerar[email protected]
RÉSUMÉ. Nous proposons un nouvel algorithme semi-supervisé qui combine un modèle de mé-
lange gaussien pour modéliser localement les données, et un graphe génératif construit sur les
composants du mélange pour capturer la structure globale des données. La combinaison est
réalisée via un processus de propagation d’étiquettes au travers du graphe. Contrairement aux
algorithmes de l’état de l’art, le modèle de graphe utilisé est génératif de telle sorte que son
optimisation peut être effectuée à l’aide de l’algorithme EM (Espérance-Maximisation) afin de
maximiser sa vraisemblance. De plus, l’unique méta-paramètre (le nombre de composants du
mélange) peut être sélectionné par un critère statistique. L’algorithme obtient des résultats ex-
périmentaux similaires aux algorithmes comparables lorsque le nombre de données étiquetées
est faible, et offre l’avantage de n’avoir aucun paramètre à régler manuellement.
ABSTRACT. We introduce a new semi-supervised algorithm based on a generative model. This
model combines a Gaussian mixture model and a generative graph built on the components of
this mixture. The combination corresponds to refit the class membership of the mixture compo-
nent with a propagation process. Both models can be optimized under the maximum likelihood
framework and the only hyper-parameter (number of components of the mixture) can be selected
with a statistical criterion. Experimental results show that we achieve accuracies comparable
to those of rival state-of-the-art algorithms when few labeled data are available. Moreover,
it offers the advantage of defining an objective statistical criterion for tuning its parameters,
cancelling the need for arbitrary hand-tuning.
MOTS-CLÉS : apprentissage semi-supervisé ; modèle de mélange ; graphe génératif ; propagation
ISI, pages 0 à 0

Un graphe génératif pour la classification 1
KEYWORDS: semi-supervised learning ; mixture model ; generative graph ; label spreading

2 ISI
1. Introduction
1.1. Apprentissage semi-supervisé
Etant donné un ensemble de données étiquetées D`={(xi, yi)}M`
i=1, où xi∈X⊂
RDest une donnée observée et yi∈ {1, ..., K}son étiquette parmi un choix de K
classes, l’objectif des approches supervisées est de modéliser la relation existant entre
les données et leur étiquette de manière à répondre à la question suivante : connaissant
une nouvelle donnée, quelle est l’étiquette associée ? Malheureusement, dans de nom-
breuses applications, s’il est aisé d’obtenir les données grâce à un système d’acquisi-
tion automatisé, le processus d’étiquetage est quant à lui généralement long et coûteux
puisqu’il requiert les efforts et l’expérience d’un expert du domaine. Afin de réduire
le besoin de données étiquetées, les algorithmes d’apprentissage dits semi-supervisés,
qui apprennent un classifieur à partir de données étiquetées et non-étiquetées, ont fait
l’objet de nombreux travaux ces dernières années (Chapelle et al., 2006).
Par la suite, on note Dul’ensemble des Mudonnées non-étiquetées, Du=
{xi}Mu
i=1 et D`l’ensemble des M`données étiquetées, D`={(xi, yi)}M`
i=1. Le nombre
total de données est : M=Mu+M`, avec Mu>> M`.
Lorsqu’on dispose de peu de données étiquetées, l’idée générale des approches
semi-supervisées est d’exploiter la structure géométrique de la distribution des don-
nées observées P(x), qui est fournie par les seules données non-étiquetées. Ceci pro-
vient de deux hypothèses généralement admises (Chapelle et al., 2003).
–Hypothèse locale : si deux données sont proches dans une zone de forte densité,
alors elles devraient appartenir à la même classe, ce qui implique que la frontière de
décision doit passer par des régions de faible densité.
–Hypothèse globale : les données sont proches d’un ensemble de variétés dont
la dimension intrinsèque est plus faible que la dimension de l’espace d’observation.
Puisque les variétés définissent des chemins de forte densité, on obtient par transiti-
vité de la première hypothèse que des données issues de la même variété devraient
appartenir à la même classe (Bousquet et al., 2004).
1.2. Plan
Dans les deux Sections suivantes, nous présentons les différents algorithmes de
l’état de l’art en les caractérisant comme étant soit génératifs, soit discriminatifs
(Rubinstein et al., 1997). La section 2 introduit les approches génératives qui tentent
de décrire le processus de génération des données (et donc les variétés) en estimant
la distribution jointe P(x, y). La section 3 introduit les approches discriminatives
qui ont pour seul objectif de définir une fonction h, liant les données et les étiquettes
(y=h(x)) de manière à minimiser une erreur de classification. Nous verrons au tra-
vers de cette section comment ces approches utilisent un graphe de proximité construit
sur les données pour capturer la géométrie des variétés.

Un graphe génératif pour la classification 3
Dans la section 3 nous proposons un nouvel algorithme semi-supervisé basé sur un
modèle de mélange gaussien (Miller et al., 1996) et sur le graphe génératif gaussien
(Aupetit, 2006). Nous comparons dans la section 4 les performances des différents
algorithmes avant de conclure dans la section 5.
2. Approches génératives
2.1. Modèles de mélange
Dans le contexte de l’apprentissage semi-supervisé, les modèles génératifs ont lar-
gement été utilisés et étudiés puisqu’ils permettent de modéliser les densités P(x, y)
et P(x). La structure complexe des données est localement capturée à l’aide d’une
somme finie et pondérée de densités simples et usuelles. Par exemple, Landgrebe et
al. (1978) et Nigam et al. (2000) utilisent respectivement un modèle de mélange de
densités gaussiennes et multinomiales pour la classification d’images et de textes.
Miller et al. (1996) proposent un modèle de mélange gaussien, où chaque densité
est associée à une ou plusieurs classes :
p(x, y;θ) =
N
X
n=1
p(n)p(y|n)p(x|n) =
N
X
n=1
πnβnyg(x|wn; Σn)[1]
où θest l’ensemble des paramètres du modèle décrit ci-dessous.
Les densités gaussiennes gsont appelée les composants du mélange et elles sont
paramétrées par leur moyenne wn∈RDet leur matrice de covariance Σn∈RD×
RD:
g(x|wn; Σn) = 1
(2π)D/2|Σn|1/2exp −(x−wn)TΣ−1
n(x−wn)[2]
où |Σn|est le déterminant de la matrice de covariance Σn.
Les paramètres π={πn}N
n=1 représentent les proportions de chaque densité gaus-
sienne dans le mélange : πnest la probabilité qu’une donnée soit issue du necompo-
sant. Elles vérifient les deux contraintes suivantes :
N
X
n=1
πn= 1 et πn≥0∀n[3]
Les paramètres β={βnk =p(y=k|n)|∀n, k}représentent la probabilité qu’une
donnée observée xsoit de la classe ksachant qu’elle est issue du necomposant. Les
paramètres βvérifient donc :
K
X
k=1
βnk = 1 ∀net βnk ≥0∀n, k [4]

4 ISI
En utilisant ce modèle paramétrique, on peut facilement utiliser les données non-
étiquetées pour exprimer la densité p(x;θ). Pour cela, il suffit de marginaliser la den-
sité jointe p(x, y;θ)par rapport à y. En utilisant le fait que PK
k=1 βnk = 1 pour tout
n={1, ..., N},ona:
p(x;θ) =
K
X
k=1
p(x, y =k) =
N
X
n=1
πng(x|wn; Σn)[5]
2.2. Estimation des paramètres
Le critère naturel pour estimer les paramètres du modèle est la vraisemblance
jointe des données étiquetées et non-étiquetées, qui s’exprime comme le produit
des deux vraisemblances puisque les données sont supposées i.i.d (indépendantes et
identiquement distribuées) (Seeger, 2001). Les estimateurs du maximum de vraisem-
blance des paramètres peuvent être déterminés aisément à l’aide de l’algorithme EM
(Dempster et al., 1977).
L(θ;D`,Du) = L(θ;D`)L(θ;Du) =
M`
Y
i=1
p(xi, yi;θ)
Mu
Y
i=1
p(xi;θ)[6]
2.3. Discussion sur les approches génératives
Les modèles de mélange présentent plusieurs avantages : (1) ils respectent par
construction l’hypothèse locale ; (2) ils permettent par nature de classifier via la règle
de Bayes des données non disponibles lors de l’apprentissage : on parle d’induction ;
(3) le seul méta-paramètre (le nombre de composants N) peut être facilement sé-
lectionné par validation croisée ou à l’aide d’un critère statistique tel que BIC
(Schwartz, 1978) ; (4) ils sont capables, à l’aide de l’algorithme EM, de traiter des
données incomplètes (Ghahramani et al., 1994).
En revanche, même si les modèles de mélange modélisent bien les données loca-
lement, ces méthodes ne prennent pas en compte la structure sous-jacente des don-
nées puisqu’aucune relation géométrique entre les différents composants n’est défi-
nie. Lorsque les variétés sont complexes, l’hypothèse globale ne peut pas être prise en
compte avec des densités simples (figure 1).
3. Approches discriminatives
La plupart des approches discriminatives apprennent un classifieur hen pren-
nant en compte la structure des données via un graphe de proximité des données.
Couramment, il s’agit du graphe des kplus proches voisins. Plus précisément, la
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
1
/
25
100%