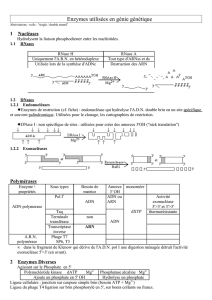
Brin transcrit : chaine de l`ADN qui, par complémentarité

1
Fin du cours chap 5
La transcription : de l’ADN à l’ARN :
Par définition, le brin transcrit est le brin d’ADN complémentaire de l’ARN : c’est le brin qui sert à la synthèse de l’ARN, donc ici
le brin2.
La transcription permet de passer du brin transcrit de l’ADN à l’ARN messager par la règle de complémentarité des nucléotides
: dans l’ARN, U est complémentaire de A.
La notion de brin codant de l’ADN est à éviter, il peut porter l’élève à des confusions, surtout que la littérature scientifique
définit le brin codant comme étant le brin non transcrit, celui que l’on « recopie » en remplaçant T par U pour obtenir l’ARN.
L'ARN messager (ARNm) est une molécule qui sert d'intermédiaire entre le noyau et le cytoplasme. Elle
est comparable à l'ADN avec quelques différences:
- le sucre est du ribose (C5H10O5),
- la base azotée thymine (T) est remplacée par l'uracile (U),
- la molécule n'est formée que d'une seule chaîne de nucléotides,
- la longueur de la molécule est nettement plus faible que celle de l'ADN, car elle n'en représente que la
copie d'un seul gène (masse molaire: entre 25 000 et 500 000).
- la durée de vie de l'ARN est très courte (quelques minutes à quelques dizaines de minutes).
Etapes de la transcription :
Brin transcrit : chaine de l’ADN qui, par complémentarité des nucléotides, sert de modèle pour la synthèse
de l’ARN messager
Brin non transcrit : deuxième brin d’ADN, complémentaire du brin transcrit.

2
Le passage ADN → ARN m est appelé trancription
La transcription nucléaire consiste à "copier" une portion d'ADN en une information identique sous forme
d'ARNm.
Cette opération s'effectue dans le noyau:
- ouverture et déroulement d'une portion de la double hélice
- synthèse d'un brin d'ARNm à partir du brin transcrit de l'ADN, grâce à un complexe enzymatique:
l'ARN-polymérase, qui incorpore des nucléotides par complémentarité A-U, C-G, T-A, G-C. Plusieurs
ARN-polymérases se succèdent le long de l'ARNm, engendrant une amplification de la transcription. Des
signaux existent sur l'ADN, qui assure une régulation de l'expression des gènes.
Pb : Comment se réalise la synthèse des protéines à partir de l’ARNm ?
La traduction de l’ARNm en protéine :
1. Le code génétique :
Livre p 56-57
Capacités et attitudes :
Mettre en œuvre une méthode (démarche historique) et/ou une utilisation de logiciels et/ou une pratique documentaire
permettant de comprendre comment le code génétique a été élucidé.
Historiquement, plusieurs hypothèses ont été formulées quant à la nature du code génétique (voir page 66, « Des clés pour…
mieux comprendre l’histoire des sciences », le premier code proposé par Gamow). L’expérience de Crick décrite dans le
document 1 (voir « ressources complémentaires ci-dessous) a permis de montrer que le code génétique est non recouvrant et
qu’il est formé de triplets de nucléotides. On pourra montrer à travers l’exemple d’une phrase du type « TON AMI LEO EST ICI
» que l’insertion d’une lettre en gardant le même découpage des mots (3 lettres) rend la suite du message incompréhensible,
ce qui n’est pas le cas lorsque l’insertion est celle d’un nombre de lettres multiple de 3. À la même époque, Nirenberg réalise
les expériences décisives citées dans le document 2 en travaillant sur des fractions cytoplasmiques différentes, recréant in
vitro un milieu de traduction contrôlé (voir « ressources complémentaires » ci-dessous). Les résultats originaux exprimés en
dosage de radioactivité dans les protéines formées ont ici été rapportés aux témoins de chaque expérience, de façon à
différencier ce qui est un dosage significatif de ce qui ne l’est pas. Ces résultats valident le code génétique présenté dans le
document 3. Le document 4 permet un réinvestissement des notions acquises lors de l’étude de la transcription et propose
une étude des conséquences des mutations en appliquant le code génétique. Les exceptions à l’universalité du code génétique
sont abordées dans l’exercice 8 page 69.

3
Doc. 1 et 3 : Lorsque le nombre de nucléotides insérés ou supprimés n’est pas un multiple de trois, le message génétique n’est
plus interprété correctement. La taille des codons est donc de trois nucléotides.
Doc. 2 : Le codon UUU correspond à l’incorporation de la phénylalanine et pas d’un autre acide aminé. Les codons AAA et CCC
ne correspondent pas à la phénylalanine.
Doc. 3 et 4 : Le 7e triplet TTC au lieu de CTC. L’ARN correspondant sera AAG au lieu de GAG, soit une lysine en position 7 à la
place d’un acide glutamique.
À partir du 10e triplet, insertion d’un nucléotide G. Le premier triplet AGA est remplacé par GAG, soit, sur l’ARN
correspondant, UCU au lieu de CUC, et donc leucine au lieu de sérine. Tous les acides aminés suivants sont également
modifiés, puisque la lecture de tous les codons se trouve décalée.
L’information contenue dans la séquence de l’ARNm détermine la séquence de la protéine formée grâce à un
système de correspondance : trois nucléotides successifs forment un codon auquel correspond un acide aminé
déterminé, toujours le même. Le système de correspondance entre les codons et les acides aminés est le code
génétique.
2. La traduction :
→ Exercice sur la traduction
→ livre p 58 doc 1 : les intervenants :
Capacités et attitudes : Mettre en œuvre une méthode (démarche historique) et/ou une utilisation de logiciels et/ou une
pratique documentaire permettant d’approcher le mécanisme de la transcription, et de la traduction.
Le document 1 situe la traduction dans son contexte biologique et présente le rôle des ribosomes. L’organisation du polysome
pourra être mise en parallèle avec celle des unités de réplication du document 4 page 55. La structure tridimensionnelle du
ribosome est complexe et probablement trop difficile à réaliser avec les logiciels de visualisation moléculaire utilisés en classe.
Sur cette image, c’est une vue simplifiée qui est présentée, après en avoir effacé les protéines ribosomales (ne subsistent que
les ARNt, ARNr et ARNm). La légende « système de lecture des codons » correspond en fait aux ARNt.
La localisation du site catalytique serait donc au point d’insertion de ces ARNt dans la grosse sous-unité. Le polypeptide en
cours de formation est exporté via un tunnel non visible ici qui traverse la grosse sous-unité.
→ Schéma traduction :

4
http://lewebpedagogique.com/arnaud/files/2011/05/synth.gif
L’information portée par l’ARNm est « lue », codon après codon, par un ribosome qui associe l’acide aminé
correspondant au reste de la protéine déjà synthétisée. La traduction débute au niveau d’un codon d’initiation
AUG et s’arrête après rencontre d’un codon stop. C’est la traduction.
II- LA MATURATION DES ARN MESSAGERS
A- Le morcellement des gènes eucaryotes :
Livre p 60 document 1 et 2
Capacités et attitudes :
Mettre en œuvre une méthode (démarche historique) et/ou une utilisation de logiciels et/ou une pratique documentaire
permettant d’approcher le mécanisme de la transcription, et de la traduction.

5
La comparaison graphique des séquences apportée par le document 1 a pour but de mettre en évidence les portions
identiques (exons) et les portions différentes (introns) entre ARNm et ARN pré-messager. Le guide pratique (page 343)
présente une explication plus détaillée du principe du dotplot et de son interprétation.
Sur le plan manipulatoire, le dotplot réalisé par le logiciel Anagène nécessite un temps de calcul important. Il est donc adapté
pour l’étude de gènes courts possédant peu d’introns. Sans entrer dans les détails de cette opération logicielle, il suffit de
constater qu’il n’y a identité entre les séquences d’ADN et d’ARNm que par tronçons.
Une autre approche consiste à étudier une image d’hybridation entre l’ADN d’un gène et l’ARN messager correspondant (voir
exercice 10 page 70). Malgré les apparences, le dotplot est cependant une mise en évidence plus directe.
Le document 2 pourra être utilisé comme une aide à la lecture du dotplot et à son interprétation. Il permet d’introduire le
vocabulaire propre à l’épissage.
→ Q1 Doc. 1 : L’ARN pré-messager est beaucoup plus long (1638 pb contre 444 pb) que l’ARNm. Trois portions (entre 100 et
200, entre 300 et 500, puis entre 1 400 et 1 500 pb) sont communes entre les deux séquences et forment l’intégralité de
l’ARNm. Les quatre autres portions ne sont pas retrouvées dans l’ARN messager.
→ Q 2 :Doc. 1 et 2 : Les portions du dotplot révélées par des diagonales correspondent aux exons. Les segments entre les
exons successifs correspondent aux introns.
Belin Edition 2011
 6
6
 7
7
 8
8
1
/
8
100%