– UE 7: Biologie cellulaire et moléculaire– - Fichier

2016-2017 Différenciation cellulaire
Différenciation cellulaire
– UE 7: Biologie cellulaire et moléculaire–
Pas d'annexe
Semaine : n°18 (du 06/02/17 au
10/02/17)
Date : 10/02/2017
Heure : de 9h00 à
10h00 Professeur : Pr. Lestavel
Binôme : n°5 Correcteur : n°34
Remarques du professeur :
- Il faut s'inscrire sur moodle pour recevoir les informations
- Diapos important sur moodle
- Apprendre les exemples dans la partie cellules souches +++
- peu d'étudiants en cours
- important d'aller en ED
PLAN DU COURS
III) Différenciation cellulaire
A) Principe et méthode d'étude
B) Définitions
C) Comment deux cellules filles qui ont le même génome peuvent avoir un
phénotype différent ?
1) Par division asymétrique
2) Par pression de l'environnement / notion de « niche »
3) Par rétro-contrôle positif
D) Les cellules souches
1) Les cellules souches embryonnaires (ES)
2) Les cellules souches adultes
E) Les signaux de différenciation
1/13

2016-2017 Différenciation cellulaire
III) Différenciation cellulaire
A) Principe et méthode d'étude
Dans notre corps, on a 200 types cellulaires différents que l'on peut identifier par analyse cytologique.
Il y a 3 types de différenciation différentes :
•La différenciation morphologique et structurale : On le fait par MO, ME, MET, MOF ...
•La différenciation biochimique et physiologique : Les cellules sont en présence de molécules et de
protéines particulières avec des fonctions métaboliques particulières.
•La différenciation moléculaire:On regarde la diversité quantitative et qualitative des ARNm et des
protéines. On peut analyser la diversité grâce à plusieurs techniques :
▪Électrophorèse bi-dimensionnelle (protéines) sur gel poly-acrylamide: On va les séparer en
fonction du pH isoélectrique (pHi) et en fonction de leur masse. Chaque spot va constituer une
protéine ou un ensemble de protéines qui sera déterminé par le pHi et la masse moléculaire.
On a toutes les protéines de la cellule qui seront exprimées dans un échantillon donné. Les
spécialistes connaissent les cartes d'électrophorèse bidimensionnelle de plusieurs molécules.
L'électrophorèse est différente en fonction du tissu humain. On peut bloquer et transférer les gènes
de l'électrophorèse dans un gel de nitrocellulose, et immunologique avec un anticorps spécifique.
Mais cette technique est difficile à réaliser dans un labo ou TP, c'est pourquoi on fera plutôt un
Western Blot.
Exemple : Analyse du spectre d'une tissu de cerveau humain et d'un tissu de foie humain.
▪Western blot (protéines): on obtient une bande observée, colorée avec un colorant non spécifique
de toute les protéines, on met sur une feuille de nitrocellulose
On peut apprécier la présence d'une protéine, et l'intensité de la bande qui sort et donne
l'abondance de la protéine.
C'est une séparation des protéines en fonction de leur taille.
▪PCR quantitative : On peut étudier la présence d'ARNm en amont d'une protéine par PCR
quantitative bactérienne ( faite quotidiennement en laboratoire ). Cela suppose que l'on connaît ce
que l'on cherche on cherche l'ARNm d'une protéine d’intérêt et avec des sondes, on amplifie une
partie du gène, et on peut regarder l'abondance en ARNm comparé à l'abondance d'un ARN de
▪Les puces à ARN (ARN): Quand on veut regarder le transcriptome ( ensemble des gènes
différentiellement exprimés dans un échantillon par rapport a un autres ) on fait les puces à ARN .
On va extraire tout les ARN d'un échantillon, on aura 2 échantillons auxquels on aura extrait tous
les ARNm, on va tous les amplifier et comparer leur abondance.
On peut comparer le transcriptome d'un, de deux ou plusieurs échantillon. On va pouvoir voir dans
tel échantillon que gène est exprimé, on sort des tableaux avec le niveau d'expression des gènes.
Les ARNm d'un échantillon seront marqués par une sonde fluorescente rouge et ceux de l'autre
échantillon par un fluorochrome vert. Les ARNm vont s'hybrider en fonction de leur structure, de
2/13

2016-2017 Différenciation cellulaire
leur séquence. Les spots de la puce à ARN vont donner une certaine fluorescence : quand l'ARNm
rouge va se fixer on aura une tache rouge, idem pour l'ARNm vert.On peut avoir une compétition
car dans les deux échantillons on peut avoir le même ARNm qui est exprimé.
La couleur va dépendre de la quantité d'ARNm exprimés dans les échantillons : on va pouvoir
déterminer l'abondance des ARNm présents dans les échantillons.
B) Définitions
Différenciation cellulaire: C'est la capacité d'une cellule non spécialisée à donner, dans un endroit donné
différent type cellulaires fonctionnels. Cela est possible par activation ou inactivation de certains gènes
de façon extrêmement régulée.
Cellule souche → Cellule engagée → Cellule différencié
Les cellules souches se divisent à l'identique pour redonner une cellule souche et donne une cellule engagée. La
cellule engagée se divisent de nombreuses fois pour donner un pool de cellules différenciées.
Toutes les cellules ont le même génome , mais chaque cellule spécialisée de l'organisme dans un organe donné va
exprimer un groupe de gènes différents. Il n'y a pas de modification du génome dans les différentes cellules des
différents organes, c'est une expression différente. On n'acquiert pas de nouveaux gènes, on éteint l'expression de
certains gènes. ++ . La différenciation résulte de l'expression sélective d'un panel de gène.
Vocabulaire à connaître :
3/13

2016-2017 Différenciation cellulaire
–Stem cell : cellule souche
–Committed cell : cellule engagée (progéniteur)
C) Comment deux cellules filles qui ont le même génome peuvent avoir un
phénotype différent ?
1) Par division asymétrique
Une cellule peut ségréger des granules P à un endroit particulier dans son cytoplasme, et lorsqu'il y aura divisions
asymétriques, l'une des cellules filles ne va pas obtenir ces molécules alors que l'autre va obtenir toutes ces
molécules et aura un comportement différent. Par exemple ce sont des facteurs de transcription, des molécules de
signalisation, d'où l'importance du cytoplasme dans la régulation.
Exemple : Division asymétrique dans la lignée germinal de C.Elegan, cette cellule fondatrice ségrége des granules
P, quand la cellule se divise dans ses plans, seule la cellule de droite aura les granules P
2) Par pression de l'environnement / notion de « niche »
Quand une cellule mère va se diviser, soit les deux cellules filles pourront rester dans l'environnement de la cellule
mère car il y a de la place, soit il n'y a de la place que pour une seule cellule, à ce moment là, une cellule fille va
être éjectée de la niche, cette cellule ne va pas subir la même pression de l'environnement que la deuxième cellule
fille qui restera dans la niche. L'environnement des cellules souches les maintient dans leur état indifférencié.
Il y a des niches qui permettent le maintient dans un état indifférencié.
Dès l'instant où la cellule va être soumise à un environnement différent, cela va la conduire à un comportement
différent par des signaux intra et extracellulaire. Ce tout va donc induire une différenciation.
4/13

2016-2017 Différenciation cellulaire
3) Par rétro-contrôle positif
Il faut imaginer que les deux cellules filles ont le même génome, elles viennent de la même cellule mère, elles
produisent une substance qui va dans l'environnement inhiber la cellule voisine. Les cellules sont identiques donc
produisent la même quantité d'inhibiteur. Elles s'inhibent l'une et l'autre. Dans l'environnement souche, les cellules
sont égales car elles vont s'inactiver et s'inhiber de façon équivalent pour avoir un comportement harmonieux.
Il arrive à un moment ou des signaux peuvent apporter une légère asymétrie : une des cellule va gagner sur l'autre.
La pression négative sera plus importante, et au final on va avoir amplification de l’asymétrie, et on obtiendra
deux cellules différentes.
On obtiendra deux possibilités avec une mémoire stable : on avait deux cellules identiques et on obtient deux
cellules finales différentes qui pourront produire des cellules différentes.
D) Les cellules souches
3 propriétés : à connaître ++
•Capacité d’auto renouvellement : division à l'identique → formation d'un clone cellulaire
•Capable de se diviser indéfiniment
•Capable de se différencier elles ne se différencient pas forcement mais en ont la capacité.
Il y a 4 catégories de cellules souches : à connaître ++
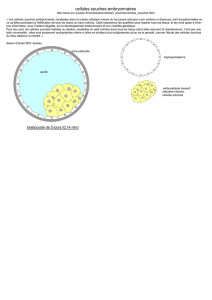
•Totipotentes :C'est une cellule qui est capable de donner toutes les lignées cellulaires d'un organisme, y
compris les cellules extra embryonnaire. ( ce qui permet à l'embryon de survivre ) Chez les mammifères,
seule le zygote et le blastomères issus de la première division cellulaire sont des cellule totipotentes : elles
vont donner naissance à l'embryon et ses annexes embryonnaires. La prof a insisté sur le vocabulaire,
important à connaître
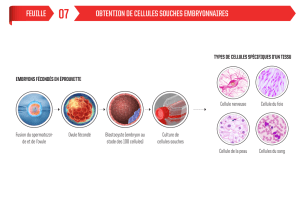
•Pluripotentes : C'est une cellule pouvant donner toutes les lignée cellulaires d'un organisme sauf les
cellules extra embryonnaires. Ce sont les cellules souches embryonnaires (ES) dans le blastocyste et les
cellules souches foetales.
•Multipotentes : C'est une cellule qui est capable de donner les différents types de cellules spécialisées
dans une lignée donnée, comme les cellules souches hématopoïétiques. Elles ont moins de capacités.
•Unipotentes: C'est une cellule qui est capable de donner qu'un seul type de cellule spécialisé comme les
spermatogonies.
5/13
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
1
/
13
100%