Corrélation entre les anomalies centrosomiques et l`ADN

doi:10.1684/abc.2011.0525
181
Pour citer cet article : Sakr R, Bernaudin JF, Daraï É, Fleury J, Prengel C, Rouzier R, Roynard P, Uzan S. Corrélation entre les anomalies centrosomiqueset
l’ADN-ploïdie dans le cancer du sein. Ann Biol Clin 2011 ; 69(2) : 181-9 doi:10.1684/abc.2011.0525
Article original
Ann Biol Clin 2011 ; 69 (2) : 181-9
Corrélation entre les anomalies centrosomiques
et l’ADN-ploïdie dans le cancer du sein
Are centrosomal abnormalities correlated to DNA ploidy in breast cancer?
Rita Sakr1
Jocelyne Fleury2
Claudie Prengel2
Patricia Roynard2
Émile Daraï1
Serge Uzan1
Roman Rouzier1
Jean-Franc¸ois Bernaudin2
1Hôpital Tenon,
Service de gynécologie,
Paris
<ritasakr@yahoo.com>
2Université Pierre-et-Marie-Curie,
Laboratoire d’histologie et de biologie
tumorale - UPRES 3499, Paris
Article rec¸u le 21 juillet 2010,
accept´
e le 28 octobre 2010
Résumé. Plusieurs études décrivent le rôle de l’ADN-ploïdie dans les instabi-
lités génomiques des cellules cancéreuses et le pronostic. Le centrosome jouant
un rôle important dans le cycle cellulaire, de nouveaux facteurs pronostiques
pourront être introduits pour mieux cibler les traitements. L’objectif de ce travail
est de rechercher dans le cancer du sein une anomalie centrosomique et la corré-
lation avec l’ADN-ploïdie. Des lames sont réalisées à partir des échantillons de
prélèvements mammaires et des suspensions cellulaires (cellules mésothéliales
et lignées cellulaires MRC5, MCF7, T47D). L’immunofluorescence indirecte
est utilisée avec l’anticorps anti-␥-tubuline puis un fragment F(ab)2conjugué
au fluoro-isothyanate. Les centrosomes sont quantifiés au microscope à fluores-
cence. L’ADN-ploïdie est définie par l’index d’ADN analysée par cytométrie
en flux. Les cellules mésothéliales normales (94 % des cellules avec un centro-
some) et les cellules MRC5 diploïdes cyclantes (68 % avec deux centrosomes)
sont des contrôles de qualité des expériences. Une corrélation entre l’ADN-
ploïdie et le nombre de centrosomes est retrouvé dans les lignées MCF7 (64 %
des cellules avec un nombre de centrosomes ≥3) mais pas dans les lignées T47D
et les dix échantillons de carcinome invasif du sein analysés dans cette étude.
L’absence de relation entre l’ADN-ploïdie et le nombre de centrosomes pour-
rait être due au petit nombre d’échantillons mammaires étudiés, aux empreintes
cellulaires ou au mécanisme tumoral. Un élargissement de l’étude sur un grand
nombre d’échantillons et de pathologies mammaires et l’étude des anomalies
morphologiques du centrosome sont prévus.
Mots clés : centrosome, ADN-ploïdie, cancer du sein
Abstract. ADN ploidy was shown to play a role in genomic instability of cancer
cells and prognosis. The implication of the centrosome in the cell cycle was also
described. Therefore, new prognostic factors could be suggested for a better-
tailored therapy. The purpose of this study is to search for correlation between
centrosomal abnormality and ADN ploidy in breast cancer. Cell prints were
prepared from cell culture of mesothelial ascitis, fibroblast cell line MRC5 and
breast cancer cell lines MCF7 and T47D. Fresh cell prints were also obtained
from cases with invasive carcinoma. The centrosome was labelled by an indi-
rect immunofluorescence assay using anti-␥-tubulin antibody and F(ab)2FITC
before quantification with fluorescence microscopy. ADN ploidy was scored
with DNA index obtained by means of flux cytometry. The normal mesothelial
cells (94% of cells with only one centrosome) and the diploid cell line MRC5
(68% of cells with two centrosomes) were used as controls. DNA ploidy was
found to be correlated with centrosomal abnormality in MCF7 cell line (64%
of cells had more than three centrosomes) but not in the 10 cases of invasive
ductal carcinoma analysed in this study. The absence of correlation between
DNA ploidy and centrosomal abnormality in breast cancer samples may be due
to the small numbers of cases, the cell prints or tumorigenesis. Correlation ana-
lysis of a larger number of cases and types of breast lesions to numerical and
morphological abnormalities of the centrosome are ongoing.
Key words: centrosome, DNA ploidy, breast cancer
Tirés à part : R. Sakr
Copyright © 2017 John Libbey Eurotext. Téléchargé par un robot venant de 88.99.165.207 le 25/05/2017.

182 Ann Biol Clin, vol. 69, n◦2, mars-avril 2011
Article original
Le cancer du sein est le cancer le plus fréquent chez la
femme. Environ 40 000 nouveaux cas par an sont diagnos-
tiqués en France et c’est la première cause de décès par
cancer chez la femme entre 35 et 55 ans. Le dépistage et
le diagnostic reposent sur l’imagerie et l’examen anatomo-
pathologique et la stratégie thérapeutique sur un contrôle
locorégional ainsi que général de la maladie [1]. Pour le
choix des stratégiques thérapeutiques, il s’est révélé impor-
tant de définir des classes de pronostics différents afin de
mieux cibler les traitements tout en respectant le rapport
bénéfice/risque [2, 3]. Par ailleurs, même en tenant compte
des facteurs de pronostic les plus pertinents, un nombre non
négligeable de patientes vont rechuter, justifiant l’intérêt
d’élargir la liste des facteurs pronostiques et prédictifs déjà
identifiés.
L’évaluation de la cinétique cellulaire n’est pas standardi-
sée, mais l’étude de la prolifération cellulaire s’est révélée
utile dans les choix thérapeutiques. Elle est évaluée par le
pourcentage de cellules en phase S ou fraction de cellules en
phase S (FPS) pouvant être estimée par cytométrie en flux
(CMF) et par l’étude d’antigènes associés au cycle cellu-
laire (Ki67). L’ADN-ploïdie étudiée en CMF est un reflet
des instabilités chromosomiques ou génomiques des cel-
lules cancéreuses. Si la cellule ne possède plus le contrôle
signalant que la mitose est anormale, elle poursuit sa divi-
sion et produit des cellules aneuploïdes. Des études récentes
ont montré le rôle important de l’ADN-ploïdie et de la FPS
comme facteurs prédictifs de rechute dans les cancers du
sein [4-10].
Le centrosome (centre de la cellule) est le centre organi-
sateur des microtubules et se trouve près du noyau [11].
Il comprend deux centrioles organisés dans un matériel
péricentriolaire mal défini appelé matrice (␥-tubuline) et
le cycle du centrosome est distinct du cycle cellulaire. La
duplication du centrosome débute à la transition G1/S. Les
deux centrioles fils arrivent à maturité en taille et en struc-
ture à la fin de G2, parallèlement à la croissance de la matrice
péricentriolaire (MPC). À la mitose, les deux centrosomes
sont matures, ont acquis le maximum de MPC et peuvent
se séparer. Le cycle de division cellulaire dépend de l’aster
microtubulaire qui est organisé par le centrosome. Le cen-
trosome joue un rôle important dans le contrôle de la ploïdie
des cellules filles ; comme l’ADN, il est dupliqué une seule
fois par cycle cellulaire sous la dépendance du point de res-
triction G1/S [12]. Avant son entrée en mitose, la cellule
doit contenir deux lots identiques d’ADN et deux centro-
somes soit quatre centrioles. La présence d’anomalies du
centrosome provoquerait une aneuploïdie par défaut ou ano-
malie de formation du fuseau mitotique ou par défaut de
cytokinèse. Une aneuploïdie associée à un nombre anor-
mal de centrosomes est souvent observée dans les cellules
tumorales [13, 14]. Dans les cancers du pancréas et du
sein, des anomalies de taille, de forme ou de nombre du
centrosome sont fréquemment observées [15, 16]. Malgré
ces différentes études corrélant les anomalies centroso-
miques au processus tumoral, la relation cause à effet n’a
pas été encore clairement démontrée [17].
Ilyaunsiècle, Théodore Boveri avait prédit que la
moindre erreur au cours de la mitose pouvait aboutir à
une instabilité chromosomique à l’origine du cancer. Des
anomalies cellulaires comme l’augmentation du nombre
des centrosomes augmentent le risque de formation de
fuseaux multipolaires qui conduisent à l’aneuploïdie, insta-
bilité génétique la plus fréquemment observée dans de
nombreux cancers chez l’homme et d’autres mammi-
fères [18, 19]. Les cancers du sein et de la vessie ont
été les premiers pour lesquels les mesures de corrélation
entre l’aneuploïdie et la tumorigenèse ont été effectuées,
l’aneuploïdie étant associée à un mauvais pronostic dans
les carcinomes mammaires [20, 21]. Parmi les mécanismes
de contrôle de la mitose, nous venons de voir que le centro-
some joue un rôle important. Cependant, il n’a pas encore
été démontré de relation entre les anomalies du centrosome
et l’agressivité des tumeurs. Par ailleurs, l’ADN-ploïdie
est un bon reflet de l’instabilité génomique et a été
démontrée liée aux facteurs de pronostic dans les cancers
du sein.
L’objectif de notre étude est, d’une part, l’analyse des ano-
malies du centrosome dans le cancer du sein et, d’autre part,
la recherche d’une relation entre le nombre de centrosomes
et l’ADN-ploïdie et les signes de prolifération cellulaire
comme la FPS, dans le but de rajouter des outils applicables
aux empreintes cellulaires.
Matériel et méthodes
Lignées cellulaires de contrôle
Dans l’objectif d’avoir un contrôle de cellules épithéliales
normales, du liquide d’ascite non carcinomateux est uti-
lisé. Deux litres de liquide d’ascite sont répartis dans des
tubes de 50 mL et centrifugés. Les culots sont lavés plu-
sieurs fois dans du PBS 10 % SVF après lyse des hématies
et les cellules sont mises en suspension dans du PBS. La
culture des différentes cellules étudiées se fait dans une
étuve à CO25%à37
◦C dans un milieu comprenant du
RPMI 1640 (Gibco) ou du DMEM 1880 (Gibco) supplé-
menté avec 10 % de SVF (Gibco), de la L-glutamine 5 mM
(Sigma), de la pénicilline 50 U/mL (Sigma), de la strepto-
mycine 50 g/mL (Sigma) et de l’hepes 20 mM (Gibco).
Le milieu de culture est renouvelé deux à trois fois par
semaine. Une fois arrivées au degré de confluence désiré,
les cellules sont trypsinées à 37 ◦C pendant cinq à dix
minutes (trypsine-EDTA 1 X, Gibco) puis mises en sus-
pension dans un volume égal de milieu de culture. Les
suspensions cellulaires sont ensuite centrifugées à 1 000 g
Copyright © 2017 John Libbey Eurotext. Téléchargé par un robot venant de 88.99.165.207 le 25/05/2017.

Ann Biol Clin, vol. 69, n◦2, mars-avril 2011 183
Anomalies centrosomique et ADN-ploïdie dans le cancer du sein
pendant quatre minutes à température ambiante. Le surna-
geant est éliminé et les cellules sont remises en suspension
dans du PBS. Pour la mise au point du marquage de
cellules diploïdes cyclantes et l’examen du cycle du cen-
trosome, les lignées de fibroblastes MRC5 sont étudiées à
30, 60 et 90 % de confluence. Pour celle des cellules aneu-
ploïdes, les lignées de cancer du sein MCF7 et T47D sont
cultivées.
Immunocytochimie
Des échantillons de tissu mammaire sont prélevés par
l’anatomopathologiste conformément à l’avis du Comité
de protection des personnes et sont conservés dans l’azote
liquideà-80
◦C. Après décongélation des prélèvements,
des empreintes cellulaires sont réalisées sur lame. Une lame
est colorée par le MGG pour vérifier la qualité de la prépa-
ration cellulaire et les deux autres lames sont séchées à l’air
pendant 24 heures puis congeléesà-20
◦C sans fixation.
Les suspensions cellulaires sont concentrées à 106cel-
lules/mL. Des pastilles de cytocentrifugation sont préparées
avec 100 L de cette suspension par lame (200 g pendant
six minutes). Pour chaque type cellulaire, une lame est colo-
rée par le MGG pour contrôler la qualité cellulaire et, pour
le liquide d’ascite, confirmer la richesse en cellules méso-
théliales. Les lames restantes de chaque type cellulaire sont
séchées à l’air pendant une nuit puis congeléesà-20
◦C
jusqu’à l’utilisation.
La méthode d’immunomarquage utilisée est une immuno-
fluorescence indirecte. Les lames préparées (empreintes
ou pastilles de cytocentrifugation) sont décongelées pen-
dant cinq minutes à température ambiante. Les cellules
mésothéliales ont servi de contrôle de cellules normales
diploïdes, les fibroblastes MRC5 de contrôle de cellules
diploïdes cyclantes pour l’étude de centrosomes dans le
cycle cellulaire et les lignées tumorales MCF7 et T47D
de contrôle de cellules tumorales ADN-aneuploïdes. Les
cellules d’intérêt sont entourées au stylo hydrofuge (Dako-
Cytomation). Une fixation par l’acétone à froid (+ 4 ◦C)
pendant dix minutes suivi d’un lavage dans du PBS 5 %
SVF pendant cinq minutes a montré les meilleurs résultats.
Les cellules sont ensuite perméabilisées avec du Triton®
X-100 à 0,1 % pendant cinq minutes à température
ambiante. Un anticorps (Ac) monoclonal de souris anti-␥-
tubuline (1,6 mg/mL) (Sigma) est utilisé pour le marquage
du centrosome. Les lames sont incubées avec du sérum AB
(SAB) humain (Bio West) à 30 % dans du PBS pendant
30 minutes puis avec l’Ac primaire à 1/300 pendant
60 minutes à température ambiante. L’isotype est une
IgG1,non relevante (0,2 mg/mL) (Sigma). Après un
lavage dans du PBS 5 % SVF pendant dix minutes, les lames
sont incubées avec un Ac secondaire de chèvre anti-souris
marqué au FITC à 1/50 (FITC-IgG,F(ab)2[1,4 mg/mL]
[Immunotech]), pendant 60 minutes à l’abri de la lumière.
Pour la contre-coloration des noyaux, un lavage des lames
dans du PBS pendant dix minutes est suivi par une incu-
bation avec du DAPI pendant une minute à l’abri de la
lumière. Les lames sont ensuite montées dans Vectashield®
Mounting Medium (1,5 g/mL) (Vector). L’observation
est faite à l’aide d’un microscope à fluorescence Leica
DM 4000B (Wetzlar, Allemagne) aux grossissements ×10,
×60 et ×100 et utilisant un filtre bleu pour le DAPI
(d’excitation = 359 nm ; d’émission = 461 nm) et un
filtre vert pour le FITC (d’excitation = 490 nm ;
d’émission = 525 nm). Le nombre de points verts (cen-
trosomes) est compté dans chaque cellule sur au moins
100 cellules par lame. Les images sont numérisées à
l’aide d’une caméra refroidie CCD (Cool Snaps cf) et d’un
logiciel d’acquisition d’image Genikon (Alphelys, Plaisir,
France).
Cytométrie en flux
La cytométrie en flux (CMF) est réalisée sur les échan-
tillons tumoraux prélevés par l’anatomopathologiste dans
la région la plus représentative de la tumeur, congelés dans
l’azote liquide et conservésà-80◦C. Après décongélation
et réalisation d’empreintes permettant de confirmer la pré-
sence de cellules tumorales, les prélèvements sont dissociés
mécaniquement et les cellules recueillies sont réparties dans
deux tubes. Dans l’un des tubes, des lymphocytes humains
(2.105lymphocytes pour 106cellules tumorales) normaux
sont rajoutés comme témoins de la population diploïde. Les
cellules sont ensuite incubées avec l’iodure de propidium
(IP), [kit DNA prep, Beckman Coulter]. L’analyse d’un
minimum de 2,104cellules est réalisée en utilisant un cyto-
mètre en flux (Epics Elite®, Beckman Coulter) équipé d’un
laser Argon ((= 488 nm) et d’un filtre rouge (= 635 nm).
Les données sont enregistrées dans un fichier sous forme
de listmod (LMD). Le contenu en ADN et la FPS sont éva-
lués à l’aide du logiciel Wincycle (Phoenix, États-Unis)
conformément aux recommandations et aux conférences
de consensus [4, 22]. Le contenu en ADN définit les cel-
lules ADN-diploïdes ou ADN-aneuploïdes et le calcul de
la FPS les répartit en classes en fonction des terciles selon
les règles de validation établies par les consensus. L’ADN-
ploïdie est définie par l’index d’ADN (ID) qui est le rapport
du canal moyen de fluorescence des cellules tumorales sur
celui des cellules diploïdes de référence : une tumeur dont
toutes les populations cellulaires ont un ID proche d’un
est dite ADN-diploïde alors qu’une tumeur ayant au moins
une population cellulaire avec un ID différent d’un est
dite ADN-aneuploïde. Par ailleurs, pour les tumeurs ADN-
diploïdes, la phase S est dite basse (B) si la FPS inférieure
ou égale à 1,2 %, intermédiaire (I) si la FPS est supérieure
à 1,2 % et inférieure ou égaleà3%,ethaute (H) si la FPS
Copyright © 2017 John Libbey Eurotext. Téléchargé par un robot venant de 88.99.165.207 le 25/05/2017.

184 Ann Biol Clin, vol. 69, n◦2, mars-avril 2011
Article original
est supérieureà3%.Pour les tumeurs ADN-aneuploïdes,
la phase S est dite basse si la FPS inférieure ou égale à
3,85 %, intermédiaire si la FPS est supérieure à 3,85 % et
inférieure ou égale à 8,7 %, et haute si la FPS est supérieure
à 8,7 %.
Analyse statistique
Le calcul des moyennes et des écarts types ainsi que les
histogrammes sont réalisés à l’aide du logiciel Statview®,
Abacus, Berkeley.
Résultats
Immunofluorescence et quantification
du centrosome
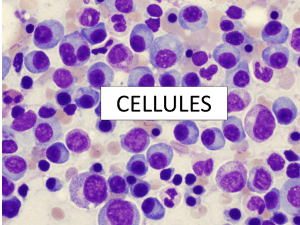
La présence d’un centrosome correspond à un point brillant
vert de fluorescence et toutes les expériences ont été repro-
duites au moins cinq fois (300 lames au total). La majorité
des cellules mésothéliales diploïdes normales (94 %) ont un
seul point fluorescent (centrosome) par cellule (figure 1).
Dans quelques cellules (6 %), deux points fluorescents
sont observés correspondant probablement à des cellules en
cours de duplication. Lorsque les cellules MRC5 diploïdes
cyclantes atteignent 60 % de confluence, elles ont un taux
élevé de mitoses et de cellules en phase S (30 %) permet-
tant d’étudier l’évolution du centrosome au cours du cycle
cellulaire, d’une part, en comptant le nombre de centro-
somes et, d’autre part, en évaluant la distance séparant les
deux points d’une même cellule. Nous avons observé 32 %
de cellules avec un seul point fluorescent et 68 % de cel-
lules avec deux points fluorescents (figure 1). Les cellules
des lignées tumorales MCF7 et T47D ont un, deux, trois
ou quatre points fluorescents (figure 1). Leur nombre est
détaillé dans le tableau 1. Dix prélèvements de carcinome
canalaire infiltrant du sein sont marqués avec l’Ac anti-
␥-tubuline (figure 2). Les résultats de la numération des
centrosomes sont présentés dans le tableau 2.
C
AB
D E
Figure 1. Immunomarquage avec l’anticorps anti-␥-tubuline observé au microscope à fluorescence Leica (×100). A) Cellules mésothéliales
d’ascite. B) Lignée cellulaire de fibroblastes MRC5. C) Cellule de la lignée fibroblastique MRC5 en cycle. D) Lignée cellulaire de cancer
du sein MCF7. E) Lignée cellulaire de cancer du sein T47D. Bleu : noyau coloré par le DAPI ; vert : centrosome immunomarqué par FITC ;
: un point vert ;
: deux points verts.
Tableau 1. Pourcentage de cellules des lignées MCF7 et T47D avec un, deux, trois et quatre centrosome(s).
Cellules avec
1 centrosome (%)
Cellules avec
2 centrosomes (%)
Cellules avec
3 centrosomes (%)
Cellules avec
4 centrosomes (%)
ID
MCF7 24 12 60 4 1,6
T47D 25 52 21 2 1,4
ID : index d’ADN.
Copyright © 2017 John Libbey Eurotext. Téléchargé par un robot venant de 88.99.165.207 le 25/05/2017.

Ann Biol Clin, vol. 69, n◦2, mars-avril 2011 185
Anomalies centrosomique et ADN-ploïdie dans le cancer du sein
BA
CD
Figure 2. Empreintes cellulaires de carcinome canalaire infiltrant du sein. A) Cellules colorées par MGG. B) immunomarquage avec
l’anticorps anti-␥-tubuline observé au microscope à fluorescence Leica (×100) : un centrosome. C) Immunomarquage avec l’anticorps
anti-␥-tubuline observé au microscope à fluorescence Leica (×100) : trois centrosomes. D) Immunomarquage avec l’anticorps anti-␥-
tubuline observé au microscope à fluorescence Leica (×100) : quatre centrosomes. Bleu : noyau coloré par le DAPI ; vert : centrosome
immunomarqué par FITC ;
: un point vert ;
: deux points verts.
Cytométrie en flux
Toutes les cellules mésothéliales sont ADN-diploïdes. Les
fibroblastes MRC5 sont ADN-diploïdes avec une FPS
variant en fonction du taux de confluence cellulaire dans
la boîte de Petri. La FPS est maximale (30) à 60 %
de confluence. Les lignées MCF7 et T47D sont ADN-
aneuploïdes avec ID à 1,6 et 1,4 respectivement. Trois cas
de carcinome canalaire infiltrant sont ADN-diploïdes et les
sept autres cas sont ADN-aneuploïdes, dont trois cas avec
un ID autour de 1,5 (proches triploïdes) et quatre cas avec
un ID autour de 1,8 (proches tétraploïdes). Un seul cas de
carcinome canalaire infiltrant a une FPS basse, deux cas ont
une FPS intermédiaire et cinq cas ont une FPS haute.
Corrélation entre le nombre de centrosomes
et l’ADN-ploïdie
Cellules normales
Dans les cellules diploïdes peu ou non cyclantes (cellules
mésothéliales), le nombre de cellules avec un seul cen-
trosome est maximal (94 %). Dans les cellules diploïdes
cyclantes (fibroblastes MRC5) avec une FPS à 30 %, le
pourcentage de cellules avec un centrosome est de 32 %
et le pourcentage de cellules avec deux centrosomes est
de 68 %. Ces types cellulaires sont de bons témoins de
cellules normales et de cellules diploïdes cyclantes. Ces
résultats correspondent à ce qui est attendu dans les cel-
lules normales cyclantes ou non et sont donc des contrôles
de qualité des expériences.
Lignées tumorales
Un nombre de centrosomes supérieur ou égal à trois
est considéré comme anormal. Dans la lignée MCF7,
100 % des cellules sont ADN-aneuploïdes avec un ID
à 1,6. Le pourcentage de cellules avec un nombre de
centrosomes supérieur ou égal à trois est de 64 % en cor-
rélation avec l’ADN-aneuploïdie. Dans la lignée T47D,
100 % des cellules sont également ADN-aneuploïdes
avec un ID à 1,4. Le pourcentage de cellules avec un
nombre de centrosomes supérieur ou égal à trois est de
23 %.
Carcinomes infiltrants du sein
Trois cas de carcinomes canalaires invasifs du sein sont
ADN-diploïdes. Sept cas de carcinomes canalaires invasifs
du sein sont ADN-aneuploïdes. Le pourcentage de cellules
avec un nombre de centrosomes supérieur ou égal à trois
est légèrement supérieur dans les cas ADN-aneuploïdes
par rapport aux cas ADN-diploïdes (15 ±9,6 % versus
13,3 ±5,7 %). Une analyse détaillée des corrélations est
illustrée par la figure 3.
Copyright © 2017 John Libbey Eurotext. Téléchargé par un robot venant de 88.99.165.207 le 25/05/2017.
 6
6
 7
7
 8
8
 9
9
1
/
9
100%