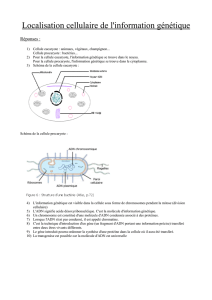
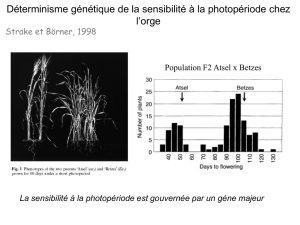
grandeur et servitudes dela génétique inverse - iPubli

S
Bertrand Jordan
Directeur de recherche
au Cnrs
0

1 %),

50 000
10%
*
1
/
3
100%