Syndromes myélodysplasiques avec délétion du bras long du

Focus
Sous la direction de Philippe Rousselot
Syndromes myélodysplasiques
avec délétion du bras long
du chromosome 5
Myelodysplasic syndromes associated with 5q- deletions
Emilie Frisan
1
Franck Viguié
2
François Dreyfus
1
Michaëla Fontenay
1
1
Institut Cochin,
Département d’hématologie,
Université Paris-Descartes,
Inserm U567,
IFR118, CNRS UMR8204,
Hôpital Cochin, Paris
2
Service d’hématologie biologique,
Hôtel-Dieu, Paris
Les délétions interstitielles du bras long du chromosome 5 [del(5q] sont
des anomalies cytogénétiques fréquemment rencontrées au cours des
syndromes myélodysplasiques (SMD) (12 %), des leucémies aiguës
myéloblastiques (LAM) secondaires à un SMD ou des SMD et LAM
secondaires à une chimiothérapie ou à une radiothérapie (40 %). Elles
sont beaucoup moins fréquentes chez les patients porteurs d’une LAM de novo
(5-7 %). Dans les LAM secondaires, elles peuvent être associées à d’autres anoma-
lies cytogénétiques telles que les del(7q) et del(17p) ou à des anomalies complexes
de pronostic défavorable.
Il existe deux entités :
– le syndrome 5q- indolent, caractérisé par une anémie et une dépendance aux
transfusions de globules rouges, la présence de mégacaryocytes hypolobés, un
faible de risque de transformation ;
– les hémopathies myéloïdes (anémies/cytopénies réfractaires avec excès de
blastes ou LAM) avec del(5q) de pronostic défavorable. L’identification des gènes
impliqués dans la physiopathologie de ces deux entités est le but de travaux récents
publiés par les équipes de Todd R. Golub dans Nature [1] et d’A.Thomas Look dans
Nature Medicine [2].
Le syndrome 5q-
Ce syndrome a été défini en 1974 par cytogénétique conventionnelle [3]. Il est
caractérisé par un défaut prédominant de croissance et de maturation de la lignée
érythroïde. La lignée granuleuse est préservée et la dysplasie des mégacaryocytes
n’empêche pas la production des plaquettes. Ce syndrome répond remarquable-
ment bien au traitement par un analogue du thalidomide, le lénalidomide [4].
La région commune de délétion du syndrome 5q- est une région de 1,5 Mb située en
5q32-5q33.1 contenant 40 gènes entre le marqueur D5S413 et le gène GLRA1 [5]
.
Aucune délétion bi-allélique ou mutation n’a été identifiée sur ces gènes. Cette
observation suggère un effet d’haplo-insuffisance, c’est-à-dire que la perte d’une seule
copie est nécessaire et suffisante pour qu’apparaisse le phénotype caractéristique du
syndrome. Parmi ces gènes, 33 sont exprimés dans les progéniteurs CD34+ normaux
et certains (SLC36A1,G3BP,ATOX1,CSF1R,RPS14,PDGFRB,TNIP1,SPARC et
ANAX6) ont une expression nettement diminuée chez les patients [6]
.
L’équipe de
Golub a développé une stratégie systématique d’inhibition de l’expression des
40 gènes par shRNA (short hairpin RNA) introduits par transfert lentiviral dans des
cellules CD34+ normales. Seule la construction ciblant RPS14
reproduit le phénotype
du syndrome 5q- avec un défaut de l’érythropoïèse prédominant sur celui de la
mégacaryopoïèse. L’invalidation partielle du gène RPS14
inhibe l’expression des
Hématologie 2008 ; 14 (2) : 112-5
Hématologie, vol. 14, n° 2, mars-avril 2008
112
doi: 10.1684/hma.2008.0237
Copyright © 2017 John Libbey Eurotext. Téléchargé par un robot venant de 88.99.165.207 le 25/05/2017.

transcrits érythroïdes, un profil transcriptionnel qui correspond à
celui des patients del(5q) répondeurs au lénalidomide [7]
.
L’hypothèse d’un effet d’haplo-insuffisance est confirmée chez
les patients qui ne portent ni mutation ou délétion cryptique
bi-allélique du gène, ni perte d’expression du transcrit, ce
qui exclut la possibilité d’une inactivation bi-allélique de
RPS14. Enfin, la surexpression par vecteur lentiviral de RPS14
dans les progéniteurs CD34+ de patients porteurs d’un syn-
drome 5q- restaure l’expression des transcrits érythroïdes et la
différenciation.
Le gène RPS14 code pour un composant de la sous-unité
ribosomale 40S. La biogenèse des ribosomes requiert 4 ARN
ribosomaux (ARNr), et au moins 80 protéines différentes dont
l’ARN polymérase I et des ribonucléoprotéines nucléolaires.
L’ARNr 18S, composant de la sous-unité 40S est produit à
partir d’un pré-ARNr de 30S. L’invalidation du gène RPS14
chez S. cerevisiae ou l’inhibition de RPS14 par shRNA dans
la lignée érythroïde humaine TF-1 induit une augmentation du
ratio 30S/18S et une apoptose. Chez les patients SMD avec
délétion 5q, l’augmentation du ratio 30S/18S témoignerait
donc d’une inhibition de la biogenèse des ribosomes [1]. Il
est intéressant de faire le parallèle avec les insuffisances
médullaires constitutionnelles prédisposant aux cancers, que
sont l’anémie de Blackfan-Diamond (ABD), la dyskératose
congénitale (DC), l’hypoplasie cartilagineuse (HHC) et le
syndrome de Schwachman-Diamond (SDS). Plusieurs muta-
tions de gènes impliqués dans la maturation des ARNr ont été
décrites. La DC dans sa forme récessive liée à l’X est associée
à une anomalie du gène codant la dyskérine 1 (DKC1),
protéine nucléolaire s’associant aux ribonucléoprotéines. Le
SDS et le HHC sont liés à des mutations de gènes SBDS et
RMRP codant des protéines nucléolaires à activité endonuclé-
asique. Enfin, l’érythroblastopénie constitutionnelle de
Blackfan-Diamond, dont le phénotype ressemble à celui du
syndrome 5q, est associée à des mutations hétérozygotes du
gène RPS19 situé sur le chromosome 19q13.2 dans 25 %
des cas, et plus rarement des gènes RPS24 ou RPS17 [8]. Les
anomalies de biogenèse des ribosomes contribueraient au
phénotype de ces anémies acquises et constitutionnelles.
Pourquoi existe-t-il une atteinte spécificité de la lignée
érythroïde alors que l’expression de RPS14 est ubiquitaire ?
La question n’est pas résolue mais il est possible que les
délétions mono-alléliques ne s’expriment que dans les cellules
à renouvellement rapide telles que les cellules érythroïdes (2
x10
11
nouvelles cellules par jour). Cependant, il n’est pas
exclu que la délétion mono-allélique d’autres gènes de la
région 5q32-5q33.1 puisse contribuer au phénotype du
syndrome 5q-.
Les délétions 5q- associées
aux SMD de haut risque
et aux LAM secondaires
Les anomalies 5q associées aux SMD avec excès de blastes
ou aux LAM secondaires à une chimiothérapie concernent
une région centrée sur la bande 5q31.1 dont les bornes sont
très variables. Cette région s’étend sur environ 3.7 Mb entre
le gène codant IL9 du côté centromérique et le gène codant
UBE2D2 du côté télomérique [9-12]. Environ 28 gènes ont
été identifiés dont IL9, TGFB1, TRPC7, CDC23, EGR1,
HSPA9B, CTNNA1 et UBE2D2 parmi lesquels de possibles
gènes candidats suppresseurs de tumeurs.
Plusieurs gènes de la région de délétion ont été étudiés.
EGR1 qui code pour un facteur de transcription à doigt de
zinc pourrait être un gène suppresseur de tumeur. Les souris
EGR1
+/-
développent des cancers hématopoïétiques, ce qui
suggère un effet d’haplo-insuffisance [13]. HSPA9B/
mortalin/mthsp70/GRP78 code pour une forme mitochon-
driale des protéines de choc thermique de 70 kDa très
conservée dans l’évolution et impliquée dans le transport, le
repliement et l’assemblage des protéines dans la matrice
mitochondriale. Chez le poisson zèbre, le mutant crimeson-
less (crs) de HSPA9B présente une dysplasie multilignée des
progéniteurs hématopoïétiques liée à un défaut de matura-
tion et à un excès d’apoptose [14]. De même, les mutations
perte de fonction de la HSPA9B chez la levure et chez
C. elegans induisent des anomalies morphologiques de la
mitochondrie et la mort cellulaire. En revanche, il n’a pas été
rapporté d’anomalies de l’hématopoïèse chez les souris
invalidées pour le gène UBE2D2 qui code pour une E2
ubiquitine ligase.
Selon l’équipe de Look, HSPA9B et UBE2D2 ne sont pas
inactivés dans les cellules CD34+CD38-lin
-
de patients
porteurs d’une hémopathie maligne à haut risque avec
délétion mono-allélique 5q- confirmée par FISH. En revanche,
le gène CTNNA1 qui code pour l’a-caténine a une expres-
sion diminuée de plus de 50 % dans les progéniteurs de type
LTC-IC (long term colony-iniating cell) CD34+CD38-
CD123+lin
-
des patients avec del(5q), ce qui suggère que
l’allèle résiduel est fonctionnellement inactif [2]. Dans la
lignée HL-60 del(5q), l’expression de CTNNA1 est en effet
éteinte par modifications épigénétiques (hyperméthylation
de l’ADN et déacétylation des histones) du promoteur. La
décitabine (agent déméthylant l’ADN) ou la trichostatine A
(inhibiteur d’histone déacétylase) restaure l’expression de
CTNNA1 et induit l’apoptose de cette lignée. Chez 2/3
des patients avec del(5q) étudiés, la perte d’expression
de CTNNA1 est due à une hyperméthylation de l’ADN d’une
région promotrice proche du site d’initiation de la trans-
cription. Chez un tiers des patients, l’expression de CTNNA1
persiste, ce qui témoigne soit d’un syndrome débutant
sous l’effet d’une haplo-insuffisance, soit de l’implication
d’un autre gène. CTNNA1 est donc un potentiel gène sup-
presseur de tumeur qui peut être inactivé par délétion d’un
allèle et par extinction épigénétique de la transcription de
l’autre allèle. La perte d’expression de la protéine a-caténine
perturberait l’adhésion des cellules souches clonales dans la
niche hématopoïétique favorisant l’expansion du clone
malin.
Hématologie, vol. 14, n° 2, mars-avril 2008
113
Copyright © 2017 John Libbey Eurotext. Téléchargé par un robot venant de 88.99.165.207 le 25/05/2017.

La réponse au traitement
par lénalidomide
Les hémopathies malignes avec del(5q) sont efficacement
traitées par le lénalidomide. Dans la description initiale de la
réponse au traitement dans un essai clinique de phase II,
45 % des patients ont une réponse cytogénétique complète
après 6 cycles de traitement, 76 % ont une réduction des
besoins transfusionnels et 67 % sont devenus indépendants
des transfusions. Cependant, des neutropénies et/ou throm-
bopénies modérées ou sévères sont observées chez plus de
40 % des patients [3]. La réponse érythroïde au lénalidomide
des patients porteurs d’une délétion 5q associée à d’autres
anomalies cytogénétiques est d’environ 20 % (dont 15 % de
réponses complètes) dans une série de patients évalués par le
Groupe francophone des myélodysplasies. L’équipe de Raza
a entrepris de rechercher un profil transcriptionnel prédictif
de la réponse au traitement. Les résultats publiés dans PLoS
Medicine en février 2008 montrent que les patients répon-
deurs, qu’ils soient porteurs ou non d’une délétion 5q, ont
une signature génique caractéristique d’un défaut de diffé-
renciation érythroïde HBA2, HBB, SPTA1, GYPA, GYPB,
ALAS2, KLF1 [7]. L’expression de 4 gènes (SPARC, VSIG4,
PP1C, TPBG) augmente dans les cellules érythroïdes norma-
les et pathologiques de syndrome 5q-, cultivées en présence
de lénalidomide [15]. SPARC, identifié comme gène suppres-
Pellagatti et al., 2007
Ebert et al., 2008
IL9
TGFB1
MADH5
TRPC7
SPOCK
HRNPA0
TTID
WNTBD
CDC23
GFRA3
CDC25C
FAM53C
EGR1
ETF1
HSPA9B
CTNNA1
MATR3
UBE2D2
CSF1R
PDGFRB
TCOF1
CD74
RPS14
NDST1
TNIP1
SPARC
ATOX1
GLRA1
5q
q11.2
q23
q13
q14
q15
q21
q22
q12
q31
q32
q33.1
q34
q35
Liu XT et al., 2007
Joslin JM et al., 2007
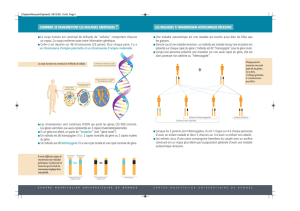
Figure 1. Représentation schématique des zones communes de délétion du bras long du chromosome 5 dans les syndromes 5q- et les
hémopathies myéloïdes à haut risque de transformation aiguë. Les gènes d’intérêt et les références bibliographiques sont indiqués par
une flèche.
Hématologie, vol. 14, n° 2, mars-avril 2008
114
Copyright © 2017 John Libbey Eurotext. Téléchargé par un robot venant de 88.99.165.207 le 25/05/2017.

seur de tumeurs dans plusieurs cancers est situé en 5q33.1.
SPARC a des fonctions antiprolifératives, antiadhésives et
pourrait être un médiateur des effets antiangiogéniques
connus du médicament. La ré-expression de SPARC par le
lénalidomide témoignerait de l’extinction du clone pathologi-
que et de la sélection positive du clone normal. L’analyse
cytogénétique confirme le plus souvent l’éradication du clone
5q- sous traitement et parfois l’émergence d’autres anomalies
cytogénétiques. A ce jour, le mécanisme d’action du lénalido-
mide reste cependant imprécis.
Conclusion
Les travaux récents apportent des arguments forts pour la
contribution d’un mécanisme d’haplo-insuffisance à l’émer-
gence du syndrome 5q- et pour la nécessité d’une inactiva-
tion fonctionnelle d’un gène suppresseur de tumeur pour le
développement d’une hémopathie myéloïde à haut risque de
transformation en leucémie aiguë myéloblastique (figure 1).
Bien qu’il y ait une région commune de délétion interstitielle,
les points de cassure des délétions 5q sont éminemment
variables d’un cas à l’autre et il n’y a pas de microdélétions
infra-chromosomiques. Il est donc peu probable que ces
maladies résultent de l’atteinte d’un seul gène, mais plutôt de
l’intrication de plusieurs mécanismes complexes. ■
RÉFÉRENCES
1.Ebert BL, Pretz J, Bosco J, et al. Identification of RPS14 as a
5q- syndrome gene by RNA interference screen. Nature 2008 ;
451 : 335-40.
2.Liu XT, Becker MW, Jelinek J, et al. Chromosome 5q deletion
and epigenetic suppression of the gene encoding a-catenin
(CTNNA1) in myeloid cell transformation. Nat Med 2007 ;
13 : 78-83.
3.Van den Berghe H, Cassiman JJ, David G, et al. Distinct
haematological disorder with deletion of long arm of no. 5
chromosome. Nature 1974 ; 251 : 437.
4.List A, Dewald G, Bennett J, et al. Lenalidomide in the myelo-
dysplastic syndromes with chromosome 5q deletion. N Engl J
Med 2006 ; 355 : 1456-65.
5.Boultwood J, Fidler C, Strickson AJ, et al. Narrowing and
genomic annotation of the common deleted region of the 5q-
syndrome. Blood 2002 ; 99 : 4638-41.
6.Lehmann S, O’Kelly J, Raynaud S, Funk SE, Sage EH, Koef-
fler HP. Common deleted genes in the 5q- syndrome : thrombo-
cytopenia and reduced erythroid colony formation in SPARC null
mice. Leukemia 2007 ; 21 : 1931-6.
7.Ebert BL, Galili N, Tamayo P, et al. An erythroid differentia-
tion signature predicts response to lenalidomide in myelodys-
plastic syndrome. PLoS Med 2008 ; 5 : e35 ; (sous presse).
8.Liu JM, Ellis SR. Ribosomes and marrow failure : coincidental
association or molecular paradigm? Blood 2006 ; 107 :
4583-8.
9.Fairman J, Chumakiv I, Chinault AC, Nowell OC, Nagara-
jan L. Physical mapping of the minimal region of loss in 5q-
chromosome. Proc Natl Acad Sci USA 1995 ; 92 : 7406-10.
10.Zhao N, Stoffel A, Wang PW, et al. Molecular delineation
of the smallest common deleted region of chromosome 5 in
malignant myeloid diseases. Proc Natl Acad Sci USA 1997 ;
94 : 6948-53.
11.Horrigan SK, Arbieva ZH, Xie HY, et al. Delineation of a
minimal interval and identification of 9 candidates for a tumor
suppressor gene in malignant myeloid disorders on 5q31.
Blood 2000 ; 95 : 2372-7.
12.Lai F, Godley LA, Joslin J, et al. Transcript map and compa-
rative analysis of the 1.5 Mb commonly deleted segment of the
human 5q31 in malignant myeloid leukaemia with del(5q).
Genomics 2001 ; 71 : 235-45.
13.Joslin JM, Fernald AA, Tennant TR, et al. Haploinsufficiency
of EGR1, a candidate gene in the del(5q), leads to the develop-
ment of myeloid disorders. Blood 2007 ; 110 : 719-24.
14.Craven SE, French D, Ye W, de Sauvage F, Rosenthal A.
Loss of Hspa9B in zebafish recapitulated the ineffective hemato-
poiesis of the myelodysplastic syndrome. Blood 2005 ; 105 :
3528-34.
15.Pelagatti A, Jädersten M, Forsblom A-M, et al. Lenalido-
mide inhibits the malignant clone and up-regulats the SPARC
gene mapping to the common deleted region in 5q- syndrome
patients. Proc Natl Acad Sci USA 2007 ; 104 : 11406-11.
Hématologie, vol. 14, n° 2, mars-avril 2008
115
Copyright © 2017 John Libbey Eurotext. Téléchargé par un robot venant de 88.99.165.207 le 25/05/2017.
1
/
4
100%