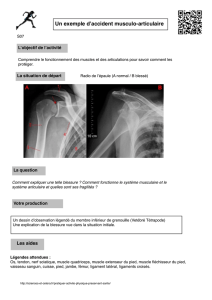
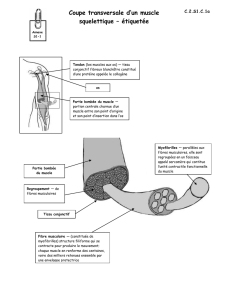
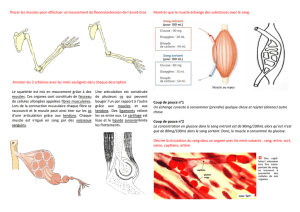
I Myogenèse du muscle squelettique

Université Paris Descartes - UFR Biomédicale
Thèse
Pour obtenir le grade de docteur de l’Université Paris Descartes
Ecole Doctorale B3MI
« Biochimie, Biothérapie, Biologie Moléculaire et Infectiologie »
P
résentée et soutenue publiquement le 28 juin 2013 par
Nissrine DAOU
Étude de l’intéraction
MyoD/NFATc2 au cours de la
myogenèse chez la souris
Présidente : Dr. Suzie LEFEBVRE
Rapporteur : Pr. Jamileh MOVASSAT
Rapporteur : Pr. Michèle BEAUDRY
Encadrante : Dr. Anne-Sophie ARMAND
Directeur de thèse : Pr. Christophe CHANOINE
Jury

2
À ma mine d’or,
À Ka3boul,

3

4
TABLE DES MATIERES
INTRODUCTION .............................................................................................................................................. 12
I
MYOGENESE DU MUSCLE SQUELETTIQUE ............................................................................................. 14
I.1
L
E SYSTEME MUSCULAIRE SQUELETTIQUE
.......................................................................................................... 14
I.1.1
Les différents tissus musculaires ................................................................................................... 14
I.1.2
Organisation du muscle squelettique ............................................................................................ 16
I.1.2.1
Structure du muscle squelettique ............................................................................................................. 16
I.1.2.2
Caractéristiques fonctionnelles du muscle squelettique .......................................................................... 19
I.1.2.3
Diversité des fibres musculaires ............................................................................................................... 20
I.1.2.3.1
Histoire de la fibre musculaire ....................................................................................................... 20
I.1.2.3.2
Les différents types de fibres ......................................................................................................... 21
I.1.3
Myogenèse primaire et secondaire ............................................................................................... 23
I.2
C
ONSTRUCTION DU MUSCLE SQUELETTIQUE
...................................................................................................... 24
I.2.1
La somitogenèse ............................................................................................................................ 25
I.2.2
Devenir des cellules somitiques et acquisition de l’identité myogénique ................................... 27
I.2.3
Les facteurs Pax3 et Pax7 ............................................................................................................. 30
I.2.4
Détachement et migration des progéniteurs musculaires impliqués dans la formation des muscles
des membres................................................................................................................................................ 31
I.2.5
Progression myogénique ............................................................................................................... 32
I.2.5.1
Expression des facteurs myogéniques au cours de l’ontogenèse musculaire ........................................ 32
I.2.5.2
Effets de l’invalidation des gènes codant pour les facteurs myogéniques ............................................... 34
I.2.5.3
Prolifération des myoblastes ................................................................................................................... 37
I.2.5.4
Arrêt de la prolifération et levée de l'inhibition de la différenciation : la sortie du cycle cellulaire et
l’activation de la différenciation ............................................................................................................................... 38
I.2.5.5
Activation du programme myogénique .................................................................................................... 39
I.2.5.6
Fusion des myoblastes en myotubes ........................................................................................................ 40
I.2.5.6.1
Les interactions cellulaires ............................................................................................................. 40
I.2.5.6.2
Le Remodelage du cytosquelette ................................................................................................... 42
I.2.6
Expression des facteurs Myf5 et MyoD et la régénération musculaire ......................................... 43
II
LA VOIE DE SIGNALISATION CALCINEURINE/ NFAT ................................................................................ 46
II.1
L
A CALCINEURINE
......................................................................................................................................... 46
II.1.1
Les propriétés de la calcineurine ................................................................................................... 46
II.1.2
Structure de la calcineurine ........................................................................................................... 46
II.1.3
Distribution tissulaire de la calcineurine ....................................................................................... 48
II.2
L
A FAMILLE
NFAT ........................................................................................................................................ 48
II.2.1
Structure des protéines NFATs ...................................................................................................... 49
II.2.2
Liaison de NFATc2 à l’ADN ............................................................................................................ 49
II.2.3
Activation des protéines NFATs ..................................................................................................... 51
II.3
I
NHIBITION DE LA VOIE CALCINEURINE
/
NFAT ................................................................................................... 52
II.4
L
A VOIE CALCINEURINE
/NFAT
ET LE DEVELOPPEMENT
......................................................................................... 54

5
II.4.1
Rôles des protéines NFAT dans le développement de différents systèmes ................................... 54
II.4.2
Rôle de la voie calcineurine dans le développement du muscle squelettique ............................... 55
II.4.3
Rôles du facteur NFATc3 dans le développement musculaire ....................................................... 55
INTRODUCTION AU TRAVAIL .......................................................................................................................... 57
RÉSULTATS ..................................................................................................................................................... 63
I
ANALYSE DES PROFILS D'EXPRESSION GENIQUE DES FACTEURS MYOGENIQUES ET DES FACTEURS NFATS
PAR HYBRIDATION IN SITU AU COURS DU DEVELOPPEMENT EMBRYONNAIRE CHEZ LA SOURIS ................... 65
I.1
P
RINCIPE DE L
’
HYBRIDATION IN SITU
(HIS)
SUR EMBRYON ENTIER
.......................................................................... 65
I.1.1
Conditions de manipulation : ........................................................................................................ 66
I.1.2
Le choix des sondes : ..................................................................................................................... 67
I.1.3
Étapes de l’hybridation in situ ....................................................................................................... 70
I.2
E
TUDE DES DOMAINES D
’
EXPRESSION DES FACTEURS MYOGENIQUES ET DES FACTEURS
NFAT
S PAR HYBRIDATION IN SITU SUR
EMBRYON MURIN IN TOTO
. ...................................................................................................................................... 71
I.2.1
Domaine d’expression des facteurs MyoD et NFATc2 .................................................................. 71
I.2.2
Expression des autres facteurs myogéniques et des autres membres de la famille NFAT ........... 72
I.3
C
ONCLUSION
.............................................................................................................................................. 74
II
NFATC2 REGULE SPECIFIQUEMENT L’EXPRESSION DE LA CHAINE LOURDE NEONATALE DE MYOSINE EN
COOPERATION AVEC MYOD AU COURS DE LA MYOGENESE CHEZ LA SOURIS (ARTICLE)................................. 74
II.1
I
NTRODUCTION
............................................................................................................................................ 74
II.2
A
RTICLE
................................................................................................................................................ 76
II.3
C
ONCLUSION
............................................................................................................................................ 116
III
L’ABSENCE D'EXPRESSION DE MYOD ET DE NFATC2 SE CARACTERISE PAR UNE APLASIE MUSCULAIRE AU
COURS DE LA MYOGENESE SECONDAIRE ...................................................................................................... 117
III.1
A
NALYSE HISTOLOGIQUE ET IMMUNOFLUORESCENCE
.................................................................................... 117
III.2
É
TUDE DU PHENOTYPE MUSCULAIRE DES EMBRYONS DOUBLE MUTANTS MYOD
(-/-) ;
NFATC
2
(-/-) ...................... 118
III.3
C
ONCLUSION
........................................................................................................................................ 119
CONCLUSION GÉNÉRALE .............................................................................................................................. 122
BIBLIOGRAPHIE ............................................................................................................................................ 128
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
 46
46
 47
47
 48
48
 49
49
 50
50
 51
51
 52
52
 53
53
 54
54
 55
55
 56
56
 57
57
 58
58
 59
59
 60
60
 61
61
 62
62
 63
63
 64
64
 65
65
 66
66
 67
67
 68
68
 69
69
 70
70
 71
71
 72
72
 73
73
 74
74
 75
75
 76
76
 77
77
 78
78
 79
79
 80
80
 81
81
 82
82
 83
83
 84
84
 85
85
 86
86
 87
87
 88
88
 89
89
 90
90
 91
91
 92
92
 93
93
 94
94
 95
95
 96
96
 97
97
 98
98
 99
99
 100
100
 101
101
 102
102
 103
103
 104
104
 105
105
 106
106
 107
107
 108
108
 109
109
 110
110
 111
111
 112
112
 113
113
 114
114
 115
115
 116
116
 117
117
 118
118
 119
119
 120
120
 121
121
 122
122
 123
123
 124
124
 125
125
 126
126
 127
127
 128
128
 129
129
 130
130
 131
131
 132
132
 133
133
 134
134
 135
135
 136
136
 137
137
 138
138
 139
139
 140
140
 141
141
 142
142
 143
143
 144
144
 145
145
 146
146
 147
147
1
/
147
100%