EDITE de Paris (ED130) -

Apport du machine learning dans la stratification
du risque cardiovasculaire
Sujet proposé par Nadjia KACHENOURA [email protected]
École doctorale: EDITE de Paris (ED130)
Domaine: Sciences pour l'ingénieur
Projet
Les maladies cardiovasculaires représentent la première cause de morbi-mortalité dans les sociétés
industrialisées. La compréhension de la physiopathologie sous-jacente inclut une caractérisation précise des
phénotypes cardiaques via des biomarqueurs d?une grande sensibilité, altérés dès le stade pré-clinique (avant
apparition de symptômes). Il existe de nombreux biomarqueurs instantanés ou intégratifs témoignant de
l?atteinte des organes cibles, qui sont issus d?imagerie multimodale ou de dosages biologiques. A elle seule,
l?imagerie génère aujourd?hui plus de 90% de la masse totale de données cliniques. A titre d?exemple, à
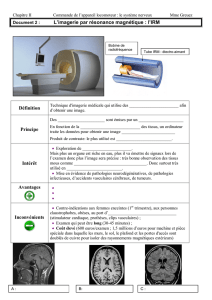
l?issue d?un examen d?IRM cardiaque, une imagerie complète du cœur et des grosses artères (aorte) peut
fournir la fonction et la composition des tissus, ainsi que l?hémodynamique du sang circulant. Au décours
d?une IRM cardiaque, nous pouvons grâce à nos méthodologies (traitements d?images automatisés mis au
point au LIB- cf références https://www.lib.upmc.fr/?page_id=352&equipe_id=3) extraire divers
biomarqueurs révélateurs des atteintes cardiaques et artérielles. Pour affiner le diagnostic, les médecins
élaborent un algorithme multiparamétrique intégrant mentalement l?ensemble des données IRM et cliniques.
Cet algorithme reste mental et non systématisé donc subjectif et dépendant de la sensibilité du médecin. Dans
ce contexte, une collaboration avec S Mesmoudi de l?Equipex Martice et de l?Institut des Systèmes
Complexes, J D Zucker de UMMISCO (UPMC/IRD), A Redheuil et P Cluzel du CHU Pitié-Salpêtrière nous
permettra, grâce aux approches d?apprentissage profond (Deep Learning) de faire face à la multiplication des
images et des biomarqueurs quantitatifs. En effet, nous proposons de mettre en place un modèle intégratif de
ces biomarqueurs et un apprentissage sur images pour une caractérisation individualisée des patients. De plus,
une fouille par text-mining de la bibliographie (BiblioGraph) nous permettra de confirmer l?originalité de nos
résultats vis-à-vis de la littérature existante.
Enjeux
1.Sélectionner des groupes de patients avec différentes pathologies cardiaques et métaboliques et des sujets
sains pour lesquels nous disposons d?une IRM cardiaque avec l?ensemble des biomarqueurs mis au point au
LIB. Puis mettre en place un système intelligent ayant pour objective une classification basée sur des réseaux
de neurones pour démontrer la pertinence de nos indices, leur redondance et associations ainsi que leur
capacité à discriminer nos patients. Ainsi, nous proposons d'utiliser une technique d'apprentissage
automatique de grande qualité qui est l'apprentissage profond afin de construire un modèle d'apprentissage
non supervisé ou semi-supervisé et extraire des caractéristiques hiérarchiques. Un graphique profond avec des
couches de traitement multiples, composés de transformations linéaires et non-linéaires multiples sera utilisé
pour réaliser un haut niveau de représentation de nos données.
2.Vérifier et démontrer la pertinence clinique et l?originalité de nos résultats. La plate-forme d'exploration de
texte mise au point par S Mesmoudi et coll et capable d'analyser la littérature pour rechercher des réseaux
existants entre biomarqueurs sera utilisée Il s?agit de générer les liens dans la littérature actuelle en utilisant
les caractéristiques majeures obtenues dans le projet et de déterminer la nouveauté et la pertinence potentielle
des nouvelles relations identifiées.
3.Focaliser sur les techniques d?apprentissage et de reconnaissance de formes afin d?identifier les
manifestations des diverses pathologies sur nos images d?IRM. Cette seconde étape sera menée dans un
premier temps sur des images statiques, puis sur les images dynamiques et enfin sur les images dynamiques
de vélocimétrie (mouvement des parois + vitesse de l?écoulement). On visera dans un premier temps
$LOGOIMAGE 1/2

l?extraction de nouveaux indices inaccessibles aux méthodes de traitement d?images conventionnelles. Pour
ensuite utiliser la nouvelle représentation des images IRM dans une classification non supervisée ou
semi-supervisée des patients.
Remarques additionnelles
Ce type de modèle multi-paramétrique unifié n?existe pas actuellement. Le développement actuel des
Systèmes Complexes facilite la mobilisation transdisciplinaire des chercheurs autour de deux axes
complémentaires. D?une part, 1) celui des méthodes, qui peuvent être transférées entre différents systèmes
multi-échelles telles que celles mises au point par l?équipe de JD Zucker afin d?intégrer les données
biologiques , et d?autre part, 2) celui de l?intégration des connaissances multi-échelles pour chaque grand
système (du biomarqueur à la décision thérapeutique). Ce modèle élaboré sur des données d?imagerie servira
de base pour faire émerger des modèles plus complets intégrant d?autres données cliniques à l?imagerie. En
effet, un modèle exhaustif décrivant la dynamique des effets de l?âge sur les artères relierait toutes les échelles
du vivant (gènes, cellules, tissus, organes, organisme, société, pharmacologie et environnement).
Pour plus de détails sur l'EDITE, consultez le site http://edite-de-paris.fr/. D'autres propositions de thèse sont
aussi présentes sur ce site.
EDITE de Paris (ED130) -- Proposition de thèse $LOGOIMAGE
$LOGOIMAGE 2/2
1
/
2
100%