C CAILLIOT [Mode de compatibilité]

Caractérisation
tumorale
L’expérience d’ R&D Unicancer
Christian Cailliot ,
Directeur de la recherche et du développement , R&D Unicancer

2
La première structure nationale académique
de recherche clinique en cancérologie en France
Appuyée sur 20 centres de lutte contre le cancer
Soins /Recherche/Enseignement
+ de 70 personnes (projection : > 100 fin 2014)
165 centres investigateurs (CHU/CH, Privés, CLCC, Internationaux)
61 essais en cours
35 essais cliniques ouverts aux inclusions
4438 patients inclus en 2013 ( projection : > 5000 en 2014)
~ 12 nouveaux essais cliniques par an
Un axe majeur : le développement de la médecine « personnalisée »
~ Toutes pathologies (Tumeurs Solides ) Sein +++
R&D UNICANCER c’est :

3
Caractérisation tumorale :
Définition :
Clinique taille, localisation, TNM, extension, imagerie …
Histologique
Grade SBR, récepteurs hormonaux …
Biologique anomalies moléculaires, génomique …
Identification des caractéristiques propres de la tumeur.
Meilleure connaissance de la biologie des cellules cancéreuses. Ce qui
a permis de découvrir des biomarqueurs spécifiques et des « cibles »
et d'y répondre par des traitements dits « ciblés ».
Objectif : Offrir une thérapeutique spécifique ciblée et adaptée à
chaque patient et à chaque tumeur = Médecine « personnalisée »

4
Cancérogénèse :
Schématiquement ,deux grands types d’événements différents
Activation d’un Oncogène Inhibition d’un « Anti-Oncogène »
Caractérisation tumorale = Identifier ces anomalies au sein de la cellule

1
2
3
4
56
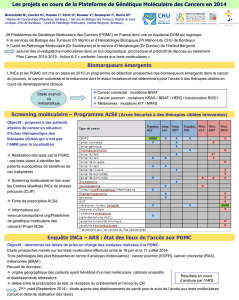
Les thérapies ciblées imposent d’ identifier
(Caractériser) les cibles cellulaires
Récepteurs
Voies
intracellulaires
Noyau de la
cellule et ADN
Une cellule
Thérapies
ciblées
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
1
/
27
100%