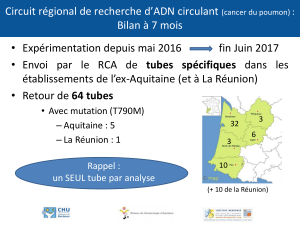
Etude du statut mutationnel EGFR dans l`ADN circulant plasmatique

UNIVERSITE DE NANTES
FACULTE DE MEDECINE
Année 2014 N°
THESE
pour le
DIPLOME D’ETAT DE DOCTEUR EN MEDECINE
D’ETUDES SPECIALISEES DE BIOLOGIE MEDICALE
par
Mehdi Sakka
Né le 04/10/1984 à Hammamet (Tunisie)
Présentée et soutenue publiquement le 23 octobre 2014
Etude du statut mutationnel EGFR dans l’ADN
circulant plasmatique chez douze patients ayant un
cancer pulmonaire non à petites cellules traités par
inhibiteur de tyrosine kinase
Président : Monsieur le Professeur Jean-Marie BARD
Directeur de thèse : Monsieur le Professeur Marc DENIS
Membres du jury : Madame le Docteur Acya BIZIEUX
Monsieur le Professeur Stéphane BEZIEAU
152

1
Table des matières
Remerciements ....................................................................................................................................... 3
Liste des figures ....................................................................................................................................... 5
Liste des tableaux .................................................................................................................................... 6
Liste des abréviations .............................................................................................................................. 8
Introduction ............................................................................................................................................. 9
Première partie : Revue bibliographique .............................................................................................. 10
I. Cancer pulmonaire en france ........................................................................................................ 10
1. Epidémiologie ............................................................................................................................ 10
2. Types histologiques ................................................................................................................... 11
3. Facteurs de risques du cancer pulmonaire ............................................................................... 12
4. Classification TNM ..................................................................................................................... 12
5. Critères d’évaluation RECIST ..................................................................................................... 14
II. EGFR : découverte, intérêt, type mutation biopsie ....................................................................... 15
1. EGFR : Rôle physiologique ......................................................................................................... 15
2. EGFR : rôle oncogénique ........................................................................................................... 16
3. Inhibiteurs de Tyrosine Kinase : erlotinib et gefitinib ............................................................... 17
4. TKI : Essai de phase III et AMM ................................................................................................. 19
5. Effets secondaires du gefitinib et de l’erlotinib ........................................................................ 22
6. AMM en France ......................................................................................................................... 23
7. Inhibiteur de deuxième génération ........................................................................................... 23
8. Plan cancer et plateformes de biologie moléculaire ................................................................. 23
9. Techniques de recherche des mutations EGFR dans le tissu biopsié ........................................ 24
10. Mutation de résistance ......................................................................................................... 24
11. Inhibiteur de 3ème génération ................................................................................................ 27
III. Intérêt de la quantification des ADN circulants dans le cancer pulmonaire non à petites
cellules ................................................................................................................................................... 28
1. Histoire de l’ADN circulant ........................................................................................................ 28
2. ADN circulant et aspect quantitatif : évaluation pronostique .................................................. 29
IV. Intérêt de la recherche d’anomalie d’EGFR dans l’ADN circulant ............................................. 34
1. Avantages par rapport aux biopsies .......................................................................................... 34
2. Intérêt en cancérologie de la recherche d’anomalie dans l’ADN circulant ............................... 36
3. Techniques utilisées pour la recherche d’anomalies d’EGFR .................................................... 37
4. Intérêt pronostique ................................................................................................................... 39
5. Intérêt dans le suivi du traitement ............................................................................................ 39

2
Deuxième partie : travail réalisé ........................................................................................................... 41
I. Matériels et méthodes .................................................................................................................. 41
1. Déroulement de l’étude ............................................................................................................ 41
2. Critères d’inclusions .................................................................................................................. 41
3. Critères d’exclusions .................................................................................................................. 41
4. Recherche de mutations activatrice de l’EGFR : dans le tissu cancéreux ................................. 41
5. Recherche de mutations activatrice de l’EGFR : dans le plasma ............................................... 44
6. Méthodologie ............................................................................................................................ 45
II. Résultats ........................................................................................................................................ 46
1. Caractéristiques des patients .................................................................................................... 46
2. Altérations du gène EGFR dans l’ADN plasmatique et réponse initiale au traitement ............. 47
3. Apparition de la mutation de résistance T790M ....................................................................... 49
4. Suivi longitudinal des patients pendant le traitement .............................................................. 49
III. Discussion .................................................................................................................................. 56
Bibliographie.......................................................................................................................................... 58

3
Remerciements
Je tiens à remercier :
Monsieur le Professeur Jean-Marie BARD
Pour avoir accepté de présider ce jury,
Veuillez trouver ici l’expression de ma reconnaissance.
Monsieur le Professeur Marc DENIS,
Pour m’avoir confié ce travail passionnant,
Pour avoir été patient et m’avoir encadré,
Veuillez trouver ici le témoignage de ma profonde gratitude.
Madame le Docteur Acya BIZIEUX
Pour sa participation dans ce projet,
Pour m’avoir reçu plusieurs fois à la Roche sur Yon,
Veuillez trouver ici l’assurance de mes sincères remerciements.
Monsieur le Professeur Stéphane BEZIEAU
Pour l’honneur que vous me faites de juger ce travail,
Je vous témoigne ma très vive reconnaissance.
Le laboratoire de biochimie et à la plateforme de génétique moléculaire des cancers,
A Audrey Vallée pour tout son travail et son encadrement,
Aux techniciens pour leur aide.

4
A ma famille à qui je dédie cette thèse
A mes parents et à ma sœur,
Pour leur soutien et leurs encouragements,
A Julie,
Qui m’a encouragé dans les moments difficiles et m’a aidé dans la relecture,
Tu es toujours là quand j’ai besoin de toi.
A mes amis
Yassine, Ellie, Kamou, Shadi, nous avons fait un long parcours depuis la Tunisie,
Guillaume, nous arrivons enfin à voir la fin,
A tous mes co-internes nantais pour ces années passées à vos côtés,
A mes co-internes brestois pour leur accueil chaleureux.
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
 46
46
 47
47
 48
48
 49
49
 50
50
 51
51
 52
52
 53
53
 54
54
 55
55
 56
56
 57
57
 58
58
 59
59
 60
60
 61
61
 62
62
 63
63
 64
64
 65
65
 66
66
1
/
66
100%