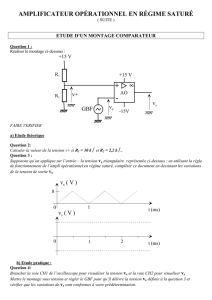
anagene

ANAGENE
(Analyse de séquences nucléiques et protéiques)
Anagène est un logiciel d’analyse de séquences nucléiques et protéiques. Il comporte une banque de données et
différents outils d’analyse et de traitement de ces données.
Anagène est constitué d’une fenêtre-mère présentant sous la barre de titre une barre de menus et une barre d’outils.
Le mode de déplacement et de sélection pilotés à l’aide de la souris ou du clavier obéissent aux règles adoptées pour
Windows. Ainsi un clic pointé sur un libellé de la barre des menus offre le choix entre les commandes disponibles.
Lorsque le pointeur de la souris est maintenu quelques instants en place sur l’un des objets de l’écran, tels par
exemple un bouton de la barre d’outils, une bulle d’aide apparaît et rappelle la fonction qui lui est associée.
Ainsi les fonctions d’Anagène sont accessibles soit au travers les différentes options de la barre de menu, soit
directement pour la plupart par l’intermédiaire des boutons de la barre d’outils.
Plan :
I - EXPRESSION DE L'INFORMATION GENETIQUE
1 - Approche de la transcription
2. Approche de la traduction
3 - Approche du code génétique
II- LES RELATIONS GENOTYPE - PHENOTYPE
1. Les phénotypes relatifs aux hémoglobinopathies
1.1 - Le phénotype drépanocytaire
1.2 - Les phénotypes thalassémiques
1.3 - Le phénotype " groupe sanguin ABO "
III - POLYMORPHISME DES GENES
1. Structure des gènes et des protéines HLA de classe I
2. Les allèles des gènes HLA de classe I
3. Recherche des différences entre les allèles d’un gène HLA
CONCLUSIONS I - EXPRESSION DE L'INFORMATION GENETIQUE
1 - Approche de la transcription
1.1 - Visualisation de l’ADN double brin et de l’ARNm
Cliquer sur le bouton Thèmes d’étude de la barre d’outils (ou sélectionner la commande Thèmes d’étude dans le menu
Fichier).
Ouvrir successivement le menu Expression de l’information génétique et le sous-menu Globine alpha.
Sélectionner Gène et ARNm codant et valider le choix en cliquant sur OK.
Les séquences nucléiques de l’ADN (Alpha brin 1 et Alpha brin 2) et de l’ARNm sont affichées dans la
fenêtre Affichage des séquences et peuvent être parcourues de la 1ère à la 429 ème base en utilisant la barre
de défilement horizontal.
1.2 - Comparaison de chaque brin d’ADN avec l’ARNm
Sélectionner Alpha ARNm cod et Alpha brin 1 en cliquant sur les boutons de sélection correspondant à chaque
séquence.

Cliquer sur le bouton Comparer les séquences de la barre d’outils (ou sélectionner l’option Comparer les séquences dans le
menu Traiter).
Valider l’option Comparaison simple et confirmer en cliquant sur OK.
La nature du traitement effectué et les deux séquences comparées s’affichent dans la fenêtre
Comparaison simple. Elles peuvent être parcourues en utilisant la barre de défilement horizontal. Le tiret
indique l’identité des bases par rapport à celles de la première séquence qui sert de référence.
En procédant de la même façon, comparer Alpha brin 2 et Alpha ARNm cod.
RESULTATS
Les différences entre le brin 1 de l’ADN et l’ARNm sont limitées aux bases Uracile qui remplacent
les bases Thymine de l’ADN.
Le brin 2 de l’ADN et l’ARNm présentent 100% de différences.
CONCLUSIONS
Le brin 2 de l’ADN apparaît comme le brin transcrit et le brin 1 comme le brin non transcrit.
La séquence de l’ARNm se déduit de celle du brin non transcrit en remplaçant la thymine par
l’uracile.
Fermer la fenêtre Comparaison simple.
2. Approche de la traduction
2.1 - Traduction de la séquence d’ARNm en polypeptide
Sélectionner la séquence Alpha ARNm cod en cliquant sur son bouton de sélection.
Cliquer sur la commande Convertir les séquences de la barre d’outils.
Valider successivement l’option Peptidique pour la séquence à afficher, l’option Traduction simple et l’option Résultat dans
la fenêtre Affichage/édition, puis confirmer ce choix en cliquant sur OK.
La séquence Pro-Alpha ARNm s’affiche dans la fenêtre Affichage des séquences et peut être
parcourue en utilisant la barre de défilement horizontal.
2.2 - Comparaison du polypeptide obtenu avec le polypeptide de la banque de données
Cliquer sur le bouton Thèmes d’étude de la barre d’outils.
Ouvrir successivement le menu Expression de l’information génétique et le sous-menu Globine alpha.
Sélectionner Séquence peptidique et valider ce choix en cliquant sur OK.
La chaîne polypeptidique Polypeptide alph s’affiche dans la fenêtre Affichage des séquences et peut
être parcourue en utilisant la barre de défilement horizontal.
Sélectionner successivement Pro-Alpha ARNm et Polypeptide alph en cliquant sur les boutons correspondant à ces
séquence peptidiques
Cliquer sur le bouton Comparer les séquences de la barre d’outils.
Valider l’option Comparaison simple et confirmer en cliquant sur OK.
Cliquer sur l’échelle pour obtenir celle correspondant aux acides aminés.
La nature du traitement effectué et les deux séquences peptidiques s’affichent dans la fenêtre
Comparaison simple. Elles peuvent être parcourues en utilisant la barre de défilement horizontal. Le tiret
indique l’identité des acides aminés par rapport à ceux de la première séquence qui sert de référence.
RESULTAT
Il y a identité des deux chaînes polypeptidiques, elles comportent chacune 141 acides aminés
identiques.
2.3 - Inversion et traduction de la séquence d’ARNm
2.3.1 - Inversion de la séquence d’ARNm

Sélectionner la séquence Alpha ARNm cod en cliquant sur son bouton de sélection.
Dans le menu Edition valider la commande Dupliquer la séquence.
Une nouvelle séquence Alpha ARNm cod s’affiche dans la fenêtre Affichage des séquences.
Après avoir, dans le menu Options, désélectionné la commande Protéger les données, sélectionner la nouvelle
séquence Alpha ARNm cod.
Sélectionner, dans le menu Edition, la commande Inverser la séquence.
Une séquence inverse d’ARNm, i-Alpha ARNm co s’affiche dans la fenêtre Edition des séquences.
Revenir au menu Options et sélectionner à nouveau la commande Protéger les données.
2.3.2 - Traduction de l’ARNm inversé
Sélectionner la séquence i-Alpha ARNm co en cliquant sur son bouton de sélection.
Cliquer sur le bouton Convertir les séquences de la barre d’outils.
Valider successivement l’option Peptidique, l’option Traduction simple et l’option Résultat dans la fenêtre Affichage/édition.
Confirmer ces choix en cliquant sur OK.
La séquence polypeptidique Pro-i-Alpha ARN s’affiche dans la fenêtre Affichage des séquences et
peut être parcourue en utilisant la barre de défilement horizontal.
2.3.3 - Comparaison du polypeptide obtenu avec celui de la banque de données
Sélectionner successivement les séquences Pro-i-Alpha ARN et Polypeptide alph en cliquant sur les boutons de
sélection correspondants.
Cliquer sur le bouton Comparer les séquences de la barre d’outils.
Valider l’option Comparaison simple et confirmer en cliquant sur OK.
La nature du traitement et les deux séquences polypeptidiques comparées s’affichent dans la fenêtre
Comparaison simple. Elles peuvent être parcourues en utilisant la barre de défilement horizontal. Les
tirets indiquent l’identité des acides aminés par rapport à ceux de la première séquence qui sert de
référence.
RESULTAT
La chaîne polypeptidique obtenue est différente de celle de la banque de données
CONCLUSION
Il y a unidirectionnalité de la traduction de l’ARNm en polypeptide
Fermer la fenêtre Comparaison simple en cliquant sur le bouton de fermeture de la barre d’outils
2.4 - Cadre de lecture
2.4.1 - Traduction de L’ARNm à partir des positions 4,7,10
Sélectionner la séquence Alpha ARNm cod en cliquant sur son bouton de sélection.
Dans le menu Edition valider la commande Dupliquer la séquence.
Une nouvelle séquence Alpha ARNm cod s’affiche dans la fenêtre Affichage des séquences.
Sélectionner la séquence Alpha Polypeptide Alph en cliquant sur son bouton de sélection.
Dans le menu Edition valider la commande Dupliquer la séquence.
Une nouvelle séquence Polypeptide Alph s’affiche dans la fenêtre Affichage des séquences.
Dans le menu Options, désélectionner la commande Protéger les données.
Sélectionner la nouvelle séquence Alpha ARNm cod, puis après avoir placé le curseur après la troisième base,
supprimer successivement les bases G, U et A en utilisant le clavier

Sélectionner la nouvelle séquence Polypeptide Alph puis après avoir placé le curseur après le premier acide aminé,
supprimer le en utilisant le clavier
Sélectionner la nouvelle séquence Alpha ARNm cod créée (elle est affichée en bleu dans la fenêtre Affichage des
séquences) en cliquant sur son bouton de sélection.
Cliquer sur le bouton Convertir les séquences de la barre d’outils.
Valider successivement l’option Peptidique pour la séquence à afficher, l’option Traduction simple et l’option Résultat dans
la fenêtre Affichage/édition, puis confirmer ce choix en cliquant sur OK.
Une nouvelle séquence Pro-Alpha ARNm s’affiche dans la fenêtre Affichage des séquences et peut
être parcourue en utilisant la barre de défilement horizontal.
Sélectionner la nouvelle séquence Pro-Alpha ARNm et la séquence Polypeptide Alph puis cliquer sur le bouton Comparer
les séquences de la barre d’outils
Valider l’option Comparaison simple et confirmer en cliquant sur OK.
La nature du traitement et les deux séquences polypeptidiques comparées s’affichent dans la fenêtre
Comparaison simple. Les tirets indiquent l’identité des acides aminés par rapport à ceux de la première
séquence qui sert de référence.
Procéder de la même façon à partir des positions 7 et 10.
RESULTAT
La traduction de l’ARNm à partir des positions 4, 7 et 10 conduit à la même séquence
polypeptidique que celle de la banque de données, diminuée chaque fois d’un acide aminé
2.4.2 - Traduction de L’ARNm à partir des positions 2, 3, 5, 6
Sélectionner la séquence Alpha ARNm cod en cliquant sur son bouton de sélection.
Dans le menu Edition valider la commande Dupliquer la séquence.
Une nouvelle séquence Alpha ARNm cod s’affiche dans la fenêtre Affichage des séquences.
Dans le menu Options, désélectionner la commande Protéger les données.
Sélectionner la nouvelle séquence Alpha ARNm cod, puis après avoir placé le curseur après la première base,
supprimer la en utilisant le clavier
Sélectionner la nouvelle séquence Alpha ARNm cod créée (elle est affichée en bleu dans la fenêtre Affichage des
séquences) en cliquant sur son bouton de sélection.
Cliquer sur le bouton Convertir les séquences de la barre d’outils.
Valider successivement l’option Peptidique pour la séquence à afficher, l’option Traduction simple et l’option Résultat dans
la fenêtre Affichage/édition, puis confirmer ce choix en cliquant sur OK.
Une nouvelle séquence Pro-Alpha ARNm s’affiche dans la fenêtre Affichage des séquences
Sélectionner la séquence Pro-Alpha ARNm et la séquence Polypeptide Alph puis cliquer sur le bouton Comparer les
séquences de la barre d’outils
Valider l’option Comparaison simple et confirmer en cliquant sur OK.
La nature du traitement et les deux séquences polypeptidiques comparées s’affichent dans la fenêtre
Comparaison simple. Elles peuvent être parcourues en utilisant la barre de défilement horizontal. Les
tirets indiquent l’identité des acides aminés par rapport à ceux de la première séquence qui sert de
référence.
Procéder de la même façon à partir des positions 3, 5 et 6.
RESULTAT
La traduction de l’ARNm à partir des positions 2, 3, 5, et 6 conduit à des séquences
polypeptidiques différentes de celle de la banque de données.
CONCLUSION
La traduction de l’ARNm se fait triplet par triplet, sans chevauchement

3 - Approche du code génétique
3 .1 - Création et traduction d’une séquence Poly-U
3.1.1 - Création d’une séquence Poly-U
Dans le menu Fichier, sélectionner la commande Créer.
Sélectionner ARN pour choisir le type de séquence à créer et donner le nom de Poly-U à cette séquence, puis
confirmer ces choix en cliquant sur OK.
Dans la fenêtre Edition des séquences, créer une séquence de 18 bases Uracile en cliquant sur la lettre U du pavé des
bases azotées.
3.1.2 - Traduction de la séquence Poly-U
Sélectionner la séquence Poly-U à l’aide de son bouton de sélection.
Cliquer sur le bouton Convertir les séquences de la barre d’outils.
Valider successivement l’option peptidique pour la séquence à afficher, l’option Traduction simple et l’option Résultat dans
la fenêtre Affichage/édition, puis confirmer ces choix en cliquant sur OK.
La séquence Pro-Poly-U s’affiche dans la fenêtre Edition des séquences.
RESULTAT
La séquence polypeptidique obtenue ne comporte qu’un seul type d’acide aminé, la
phénylalanine.
CONCLUSION
Le codon UUU code pour la phénylalanine
3.2 - Création et traduction d’une séquence Poly-UUC
3.2.1- Création d’une séquence Poly-UUC
Dans le menu Fichier, sélectionner la commande Créer.
Sélectionner ARN pour choisir le type de séquence à créer et donner le nom de Poly-UUC à cette séquence, puis
confirmer ces choix en cliquant sur OK.
Dans la fenêtre Edition des séquences, créer une séquence de 6 UUC en cliquant sur les lettres U et C du pavé des
bases azotées.
3.2.2- Traduction de la séquence Poly-UUC
Sélectionner la séquence Poly-UUC à l’aide de son bouton de sélection.
Cliquer sur le bouton Convertir les séquences de la barre d’outils.
Valider successivement l’option peptidique pour la séquence à afficher, l’option Traduction simple et l’option Résultat dans
la fenêtre Affichage/édition, puis confirmer ces choix en cliquant sur OK.
La séquence Pro-Poly-UUC s’affiche dans la fenêtre Edition des séquences.
RESULTAT
La séquence polypeptidique obtenue ne comporte qu’un seul type d’acide aminé, la phénylalanine.
CONCLUSIONS
Le codon UUC code pour la phénylalanine
Les codons UUU et UUC codent pour le même acide aminé, la phénylalanine, le code
génétique est redondant (ou dégénéré)
3.3- Création et traduction d’une séquence Poly-UC
3.3.1- Création d’une séquence Poly-UC
Dans le menu Fichier, sélectionner la commande Créer.
Sélectionner ARN pour choisir le type de séquence à créer et donner le nom de Poly-UC à cette séquence, puis
confirmer ces choix en cliquant sur OK.
Dans la fenêtre Edition des séquences, créer une séquence de 9 UC en cliquant sur les lettres U et C du pavé des
bases azotées.
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
1
/
14
100%