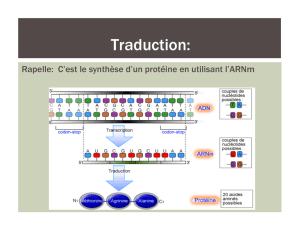
La traduction Le code génétique Traduction : réaction chimique en

La traduction
Le code génétique
Traduction : réaction chimique en trois temps
- Initiation
- Elongation
- Terminaison
Une enzyme : le ribosome
Des substrats : ARNm (4 lettres) + aa-ARNt
Produit : Peptide (20 AA différents)
Base du code génétique
L’ARNm est lu : 5’ → 3’
Synthèse des protéines : N-term → C-term
L’ARNm est décodé par groupe de 3 lettres (codons)
- 43 = 64 codons possibles
- 20 acides aminés donc redondance : groupes de codon
Pas de chevauchement des codons : cadre de lecture défini

Le code génétique est non ambigu : chaque codon a un signal unique
- Acides aminés ou Codons d'initiation (AUG)
- 3 codons STOP
Dégénérescence : 61 codons donnent 20 acides aminés
Faite majoritairement sur la 3ème base du codon
On retrouve des similarités chimiques entre codons
Rôle au niveau du ribosome (cf. élongation)
La redondance minimise les effets dus à des mutations
Mutations synonymes (dans exons) : pas de changement de la séquence protéique
Touche majoritairement la 3ème base du codon mais aussi la 2ème base
On peut aussi obtenir des acides aminés de nature équivalente par mutation de la 1ère base du codon
Exemple : Leu → Ile
Usage des codons : certains codons sont plus retrouvés que d’autre pour un même AA
- Spécifique de chaque génome
- Relation avec la [aminoacyl-ARNt]
Variable selon organismes

Les clés du code génétique
32 ARNt standards
Porte l’anti-codon permettant l’appariement avec l’ARNm
Règle appariement non standard : Wobble
20 ARNt synthétases
Une enzyme par acide aminé
Permet la production des aminoacyl-ARNt
Un ribosome
Ribozyme : ARN qui possèdent la propriété de catalyser une réaction chimique spécifique
Le Wobble :
Appariement non standard codon-anticodon entre la 3ème base du codon et la 1ère base de
l’anticodon (distance légèrement plus grande comparé aux deux autres appariements)
Explique pourquoi le nombre d’ARNt est plus faible que le nombre de codon
Si appariement Watson & Crick : 61 ARNt
Mais chez l’homme : 32 ARNt standards (31 + ARNt initiateur)
Un anticodon de l’ARNt reconnaît plusieurs codons sur l’ARNm
Due à la dégénérescence

Hypothèse du flottement ou tremblement (Wooble)
1ère et 2ème base du codon : appariements Watson & Crick
3ème base du codon : Wobble
Quatre possibilités :
- Pas de reconnaissance codon-anticodon avec la 3ème base du codon
- Anticodon : G → Codon : C ou U (Bases pyrimidines)
- Anticodon : U → Codon : A ou G (Bases puriques)
- Modification chimique des bases : donne l’Inosine (désamination de l’Adénine)
Anticodon : I → Codon : C, A, U
Le Wooble permet d’obtenir des codons synonymes donc diminution du nombre d’ARNt.
Le code génétique est universel
Présent il y a -3,5 Ga
Utilisé par tous les organismes : le code génétique est universel
Les exceptions prouvent universalité
- Touchent quelques codons
- Base d’évolution : règles de Wobble
- Evènements rares / Cas particuliers
Exception la plus fréquente (Wobble) : UGA = Trp
A la base l’évolution des génomes mitochondriaux
Retrouvé aussi dans les mycoplasmes (bactéries)
Dans la mitochondrie humaine, on retrouve d’autres exceptions :
- AUA = Met
- AGA / AGG = codon Stop
22 ARNt (plus petit stock connu)

On retrouve dans certaines protéines des AA modifiés :
- Les Sélénoprotéines (dans tous les organismes)
21ème AA : la sélénocystéine (Ex : Glutathion péroxydase)
Se fait au moment de la traduction par recrutement par l’ARNm d’un facteur d’élongation spécifique
Codon UGA (normalement codon Stop) → Reconnu par la Séléno-Cys-ARNt
- Pyrrolysine (que dans certaines archaea bactéries)
Même mécanisme : Codon UAG (normalement codon Stop)
Eléments nécessaires à la traduction
ARNt synthétase : acide aminé + ARNt → aminoacyl-ARNt
Ribosome : ARNm + aminoacyl-ARNt → Peptide
Aminoacyl-ARNt
Correspondance codon ARNm – acide aminé
Codon sur ARNm
ARNt : Molécule adaptatrice qui permet le chargement d’AA par des liaisons covalentes
Trois structures tige-boucle : forme de feuille de trèfle
Boucles D et Ψ : modification chimiques essentielles pour l’interaction avec le ribosome
Boucle anticodon : modification chimiques essentielles pour l’interaction avec l’ARNm
Modifications chimiques à l’extrémité 3’ : interaction avec l’acide aminé
 6
6
 7
7
1
/
7
100%