changement de langage

Traduction- 2TSbc
page1
METABOLISME DES PROTEINES :
Mécanisme de la traduction
Introduction :
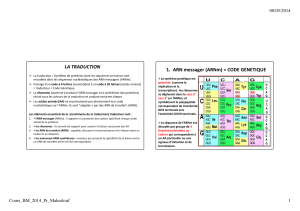
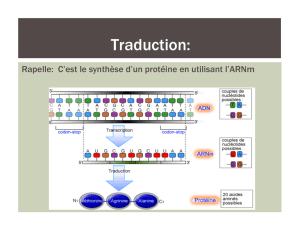
Traduction = synthèse des protéines sur une matrice d'………
1- Caractéristique du code génétique : changement de langage entre
l'ARNm et la protéine correspondante
1.1- Code à 3 lettres : code situé sur l'ARNm (Khorana en 1966)
Un groupe de 3 bases ou triplet de bases de l'ARN m code pour 1 acide aminé.
Ces 3 bases de ARNm s'appellent codon.
Possibilité de combinaison des 4 bases en codon : 43 = 64 codons différents.
- 61 codons ayant un sens (qui correspondent chacun à 1 acide aminé), dont 1 codon
d'initiation (AUG),
- 3 codons non sens ou stop ou de terminaison (UAA, UAG, UGA).
1.2 Code dégénéré
Le code est dégénéré : 61 codons → 20 acides aminés.
Donc plusieurs codons pour le même acide aminé (codons synonymes).
En général les codons synonymes différents par la nature du d'une base (le plus souvent la 3ème).
Exercice : calculer le nombre de codons pour chaque acide aminé.(voir tableau code génétique page
suivante)
Phe Ser Tyr Asp
Leu Pro His Glu
Ile Thr Gln Cys
Val Ala Asn Arg
Met Trp Lys Gly
Il n'y a pas de ponctuation entre les triplets, ils sont lus à la suite les uns des autres.
Seule particularité : 1 codon de départ (établit le cadre de lecture)
3 codons stop.

Traduction- 2TSbc
page2
1.3- Le code génétique est quasi universel
Il est valable pour tous les organismes vivants (bactéries, végétaux, êtres humains), il a donc
été préservé au cours de l'évolution, sauf chez les mitochondries où il y a quelques modifications.
2-.Eléments nécessaires à la synthèse protéique
2.1- ARNm : matrice
2.1.1- Structure ARNm
- Structure monocaténaire, sauf chez certains virus.
- Longueur variable (dépend des protéines codées)
- Sont en faible quantité dans les cellules (5% des ARN chez Escherichia coli).

Traduction- 2TSbc
page3
- Structure tridimensionnelle : repliement de la chaîne en épingle à cheveux (liaisons …
entre bases complémentaires). Il est de plus en plus évident que la formation d'appariements
intramoléculaires soit un moyen de régulation de la traduction.
2.1.2- ARNm : matrice
Porte le message de l'ADN (ordre dans lequel seront attachés les acides aminés).
-Il est traduit dans le sens 5' → 3'
-La totalité ne sert pas de matrice :
*chez les eucaryote : chapeau et queue polyA qui ne sont pas traduits
*chez les procaryotes et les eucaryotes : en amont coté 5' et en aval coté 3' il existe
des séquences non traduites.
Chez les procaryotes, les ARNm peuvent être polycistroniques ou polygéniques :
1 ARNm → plusieurs protéines (cas opéron lactose).
Cistron (ancien terme) : portion d'ADN qui code pour 1 chaîne polypeptidique.
-Chez les procaryotes, il y a couplage transcription - traduction : l'ARNm est traduit alors que
la trancription n'est pas terminée.
2.2- ARNt : molécules adaptatrices
2.2.1- Structure
- Petites molécules d'ARN monocaténaire de 70 à 90 nucléotides (procaryotes) 73 à 95
nucléotides (eucaryotes)
- Ils représentent 15 à 20% des ARN chez Escherichia coli.
- La structure tridimensionnelle est conservée, dite en feuille de trèfle. Elle se replie sur elle
même en forme de L grâce à la formation de liaisons H entre les bases des
ribonucléotides.

Traduction- 2TSbc
page4
- Nombreuses bases inhabituelles (≈ 10 par molécules)
Les ARN de transfert
contiennent de nombreux
nucléotides modifiés
chimiquement après
transcription. Seuls quelques
modifications sont
représentées ici.L’inosine(I)
résulte d’une désamination de
l’adénine dans l’adénosine.La
dihydrouridine(D) résulte
d’une réduction de l’uracile
dans l’uridine. Dans la
pseudouriidine(Y), la liaison
de l’uracile au ribose se fait
par le biais du carbone N°5.
2.2.2- Rôle d'adaptateur
C'est l'intermédiaire entre l'ARNm et les acides aminés.
-L'ARNt porte l'anti-codon : groupe de 3 ribonucléotides portant les bases complémentaires
du codon de l'ARNm (liaisons H).
Ex : anticodon : 5' GAA3'
Codon : 3'
L'ARNm est lu dans le sens ….
Il y a plus d'ARNt que d'acides aminés : ARNt avec des anti-codons différents qui codent
pour les mêmes acides aminés = ARNt isoaccepteurs.
-L'ARNt se lie à l'acide aminé par liaison covalente sur le dernier nucléotide 3' (A).
Il y a deux étapes":
1ère étape : activation de l'acide aminé par l'AMINOACYL-ARNt-SYNTHETASE
2ème étape : fixation

Traduction- 2TSbc
page5
2.3- Ribosome: site de la synthèse protéique
2.3.1- Structure
C'est un complexe de grande taille formé d'ARN ribosomique (représente 80% des ARN) et
de protéines ribosomiques.
Ils sont formés de 2 sous unités (une petite et une grosse).
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
1
/
11
100%