Texte rédigé - Académie de Lyon

Les OGM : Organismes Génétiquement Modifiés
Intervention de Mme MOUNIER, Maître de Conférences
UCBL Lyon I, CGMC, Bat Gregor Mendel
Stage à Public désigné 00180 du PAF SVT 2008-2009 – Académie de Lyon
« OGM » implique un transfert de gène, donc l’étude d’OGM nécessite de bien connaître la
structure de l’ADN et des outils qui permettent de le manipuler.
LES OUTILS DU GENETICIEN
1) La structure de l’ADN
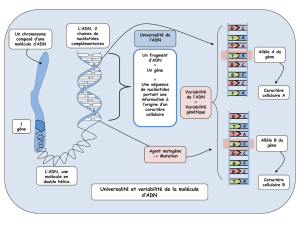
Un gène est une partie de la molécule d’ADN qui peut être transcrit en ARN. Le
génome humain contient plus de 3 milliards de paires de bases (par génome haploïde) mais
seulement 1-2% de gènes. Un gène eucaryote peut contenir des exons et des introns, ces
derniers ne sont pas présents dans la molécule d’ARN mature.
L’ADN est une longue molécule double brin qu’on lit par convention de 5’ vers 3’. Les
deux brins sont reliés au niveau de leurs bases azotées, par appariement des bases
complémentaires, A-T et C-G, par le biais de liaisons hydrogènes, liaisons faibles pouvant
être rompues par la chaleur, 90 - 100°C. Il y a 3 liaisons H entre G et C contre seulement 2
entre A et T. Par conséquent les régions de l’ADN riches en G et C sont plus stables. A
l’intérieur d’un brin, les nucléotides adjacents sont reliés entre eux par une liaison
phosphodiester, liaison covalente qui est crée ou rompue par action d’une enzyme.
Le sens de transfert de l’information génétique, ou dogme de la génétique
moléculaire, est unidirectionnel : l’ADN est transcrit en ARN, seuls les ARNm sont traduits en
protéines. Mais ce dogme est égratigné car les rétrovirus peuvent faire de l’ADN à partir
d’ARN.
Un des 2 brins d’ADN est le brin matrice, utilisé pour la fabrication de l’ARN (le brin à
partir duquel l’ARN polymérase fabrique l’ARN), l’autre brin est le brin codant (ou non
matrice), car sa séquence est identique à celle de l’ARNm « qui a le code »
2) La manipulation de l’ADN
Quand on manipule l’ADN in vitro et si on le réintroduit dans une cellule, on doit
observer et analyser les effets obtenus au niveau de la cellule, l’organisme et la population.
On peut manipuler plus facilement l’ADN car il est très résistant, l’ARN est très fragile de par
sa structure.
La découverte de l’enzyme transcriptase reverse a été un progrès technique
considérable car on a pu produire un ADN, manipulable, à partir des ARN.
Les principales enzymes permettant de manipuler l’ADN sont les nucléases (couper),
les polymérases (copier) et les ligases (coller). Notez qu’il existe d’autres enzymes, dite de
modification de l’ADN.
Les enzymes de restriction ou endonucléases permettent de couper l’ADN, chacune
au niveau d’un site spécifique, le site de restriction. Histoire plus longue !
L’enzyme Eco RI est la première enzyme de restriction isolée chez Escherichia coli,
souche R. Les endonucléases isolées ensuite chez cette bactérie ont été nommées Eco RII,
RIII etc.
Eco RI reconnaît la séquence 5’GAATTC3’ (ce site est un palindrome car
complémentaire de CTTAAG).
Il existe trois types de coupures selon les enzymes :
5’ 3’ 3’ 5’
3’ 5’ sortant franche 5’ 3’ sortant

Afin de copier de l’ADN, on fournit à l’ADN polymérase (qui polymérise de 5’ en 3’)
dans un tube (on ne les utilise plus !) : un modèle (le brin matrice), une amorce ADN ou ARN
simple brin, qui fournit 3’ OH libre, puis on ajoute les 4 dNTPs (désoxy ribo nucléotide tri
phosphate).
On utilise une ADN ligase qui relie les deux brins d’ADN dont les extrémités sont
compatibles en créant une liaison phosphodiester entre deux nucléotides adjacents.
Une nouvelle technique utilisant le mécanisme de l’interférence ARN (prix Nobel
2006) est largement utilisée dans les laboratoires
21-L’hybridation moléculaire
L’hybridation moléculaire est le fondement de la génétique moléculaire : grâce à la
complémentarité des bases, deux acides nucléiques sont capables de s’hybrider. Si on
chauffe l’ADN, on le dénature et les deux brins se séparent. Si on mélange plusieurs
molécules différentes d’acides nucléiques simple brin, elles pourront s’hybrider, se
renaturer : on obtiendra un homoduplex (= la molécule ADN d’origine) et si les séquences
sont totalement ou partiellement complémentaires, on obtient un hétéroduplex. Cette
hybridation est d’une spécificité absolue entre les 2 molécules hybridées : elle permet de
repérer une séquence parmi des millions. On peut ainsi utiliser cette propriété pour faire des
criblages : rechercher une séquence particulière dans un échantillon d’ADN.
L’expérimentateur, connaissant ces propriétés, peut rechercher une cible (séquence
d’ADN) grâce à une sonde moléculaire par une hybridation spécifique ou non, en jouant sur
les conditions d’hybridation (température, agents de salinité etc.).
Une sonde moléculaire est une fraction d’acides nucléiques, ADN ou ARN, sous
forme simple brin, qui permet de repérer une séquence cible, elle même simple brin, par
hybridation moléculaire. On la marque afin de la repérer une fois hybridée : au niveau des
extrémités ou dans toute sa longueur avec des composés contenant un atome radioactif
(phosphore 32, par exemple, de durée de vie de 15 jours) ou un élément fluorescent. Voir
l’illustration avec l’exemple de marquage de chromosomes : on traite les chromosomes en
enlevant les protéines et on dénature l’ADN ; on ajoute la sonde fluorescente qui se fixe sur
le gène et permet le localisation du site d’hybridation.
Enfin, la notion de puce à ADN (qui fait appel à l’hybridation moléculaire, la
miniaturisation, le traitement informatique) : on réalise des micro-dépôts sur des plaques
comportant des puits soit d’ADN complémentaire, soit des oligonucléotides de synthèse :
chaque puits contiendra un ADN correspondant à une région plus ou moins longue d’un
gène. On peut faire jusqu’à 300000 dépôts par plaque de 1 cm2. On hybride la puce avec
des ADN complémentaires marqués provenant d’une transcription inverse des ARNm
présents dans une cellule soit normale soit d’une autre condition physiologique, normale ou
pathologique. Le traitement informatique des résultats donne une idée de l’activité
transcriptionnelle des deux conditions testées, notion du transcriptome (mise en évidence de
gènes actifs (transcrits) dans un contexte particulier et comparaison des taux d’expression
dans les 2 situations (idée de l’activité transcriptionnelle dans une situation donnée).
2- Le clonage
Un clone est une population (de molécules, de cellules, d’individus etc.) qui ont un
ancêtre commun (tous identiques à l’ancêtre commun).
Le clonage moléculaire d’un fragment d’ADN consiste à couper grâce à des enzymes de
restriction l’ADN qui contient le fragment que l’on veut cloner. On digère également et le
vecteur de clonage (plasmide par exemple) avec la même enzyme pour que les extrémités
d’ADN soient compatibles.
Après ligation, si le fragment d’ADN a été inséré dans le plasmide, on obtient un plasmide dit
« recombinant », que l’on va introduire dans une cellule vivante pour l’amplifier
(transformation de la cellule hôte). Ensuite on amplifie la bactérie transformée par culture, ce
qui permettra d’obtenir en grande quantité de plasmide et de là du fragment d’ADN cloné.

En tube à essai, sur des micro-quantités, on coupe L’ADN dont on dispose avec des
enzymes de restriction et on obtient le fragment d’ADN que l’on veut cloner. On coupe alors
le vecteur (plasmide) avec la même enzyme pour que les extrémités soient compatibles. On
ajoute un gène de sélection dans le plasmide. On obtient alors un plasmide dit
« recombinant ». On passe ensuite le plasmide recombiné dans une cellule vivante pour
l’amplifier (c’est la transformation). On manipule la bactérie en la brusquant (pour faire entrer
le plasmide).
La phase dite d’amplification consiste en une multiplication de la bactérie génétiquement
modifiée.
3- Les protéines produites par les bactéries (protéines recombinantes)
La production de l’insuline n’est pas simple car le polypeptide qui serait obtenu par
la bactérie transformée ayant le gène eucaryote de l’insuline, ne serait pas actif comme
hormone. En effet, dans la cellule eucaryote, le polypeptide issu de la traduction de l’ARNm
doit subir une maturation (élimination des régions terminales, puis clivage interne), ce que la
bactérie ne sait pas réaliser. On a donc fait deux gènes synthétiques : l’un codant le
polypeptide A et l’autre le polypeptide B. On purifie ensuite les deux polypeptides après le
clonage des deux gènes synthétiques dans les bactéries et on les réassocie pour obtenir
l’insuline active (réalisé dans les années 80).
L’Hormone de croissance
L’Erythropoiétine facteur VIII, Interféron gamma, facteur de croissance,
IL2,…mais aussi des enzymes de l’agroalimentaire (fromagerie, boulangerie,…) : on ajoute
dans certains des ferments (enzymes) fabriqués par génie génétique.
On produit également des enzymes de dégradation des graisses, des enzymes
utilisées dans le blanchiment des papiers et textiles, la dépollution, en laboratoire pour
modifier L’ADN (avant on les isolait dans des bactéries maintenant, on les produit) : ex
Thermophilus aquaticus (tac Taq polymérase : enzyme pouvant agir à 95°C, utilisée pour la
PCR)
4- Le transfert de gènes chez les eucaryotes
La transfection est le transfert de gènes dans les cellules en culture. Mélange entre
infection et transformation : le terme est plus appliqué aux cellules eucaryotes dont le
principe est le suivant :
a. Construire un vecteur avec le gène d’intérêt et un ou plusieurs gènes de sélection :
On introduit le vecteur dans les cellules eucaryotes selon diverses techniques. On
sélectionne ensuite les cellules qui ont incorporé le vecteur (cf schéma sur présentation).
Dans la plupart des cas, l’ADN pénètre dans la cellule par endocytose. Le vecteur utilisé
dans ces expériences est appelé vecteur d’expression : il doit contenir les éléments
permettant le clonage du transgène dans les bactéries et l’expression du transgène dans la
cellule hôte eucaryote. Pour optimiser son expression, il doit contenir une séquence poly A et
des séquences de donneur et d’accepteur d’épissage.
Le vecteur doit atteindre le noyau (par des les mécanismes pas toujours bien élucidés) pour
être exprimé (transcrit), et peut s’incorporer dans un chromosome de la cellule hôte.
b. Il y a deux sortes de transgènes : le gène d’intérêt avec tout ou partie de ses
propres séquences régulatrices, ou couplé à des séquences régulatrices provenant d’un
autre gène
c. un gène rapporteur qui va rapporter ce qui se passe dans la cellule, couplé aux
séquences régulatrices du gène d’intérêt :

. CAT (Chloramphénicol Acétyl Transférase enzyme bactérienne) : que l’on peut
mettre en évidence par test ELISA,
. Lac Z : enzyme bactérienne codant la beta galactosidase qui clive le lactose, si on
ajoute un analogue du lactose, le XGal, l’enzyme clive ce substrat qui se transforme en un
composé bleu, et donc donne une coloration bleue.
. Luciférase (isolé du ver luisant) ou GFP (Green Fluorescent Protein isolée d’une
méduse).
Les méthodes d’insertion du transgène dans les cellules eucaryotes sont de trois
types :
a- Chimiques : l’ADN peut être mélangé avec des lipides sous forme de liposome. La
bicouche membranaire fusionne avec le liposome s’ouvre et l’ADN passe dans la cellule.
Une autre méthode consiste à précipiter l’ADN avec du phosphate de Calcium qui sera plus
facilement internalisé dans la cellule.
b- Biologiques : vecteurs viraux (virus du singe SV40, adénovirus, rétrovirus, herpes)
dont le génome a été modifié ; on utilise le tropisme du virus pour la cellule cible.
c- Mécaniques (électroporation) on fait subir aux cellules des chocs électriques brefs et
intenses ce qui produit des trous dans la membrane. Il existe aussi le canon à particules
quand la cellule a une protection (paroi des cellules végétales, œufs d’insectes). Enfin la
micro injection dans le noyau : ex ovocyte.
Le taux de succès de ces diverses techniques est variable, mais grâce aux systèmes de
sélection, on arrive à obtenir des cellules transformées recherchées malgré parfois un faible
taux de réussite.
Le gène peut s’exprimer transitoirement : lors de la division cellulaire notamment, le
plasmide ne se réplique pas et il se dilue dans la population de cellule eucaryotes filles.
Ou bien ou de façon stable le plasmide s’insère dans les chromosomes et se
transmet ainsi d’une génération à la suivante.
Quelques applications : Vérification au niveau cellulaire. ???
Photos de cellules transfectées : fibroblastes : expression du gène de la β
galactosidase (cellules bleues) mais toutes ne sont pas transformées. Cellules de
foie avec GFP et gène fluorescent. On repère que le gène soit bien transféré puis on
regarde que les cellules produisent bien l’enzyme.
Stade suivant au niveau de l’organisme: cellule de la moelle osseuse de la souris.
Injecter des cellules transformées dans un organisme pour savoir si le gène transféré
peut être actif.
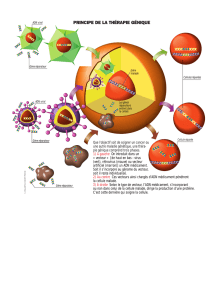
Un exemple (Hopital Necker en 2000), exemple de thérapie génique somatique : sur
une immunodéficience liée à X, mutation d’un gène codant une sous-unité
d’Interleukine. Les patients ont une immunodéficience sévère (enfants bulles) : on a
introduit le gène sauvage, dans le vecteur, puis ce vecteur recombinant est transfecté
dans des cellules de la moelle osseuse prélevée chez l’enfant malade, puis mises en
culture.. Ces cellules transformées ont été réinjectées dans le sang circulant et ont
migré vers la moelle dans laquelle le transgène a fabriqué le polypeptide manquant.
Cette méthode était porteuse d’espoir. Mais quelques années après 2 ou 3 de ces
enfants sont morts d’une leucémie : peut être le vecteur viral est en cause (mutation
le rendant pathogène?). Le protocole a été arrété.
En 2006, sur 630 essais de thérapie génique (somatique) , on comptabilise 63 % d’essais
pour guérir de cancer, 12 % pour des pathologies monogéniques,… les vecteurs sont le plus
souvent des vecteurs viraux car ils rentrent pénètrent avec plus d’efficacité plus facilement
dans les cellules.
Thérapie génique : in situ (mucoviscidose, dans la gorge)
In vivo
Ex vivo

Beaucoup d’espoir mais peu de réussite.
On s’oriente vers la thérapie cellulaire avec des cellules souches.
 6
6
 7
7
 8
8
1
/
8
100%