Virologie : méthodes de diagnostic - microbiologie

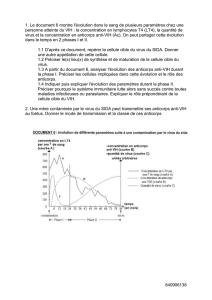
BTS ABM
AFBB
Page 1
Microbiologie
VIROLOGIE
VIROLOGIE : METHODES DE DIAGNOSTIC
1. Techniques de prélèvement
Cf. fiche « Phase pré-analytique des analyses de virologie médicale ».
2. Méthodes de diagnostic direct
2.1. Méthodes de diagnostic rapide
La détection directe d’antigènes viraux dans les produits pathologiques présente l’avantage d’être
relativement simple et rapide à mettre en œuvre. Elle permet la mise en route de mesures prophylactiques
ou thérapeutiques immédiates, mais ne s’applique qu’aux infections où des quantités importantes de virus
sont produites.
Les virus sont riches en constituants protéiques ou glycoprotéiques pouvant faire l’objet d’une
reconnaissance immune par des anticorps spécifiques. Ces anticorps constituent des outils diagnostiques
précieux ; il peut s’agir d’anticorps polyclonaux d’origine humaine (produits à l’occasion d’infections
naturelles) ou le plus souvent animale (obtenus par immunisation expérimentale) ; de plus en plus, sont
utilisés des anticorps monoclonaux (d’origine habituellement murine) dont le grand intérêt réside dans
la précision de l’épitope reconnu. Il peut être intéressant d’utiliser des anticorps dirigés contre des
antigènes de groupe capables de reconnaître plusieurs virus appartenant à un même genre (adénovirus ou
entérovirus) ou au contraire de cibler un épitope spécifique de type (différenciation entre virus herpes
simplex types 1 et 2).
La méthode consiste à visualiser le complexe antigène-anticorps ; pour ce faire de nombreuses
techniques sont disponibles :
- agglutination de particules sensibilisées (hématies, latex, particules de gélatine),
- fluorochromes,
- marqueurs enzymatiques (technique ELISA et ses très nombreuses variantes).
Le système de marquage peut être lié directement à l’anticorps primaire ou, le plus souvent, fixé sur un
anticorps secondaire dirigé contre les immunoglobulines de l’espèce animale chez laquelle a été produit
l’anticorps primaire.
Avantages
Inconvénients
Règles de conservation et de transmission des
prélèvements moins strictes ;
Facilité de mise en œuvre ;
Rapidité d’exécution ;
Nombreux tests commercialisés.
Faible sensibilité ;
Coût relativement élevé ;
Nombre limité de virus recherchés.

BTS ABM
AFBB
Page 2
Microbiologie
VIROLOGIE
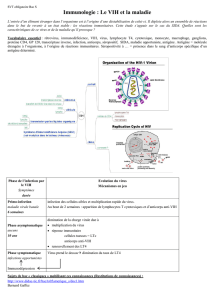
Principales applications des techniques de détection directe d'antigènes viraux dans les
prélèvements pathologiques en virologie médicale :
Exemple 1 : PASTOREX® ROTAVIRUS (Bio-Rad)
Les Rotavirus sont reconnus comme les agents les plus fréquemment rencontrés lors des gastro-entérites aigües. Au début et
durant la phase aiguë de la maladie, des particules virales de Rotavirus sont excrétées dans les selles en quantité importante
(108 particules virales par gramme de selles).
Des particules de latex, sensibilisées avec la fraction immunoglobulinique (IgG) d’un antisérum de lapin anti-Rotavirus,
sont agglutinées en présence de Rotavirus présents dans les selles. Des particules de latex sensibilisées avec une fraction
immunoglobulinique (IgG) d’un sérum de lapin normal sert de latex de contrôle négatif.
Exemple 2 : PATHFINDER® HERPES SIMPLEX VIRUS (Bio-Rad)
Le système de détection directe des antigènes du virus herpes simplex (HSV) de types 1 et 2 Pathfinder® est un test par
fluorescence directe permettant d'identifier et de typer le virus herpes simplex dans des échantillons cliniques directs et des
isolats de culture cellulaire. Le diagnostic en laboratoire d'une infection à HSV joue plusieurs rôles dans la prise en charge du
patient. Un diagnostic spécifique d'infection à herpes virus permet d'instituer rapidement un traitement par l'agent antiviral le
plus approprié. Outre les informations qui permettent d'orienter le traitement, un diagnostic spécifique peut s'avérer utile pour
prévenir une infection à HSV. Chez certaines femmes enceintes chez lesquelles l'infection est active au moment de
l'accouchement, la pratique d'une césarienne permet de réduire le risque de transmission du virus au nouveau-né.

BTS ABM
AFBB
Page 3
Microbiologie
VIROLOGIE
La méthode d'identification du HSV communément admise à l'heure actuelle consiste à isoler le virus dans une culture de
cellules. Les méthodes de culture cellulaire amplifient les quantités de virus, qui sont initialement faibles, mais nécessitent de 1
à 7 jours pour être menées à leur terme.
Les récents progrès de la production d'anticorps monoclonaux ont donné des anticorps qui présentent une grande spécificité
vis-à-vis du HSV-1 et du HSV-2. Les études cliniques ont démontré l'efficacité de ces anticorps dans le diagnostic des
infections à HSV. Le système de détection directe des antigènes du virus herpes simplex (HSV) de types 1 et 2 Pathfinder®
utilise quatre anticorps monoclonaux conjugués à la fluorescéine pour identifier et typer le HSV dans les échantillons
cliniques directs et les isolats de culture cellulaire. Ce test allie la spécificité et la reproductibilité des anticorps monoclonaux à
la vitesse et à la simplicité d'un test par fluorescence directe. L'identification et le typage présomptifs du HSV sont possibles
dans l'heure qui suit la réception d'un échantillon clinique direct.
Ce test utilise des anticorps monoclonaux conjugués à la fluorescéine (2 anti-HSV-1, 2 anti-HSV-2) afin d'identifier et de typer
le virus herpes simplex. Les anticorps anti-HSV-1 réagissent avec des polypeptides dont les poids moléculaires sont
respectivement de 150.000 et 85.000 daltons. L'un des anticorps anti-HSV-2 réagit avec des polypeptides dont les poids
moléculaires de 50.000 daltons environ ; l'autre anticorps anti-HSV-2 réagit avec un polypeptide dont le poids moléculaire
approximatif est de 79.000 daltons. Déposés dans des puits séparés d'une lame contenant l’échantillon, les anticorps
monoclonaux dirigés contre le HSV-1 et le HSV-2 réagissent avec les antigènes viraux dans les cellules infectées. Une
étape de lavage élimine les anticorps non liés. Au microscope à fluorescence, les cellules infectées par le HSV présentent
une fluorescence vert pomme caractéristique sur un fond contre-coloré en rouge.
Exemple 3 : PATHFINDERTM RSV (Bio-Rad)
PATHFINDERTM RSV est un test immunoenzymatique (ELISA) qualitatif pour la détection directe du virus respiratoire
syncytial (RSV) dans les liquides de lavage ou d’aspiration nasale.
Le virus respiratoire syncytial est une des causes principales de bronchiolite et de pneumonie chez le nourrisson et l’enfant.
L’affection est la plus sévère au cours de la première année. Les épidémies dues à ce virus surviennent pratiquement chaque
année en automne, hiver et printemps. Le RSV est responsable d’une affection respiratoire importante chez l’adulte sain.
Cependant, l’affection chez l’adulte tend à être moins forte que chez le jeune enfant. Un diagnostic rapide d’une infection à
RSV a d’autant plus d’importance aujourd’hui que l’on a mis au point un traitement efficace. Il permet de poser un diagnostic,
de débuter le traitement et de contrôler précocement la propagation de l’affection. Les méthodes de culture cellulaire
traditionnelles peuvent prendre jusqu’à 3 semaines et dépendent fortement de la qualité du transport de l’échantillon. Les tests
immunologiques ont l’avantage d’être rapides, techniquement simples et d’un coût moins élevé que les cultures à isolement de
virus. Les méthodes d’immunofluorescence et de dosage immunoenzymatique (EIA) ont montré leur efficacité dans la
détection du RSV.
Le PATHFINDERTM RSV utilise des anticorps polyclonaux et monoclonaux pour la détection du RSV directement dans
des échantillons de patients. Cette méthode de dosage allie la spécificité et la reproductibilité des anticorps monoclonaux à la
rapidité et l’efficacité d’un EIA. Des résultats qualitatifs sont disponibles en 90 minutes après le début du dosage. Cette
méthode d’analyse représente une alternative fiable par rapport aux cultures cellulaires qui nécessitent beaucoup de temps et
aux techniques de fluorescences subjectives qui demandent beaucoup de travail.
Les antigènes RSV présents dans l’échantillon se fixent aux anticorps polyclonaux anti-RSV capté sur le fond du tube.
Les anticorps monoclonaux conjugués à la peroxydase de raifort (HRP) dirigés contre le RSV se fixent à l’antigène lui-
même fixé sur la paroi du tube. Une étape de lavage permet l’élimination de l’échantillon et du conjugué non fixés. Le
substrat de peroxydase ajouté au tube entraînera une coloration bleue si des anticorps conjugués à la HRP sont
présents. Un examen par spectrophotométrie de l’échantillon détermine la présence ou l’absence de RSV. Un échantillon

BTS ABM
AFBB
Page 4
Microbiologie
VIROLOGIE
présentant une coloration bleue ou dont la valeur d’absorbance est supérieure ou égale à la Valeur Seuil supérieure de la Zone
d’Incertitude est positif en antigène RSV. Un échantillon ne montrant aucune coloration bleue ou dont la valeur d’absorbance
est inférieure ou égale à la Valeur Seuil inférieure de la Zone d’Incertitude est négatif en antigène RSV.
Figure 1 : schémas de principe des méthodes utilisant un marquage :
Marquage direct Marquage indirect « Sandwich »

BTS ABM
AFBB
Page 5
Microbiologie
VIROLOGIE
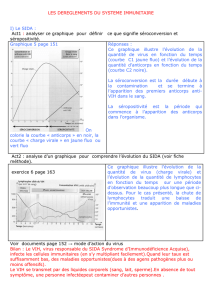
2.2. Méthodes de culture des virus
Parasites intracellulaires obligatoires, les virus ne peuvent se multiplier que dans des cellules vivantes.
L’isolement de virus à partir des prélèvements biologiques a représenté pendant de nombreuses années, le
seul moyen de diagnostic direct des infections virales.
Trois systèmes sont susceptibles de permettre la réplication des virus :
l’animal de laboratoire,
l’œuf de poule embryonné
les cellules en culture (seul système utilisé en pratique courante).
2.2.1. in vivo : inoculation à l’animal et ovoculture
Le recours à l’animal de laboratoire ou à l’œuf de poule embryonné est devenu exceptionnel, même si ces
procédés sont encore employés pour l’isolement de certains virus :
- souriceaux nouveau-nés pour les coxsackievirus A ;
Les coxsackie A donnent des « paralysies » flasques les coxsackie B donnent des paralysies par nécrose cérébrale, cela chez les
souriceaux inoculés à la naissance. Certains coxsackie A ne se multiplient que chez le souriceau nouveau-né et pas en culture
de cellules, en particulier le coxsackie A1. Tous les coxsackie B se multiplient en culture de cellules. Le nom de coxsackie
vient de la ville des USA où l'on a isolé le premier d'entre eux.
(Source : site de virologie complet sur http://www.chups.jussieu.fr/polys/viro/poly/)
- œufs de poule embryonnés pour les virus de la grippe.
Figure 2
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
1
/
14
100%