Introduction : But de l`article

Genetic Suppression of Polyglutamine
Toxicity in Drosophila.
Parsa Kasemi-Esfarjani, et al ; Science 287, 1837 (2000)
Suppression génétique de la toxicité
liée aux agrégats de polyglutamines
chez la drosophile
Introduction : But de l’article
La chorée de Huntington, comme d’autres, est caractérisé notamment par des anomalies cellulaires :
des agrégats (cytosoliques et /ou nucléaires) de polyglutamines. Ceux-ci causent une toxicité
engendrant une mort cellulaire. Il est donc intéressant d’utiliser cette caractéristique comme modèle
pour la maladie d’Huntington. On créée donc des drosophiles transgéniques présentant un
phénotype du type de cette maladie : par mutagénèse dirigée, on introduit dans le génome des
séquences polyCAG, qui codent donc pour des polyglutamines. A partir de ces souches mutantes, on
peut alors chercher des gènes suppresseurs par mutagénèse. On peut alors espérer enrayer la
maladie par l’utilisation de ces gènes suppresseurs. Ici on en utilise 2 différents dont l’expression
donne des protéines, dHDJ1 et dTRP2, qui rétablissent en partie le phénotype sauvage dans les
lignées transgéniques présentant des agrégats de polyglutamines. DHDJ1 est un homologue de la
protéine de choc thermique humaine 40/hDJ1, et dtRP2 est un homologue de la protéine répétée
tétratricopeptide humaine.
Matériel et Méthode
L’approche utilisée ici consiste à réalisé un crible du génome de la drosophile pour trouver des gènes
supprimant de manière dominante la toxicité de la polyglutamine.
L’élément P
Le gène d’intérêt est inséré dans un élément P
(multi-site de clonage) avec lequel on
transforme une bactérie, qui est multipliée. Le
plasmide contient un gène de résistance à un Ab,
ce qui permet de sélectionner les bactéries le
portant. Celui-ci est alors isolé, et est alors
injecté dans un embryon de drosophile. On
injecte également une source de transposase (un
plasmide qui contient le gène de la transposase
mais qui ne peut pas s’insérer dans le génome).
Pied P
w+
Site de clonage
Pied P
Origine de réplication
Ampir

On utilise un système de sélection pour repérer les lignées qui nous intéressent. On multiplie alors
les drosophiles.
Description et élaboration des lignées transgéniques poly-Gln
Par PCR, on synthétise des polyCAG de différents types : Les normaux font 20 répétitions
nucléotidiques (ce sont les témoins, chez l’homme les cas non pathologiques présentant des
répétitions de 20 CAG), puis les augmentés font 127 répétitions nucléotidiques. Ce polyCAG est placé
en aval d’un promoteur UAS dans un plasmide transposable (élément P), ensuite utilisé pour créer
une lignée transgénique de drosophiles. Le promoteur UAS est inductible par la protéine GAL4. On
place sur la construction, une séquence codant pour un épitope de l’hémagglutinine (noté HA), de
façon a ce que cette séquence soit transcrite avec le polyCAG, puis traduite par la suite en épitope.
On s’en servira pour un marquage des polyglutamines. On nomme cette construction UAS-
polyCAG_HA.
On crée une seconde lignée transgénique comportant une séquence GAL4 (cDNA du gène GAL4)
placée en aval d’un promoteur GMR. Le promoteur GMR est une structure promotrice permettant
une expression uniquement dans l’œil. Le promoteur est activé par une protéine spécifique des
disques imaginaux des yeux, jouant alors le rôle de facteur de transcription au niveau du futur œil.
On croise ensuite les mouches de la lignée transgénique comportant la construction GMR-GAL4 (on
créer une lignée à partir d’une seule mouche présentant le génotype GMR-GAL4), par des mouches
de lignées transgéniques comportant les séquences UAS-polyCAG_HA. On sélectionne 3 lignées de
chacun des types d’insertion : 3 lignées pour UAS-20CAG et 3 lignées pour UAS-127CAG. On utilise
des mouches issues de différentes lignées pour tester l’influence du fond génétique (l’endroit
d’insertion peut avoir un effet sur le phénotype). Il se peut que certaines lignées expriment mieux les
polyglutamines que d’autres.
Après les croisements, on se retrouve avec des mouches exprimant GAL4 au niveau des yeux et
comportant la construction UAS-PolyCAG_HA. GAL4, dont la transcription est induite par des facteurs
de transcription présents au niveau de l’œil, va coder pour la protéine GAL4, qui va alors activer le
promoteur UAS. Le promoteur UAS activé va permettre la transcription de PolyCAG_HA, qui va être
transcrit en polyGln_épitopeHA.
5’
3’
GAL 4
Promoteur GMR
5’
3’
Poly CAG
Promoteur UAS
HA
Figure 1 : construction génomique incorporée dans le génome de la drosophile
Figure 2 : Construction génomique incorporée dans le génome de la drosophile

Etude des gènes suppresseurs
On crée des lignées transgéniques par insertion d’éléments P, dans les cellules germinales (voir
protocole mutagénèse avec élément P), que l’on croise avec les individus des lignées possédant un
phénotype type Huntington (agrégats de polyglutamine). 7000 séquences autosomales sont insérées
dans des éléments P, que l’on utilise pour la création des lignées transgéniques. Seulement quelques
5’
3’
Poly CAG
UAS
HA
5’
3’
GAL 4
GMR
Facteur de transcription
spécifique de l’œil
Transcription de GAL4
Protéine Gal4
ARNm Gal4
Traduction de l’ARNm Gal4
Induction de la transcription
Induction de la
transcription
Poly CAG
HA
ARNm
Transcription
Traduction
Polyglutamine avec épitope HA
Figure 3 : Chaine de réaction aboutissant à la synthèse des polyglutamines dans les lignées transgéniques

lignées engendrées par ce croisement sont intéressantes. Deux principaux types se démarquent : on
a observé 29 lignées qui perfectionnaient la dégénérescence, et 30 qui tendaient à réduire la
pathologie. Dans cet article nous sont rapportés les résultats pour les deux premières lignées pour
lesquelles les effets de suppression ont été directement confirmés : Lignée EU3500 et lignée EU3220.
Résultats
Description et élaboration des lignées transgéniques poly-Gln
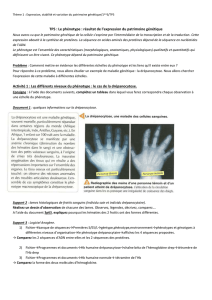
Pour les lignées 20Q : On a un phénotype sauvage
Pour les lignées 127Q : on trouve un phénotype montrant des agrégats au niveau des remnants des
rétines. On se sert alors d’une de ce type de lignée pour réaliser la recherche de gènes suppresseurs.
Etude des gènes suppresseurs
Les deux gènes suppresseurs n’agissent pas de la même façon.

Technique d’études
On utilise différentes techniques pour visualiser les différents phénotypes :
- Microscopie électronique à balayage
- Microscopie photonique : observation de l’aspect extérieur de l’œil, immunomarquage de
coupes histologiques
Microscopie photonique
Aspect extérieur
On observe simplement les yeux de drosophile au microscope photonique, on peut alors apprécier
l’intensité de coloration des pigments rouge. Cette coloration est telle car la drosophile exprime le
gène w+, de phénotype rouge, sauvage.
Immunomarquage
L’épitope HA pourra être reconnu par des anticorps dirigé contre l’hémagglutinine : on réalisera donc
un immunomarquage de coupes histologiques d’œil, utilisant des anticorps primaires anti-HA, puis
des anticorps secondaires fluorescents, pour mettre en évidence la présence ou non d’amas de
polyglutamines chez les drosophiles. Les anticorps secondaires utilisés sont flanqués par FITC
(Fluorescein isothiocyanate). La fluorescéine absorbe les radiations bleues (max 490nm) et restitue
une fluorescence verte (max 520nm).
On réalise également un immunomarquage DAPI (Di aminido phényl indol) qui permet un marquage
non spécifique de tous les noyaux. On peut alors localiser les corps cellulaires sur les coupes. Eclairé
en lumière violette (max 372nm), il émet une fluorescence bleue (max 456nm).
Microscopie électronique à balayages
On observe les yeux par microscopie électronique à balayage après avoir utilisé des techniques de
fixation adaptées.
 6
6
 7
7
 8
8
 9
9
1
/
9
100%