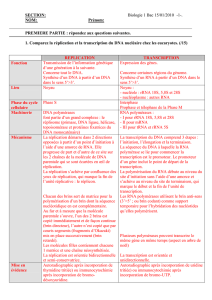
Cours de Biologie Moléculaire et Génétique : Acides Nucléiques, ADN, ARN
Telechargé par
marivaux2008

1
Biologie moléculaire I
Introduction générale
Les êtres vivants ont besoin de se
pérenniser. Ainsi, dans chaque espèce
biologique, les individus (des plus simples
comme les virus au plus complexes tels que les
animaux et plantes supérieurs) sont dotés
d’aptitude de se reproduire; c’est-à-dire un
individu donnant naissance à une descendance
qui lui ressemble, du moins en termes
d’appartenance à la même espèce. La
génération d’un individu ressemblant à son
parent implique la transmission des caractères
génétiques du parent à sa descendance. Ces
caractères sont stockés dans les molécules
sièges de l’information génétique (DNA ou
RNA, dans le cas des virus à RNA). Pour être
transmis du parent à la descendance, cette
information doit être copiée le plus fidèlement
possible, en sorte que le descendant ait la
même information génétique que son géniteur.
Aussi, pour apprécier la ressemblance du
parent et descendance cette information
génétique doit s’exprimer. Il se dégage donc à
ce niveau deux processus biologiques majeurs:
transmission de l’information génétique d’un
individu (virus, procaryotes et eucaryote) à sa
descendance et l’expression de cette
information génétique.
Au niveau moléculaire, la transmission
de l’information génétique d’un individu à sa
descendance et l’expression de l’information
génétique chez un individu impliquent
principalement trois types molécules: DNA,
RNA et protéines. En fait ces deux processus
biologiques correspondent en réalité à des
transferts de l’information génétique d’une
molécule à une autre.
On distingue plusieurs modalités de
transfert de l’information d’une molécule à
une autre:
DNA →DNA qui correspond à la réplication
ou duplication (parent vers la descendance)
RNA→RNA qui correspond à la réplication
ou transcription (chez les virus) (parent vers
la descendance ou expression)
DNA→RNA qui correspond à transcription
(expression)
RNA→ protéines correspond à traduction
(expression
RNA→DNA qui correspond à la
rétrotranscription
Dans le cadre de ce cours nous nous
attarderons sur la réplication (DNA→DNA) et
la transcription (DNA→RNA). De fait le cours
s’articulera autour de trois chapitres:
Chapitre I. Généralités structurales des acides
nucléiques.
Chapitre II. Réplication du DNA
Chapitre III. Transcription du RNA DNA-
dépendante.
2
Chapitre I. Généralités structurales des
acides nucléiques
Introduction
Chez tous les êtres vivants connus, il en
existe deux types d’acides nucléiques, l'acide
désoxyribonucléique ou ADN, et l'acide
ribonucléique ou RNA. DNA et RNA sont de
longs polymères linéaires (ou circulaires pour
le DNA) de nucléotides, unis par des liaisons
phosphodiesters.
Deux chaînes de DNA ou deux segments
d’une chaîne de RNA sont susceptibles de
s’associer grâce à des appariements de leurs
bases par des liaisons hydrogène de type
Watson-Crick, ce dont il résulte une structure
secondaire et une structure tertiaire adaptées
aux fonctions, et éventuellement, à la
duplication des acides nucléiques.
L’information génétique est stockée dans la
séquence ou succession des bases azotées des
chaînes polynucléotidiques.
Dans le DNA, une séquence ou
succession des bases azotées dans une chaîne
polynucléotidique peut constituer une unité
capable de coder pour la synthèse d’un RNA
biologiquement actif. Cette unité est qualifiée
de gène. Dans une molécule du DNA des
gènes peuvent se chevaucher. L’ensemble des
gènes d’un individu biologique constitue le
génome. L’ensemble des RNA transcrits du
génome constitue le transcriptome. Certains
types particuliers de RNA sont traduits en
protéines dont l’ensemble chez un individu
biologique est appelé protéome.
I- Nucléotides
Les nucléotides sont les monomères
des acides nucléiques. Ces nucléotides sont
formés à partir de trois molécules simples:
l’acide phosphorique (H
3
PO
4
), des oses à 5
carbones (pentoses) et des bases azotées
(purines ou pyrimidines).
1- Molécules simples
a- Acide phosphorique
Le phosphate inorganique est un ion stable
formé à partir de l’acide phosphorique H
3
PO
4
.
On l’écrit souvent Pi. Des esters de phosphate
peuvent se former entre un phosphate et un
groupement hydroxyle libre (alcool, énol,
phénol...). La condensation d’un phosphate et
d’un autre acide, par exemple un autre
phosphate, donne un anhydride. Il y a aussi des
anhydrides mixtes avec les acides
carboxyliques par exemple.

3
b- Ribose et désoxyribose
Le ribose est un pentose de la série D,
dont tous les hydroxyles sont orientés à
droite (représentation de Fisher). Dans les
acides ribonucléiques (RNA), il est cyclisé en
ribofuranose: anomère β spécifiquement.
Le désoxyribose, composant des acides
désoxyribonucléiques (DNA) est dérivé du
ribose par une réduction de la fonction alcool
secondaire du carbone n°2. Le désoxyribose
confère à cet acide nucléique une plus grande
stabilité propre à sa fonction de conservation
de l’information génétique.
C- Bases azotées
C1- Purine et pyrimidine
Les bases azotées des acides
nucléiques appartiennent à deux classes de
molécules selon le noyau aromatique qui en
constitue le squelette. Le noyau pyrimidine
est le plus simple: c’est un noyau aromatique
à six atomes, quatre carbones et deux azotes;
les deux azotes en position méta (n° 1 et 3).
Le noyau purine est constitué de deux
noyaux hétérocycliques accolés, un de six
atomes et l’autre de cinq atomes, ayant deux
carbones en commun au milieu. Par rapport à
ces carbones communs, les azotes occupent
des positions symétriques (n° 1 et 3 à gauche,
n° 7 et 9 à droite). Les différentes bases
rencontrées dans les acides nucléiques en
dérivent selon les substituants que portent les
atomes de ces noyaux. De nombreux
médicaments appartiennent aussi à ces deux
classes de bases azotées.
4
C2- Les bases azotées
Les bases azotées sont des molécules
aromatiques dont le noyau est soit une purine
(bases puriques), soit une pyrimidine (bases
pyrimidiques).
- Les bases puriques
Les bases puriques sont au nombre de 2:
l’adénine et la guanine. Les purines ont un
double noyau aromatique comportant à
gauche un cycle hexagonal de 4 carbones et 2
azotes et à droite un cycle pentagonal de 3
carbones (dont 2 communs avec le
précédent) et 2 azotes. L’adénine est
constituée d’un noyau purine dont le carbone
6 est substitué par une fonction amine. Elle
est la seule des bases nucléiques dont la
formule ne contient pas d’atome d’oxygène.
La guanine est constituée d’un noyau purine
dont le carbone 2 est substitué par une
fonction amine et le carbone 6 par une
fonction cétone.
- Les bases pryrimidiques
Les bases pyrimidiques sont au
nombre de 3: la cytosine, l’uracile et la
thymine. Les pyrimidines ont un noyau
aromatique hexagonal de 4 carbones et 2
azotes. La cytosine est constituée d’un
noyau pyrimidine dont le carbone 4 est
substitué par une fonction amine et le
carbone 2 par une fonction cétone. L’uracile
est constituée d’un noyau pyrimidine dont
les carbones 2 et 4 portent des fonctions
cétone. La thymine est aussi constituée d’un
noyau pyrimidine dont les carbones 2 et 4
portent des fonctions cétone, mais dont le
carbone 5 est substitué par un méthyl.
2- Nucléosides
a) Les nucléosides
Les nucléosides sont constitués d’un
pentose, le D-ribofuranose dans les

5
ribonucléosides ou le 2-désoxy-D-
ribofuranose dans les désoxyribonucléosides,
uni à une purine telle que l’adénine (A) ou la
guanine (G), ou une pyrimidine telle que la
cytosine (C), l’uracile (U) ou la thymine (T),
grâce à une liaison β-glycosidique établie
entre l’atome d’azote N-9 des purines ou N-1
des pyrimidines et le carbone C-1’ du
pentose.
Chez les êtres vivants, on identifie
essentiellement quatre ribonucléosides,
l’adénosine, la guanosine, la cytidine et
l’uridine, et quatre désoxyribonucléosides, la
désoxyadénosine, la désoxyguanosine, la
désoxycytidine et la thymidine où le préfixe
désoxy est généralement omis parce que dans
la cellule la thymine est toujours unie au
désoxyribose.
Le cycle du ribofuranose peut se
présenter sous quatre conformations
possibles : C-2’ endo, C-2’ exo, C-3’ endo et
C-3’ exo; dans tous les cas, l’un des deux
atomes de carbone, C-2’ ou C-3’, est hors du
plan défini par les quatre autres atomes du
cycle, du même côté que l’atome C-5’
(configuration endo) ou du côté opposé
(configuration exo).
Les purines et les pyrimidines sont
des molécules hétérocycliques légèrement
basiques et très conjuguées des résonances
entre les atomes de leur cycle donnent à la
plupart des liaisons un caractère de double
liaison partielle ce dont il résulte que les
purines sont des molécules presque planes
avec cependant une très légère distorsion et
les pyrimidines des molécules
rigoureusement planes. Une autre
conséquence de la résonance est leur forte
absorption dans l’ultraviolet, à 260 nm
environ. Dans les nucléosides puriques,
seules deux conformations de la liaison
glycosodique sont stériquement permises, syn
et anti; les nucléosides pyrimidiques se
présentent généralement dans la
configuration anti.
6
Figure 1. Nucléosides et nucléotides
b) Nucléotides
Les nucléotides sont des nucléosides
phosphorylés. Les nucléotides cellulaires
sont les ribonucléosides et les
désoxyribonucléosides 5’ mono-, di- ou
triphosphate, abrégés en NMP, NDP, NTP,
dNMP, dNDP et dNTP, respectivement (d
pour désoxy). Les groupes phosphate sont
repérés par les lettres α, β et γ. Le ribose ou

7
le 2-désoxyribose et le phosphate α sont unis
par une liaison ester DG°’ = – 10 kJ mol –1
et les phosphates α, β et γ par deux liaisons
anhydride d’acide phosphorique DG°’ = – 30
kJ.mol
–1
. Les principaux ribonucléotides
cellulaires sont l’adénosine, le guanosine-, le
cytidine-et l’uridine- 5’-monophosphate,
abrégés en AMP, GMP, CMP et UMP,
respectivement ; l’adénosine-, le guanosine-,
le cytidine- et l’uridine 5’-diphosphate, ou
ADP, GDP, CDP et UDP ; l’adénosine, le
guanosine-, le cytidine- et l’uridine 5’-
triphosphate, ou ATP, GTP, CTP et UTP.
Les désoxyribonucléotides correspondant
sont précédés de la lettre d, sauf pour les
dérivés de la thymidine étant donné que dans
la cellule la thymine n’est unie qu’au
désoxyribose.
Les ribonucléosides et les
désoxyribonucléosides 5’-di- ou triphosphate
sont des acides relativement forts dont les
groupes phosphoryle se dissocient en libérant
trois ou quatre protons, respectivement. Les
anions forment des complexes stables avec
les cations divalents tels que Mg
2+
et Ca
2+
.
Les nucléotides jouent des rôles centraux
dans le métabolisme: l’ATP est l’unité
énergétique dans la plupart des processus
cellulaires, le GTP est une source d’énergie
essentielle dans la synthèse des protéines,
l’UDP-glucose est l’intermédiaire activé dans
le métabolisme du galactose et dans la
synthèse des polyosides, tout comme le CDP-
diacylglycérol est l’intermédiaire activé dans
la synthèse des phospholipides. Des
nucléotides adényliques sont des constituants
de cofacteurs enzymatiques, tels que le
NAD+, le NADP+, le FAD et le CoA. Enfin,
l’ATP, le GTP, le CTP et l’UTP sont les
substrats dans la synthèse des acides
ribonucléiques (RNA), tandis que le dATP,
le dGTP, le dCTP et le TTP sont les substrats
dans la synthèse de l’acide
désoxyribonucléique (DNA). Les cellules
possèdent aussi des nucléotides cycliques tels
que l’adénosine 3’,5’-phosphate (AMP
cyclique ou cAMP) et le guanosine 3’,5’-
phosphate (GMP cyclique ou cGMP) qui
interviennent dans la régulation du
métabolisme.
c) Nomenclature des unités nucléotiques
Les bases azotées sont
conventionnellement désignées par une
initiale et par une couleur: l’adénine par A et
la couleur verte, la guanine par G et la
couleur jaune, etc...
Chaque base peut entrer dans la structure de
deux nucléosides, selon que le sucre est un
ribose ou un désoxyribose.
Chaque nucléoside peut être lié à un,
deux ou trois phosphates. On les désigne par
des sigles conventionnels: GMP pour
guanosine monophosphate, CDP pour
cytidine diphosphate, ATP pour adénosine
triphosphate, etc...
On désigne par nucléotides les
nucléosides monophosphates: AMP ou acide
8
adénylique, dTMP ou acide
désoxythymidylique, etc...
Les nucléosides polyphosphates sont des
diphosphates: ADP ou GDP... ou encore des
triphosphates, les plus riches en énergie: ATP
ou GTP ; etc...
Les acides nucléiques sont formés par
une polycondensation de nucléotides AMP,
CMP, GMP et UMP pour les acides
ribonucléiques, dAMP, dCMP, dGMP et
dTMP pour les acides désoxyribonucléiques.
3- Les liaisons phosphodiesters
Des ribonucléotides sont les unités
structurales des RNA et des
désoxyribonucléotides celles du DNA.
Dans les RNA et dans le DNA, les
nucléotides s’unissent les uns aux autres par
une liaison où le groupe 5’-phosphate de
l’un estérifie le groupe 3’-hydroxyle de
l’autre. L’acide phosphorique en 5’ engage
donc deux de ses fonctions acide dans une
liaison dite, pour cette raison,
phosphodiester, sa troisième fonction acide
demeurant libre; au pH physiologique, cette
dernière est complètement ionisée et
chargée négativement. Les enchaînements
de ribonucléotides ou de
désoxyribonucléotides au moyen de liaisons
de ce type présentent une extrémité 5’ où un
groupe 5’-phosphate à deux de ses
fonctions acide libres et une extrémité 3’ où
un groupe 3’ hydroxyle est libre. Ainsi, une
chaîne polynucléotidique, tout comme une
chaîne polypeptique, présente une polarité.
Par convention, on écrit et lit toujours une
chaîne polynucléotidique dans le sens 5’
P→3’ OH; ACG et GCA correspondent
donc à des composés différents. Au sein des
cellules, les charges négatives des acides
nucléiques sont habituellement neutralisées
par des interactions avec des ions
Tableau I. Nomenclature des bases azotées, nucléosides et nucléotides.

9
métalliques tels que Mg
2+
ou des protéines
qui contiennent de nombreux résidus
basiques, tels que ceux de l’arginine ou de
la lysine, et sont donc chargées
positivement. Les bases puriques et les
bases pyrimidiques sont susceptibles de
s’empiler parallèlement les unes sur les
autres en raison d’une combinaison de
forces de van der Waals et d’interactions
dipolaires.
Figure 2. enchaînement des nucléotides
4- Liaison hydrogène
Une liaison hydrogène est une liaison de
faible énergie entre deux atomes attirés
l’un vers l’autre pour des raisons
électrostatiques l’un étant riche en
électrons donc nucléophile et l’autre
n’ayant que les protons de son noyau
donc électrophile.
• Ainsi l’atome d’hydrogène, dont l’unique
électron est par nécessité dans l’orbitale qui
unit cet atome au reste de la molécule, ne
peut qu’être électrophile et comme tel attiré
par les atomes ayant des doublets
électroniques libres.
• Les atomes d’azote et surtout d’oxygène
possèdent respectivement deux et quatre
électrons de leur couche périphérique qui ne
participent pas aux orbitales des liaisons
covalentes de la molécule. Ceci leur confère
un caractère nucléophile qui leur permet
d’exercer une attraction sur les atomes
électrophiles voisins, en particulier les
atomes d’hydrogène: cette attraction
constitue une liaison hydrogène.
• Lorsque les orbitales qui unissent d’un côté
l’atome d’hydrogène à la molécule de gauche
et de l’autre côté les doublets électroniques
libres avec l’atome nucléophile sont dans le
même axe, la liaison hydrogène est plus
forte.
5- Hybridation des bases azotées
Lorsqu’un acide nucléique est en solution les
molécules forment des liaisons hydrogènes
associant, les nucléotides deux par deux, de
10
sorte qu’un nucléotide à adénine se lie avec
un nucléotide à thymine (ou à uracile dans un
RNA) et un nucléotide à guanine avec un
nucléotide à cytosine.
• On désigne cette liaison sous le terme
d’hybridation.
A-T -21
kJ
C-G – 63 kJ
• L’hybridation adénine-thymine est moins
stable (2 liaisons hydrogène, -21 kJ) que
celle entre guanine et cytosine.
• L’hybridation guanine-cytosine est plus
stable (3 liaisons hydrogène, -63 kJ) que
celle entre adénine et thymine.
Figure 3. brins appariés
II- DNA
Une chaîne (ou brin) de DNA est un
polymère linéaire de désoxyribonucléotides
unis par des liaisons phosphodiester; les
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
1
/
18
100%