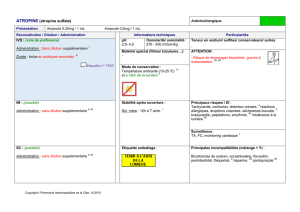
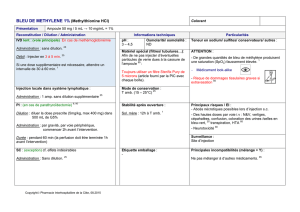
Techniques de dénombrement des bactéries d’Escherichia Coli :
Par culture :
- Réalisation de dilutions décimales ;
- Dénombrement sur boite de Pétri ;
- Dénombrement par spectrophotométrie.
Par qPCR :
- Extraction de l’ADN total à partir de la culture ;
- Vérification de l’ADN sur gel d’agarose ;
- Dénombrement de nombre de copies de l’ADNr 16S des bactéries de la culture par
qPCR.
Dénombrement de levures par cellule de Malassez.

Dénombrement d’Escherichia Coli par culture :
Notre culture possède une microflore bactérienne qu’on va essayer de dénombrer, et
cela en réalisant des dilutions en cascade en prélevant 1 mL de notre solution et qu’on dépose
dans 9 mL de sérum physiologique (10-1), puis on prélève 1 mL aussi de ce tube qu’on transfère
dans un 2eme tube pour la dilution (10-2), et ainsi de suite jusqu'à la dilution (10-8).
Par la suite on prend les tubes de dilutions de -5 à -8, on prélève 100 µL qu’on étale sur
des boites de pétri, en double boite par dilution, et l’étalement se fait à l’aide d’un râteau pour
bien étaler la suspension sur la gélose. Ensuite on va incuber nos boites pendant 24h à 37°C et
le couvercle en bas.
- Dénombrement sur boite de Pétri :
Le lendemain on procède au comptage des colonies sur chaque boite de pétri.
Dénombrement des colonies sur les boites :
N° Boîte
Facteur de dilution
Nbre colonies
1
-8
0
2
-8
2
1
-7
1
2
-7
0
1
-6
3
2
-6
8
1
-5
0
2
-5
0
Les résultats obtenus dans notre expérience ne sont pas significative car on n’a pas assez
de colonies qui ont poussées après incubation, et probablement cela est dû à une erreur de
dilution ou bien on n’a pas pris assez de bactérie lors de l’ensemencement.
Pour cela on a pris les résultats du groupe DZ qui sont les suivants :
N° Boîte
Facteur de dilution
Nbre colonies
Moyenne
1
-6
18
20,5
2
-6
23
1
-5
71
60
2
-5
49
Sachant que nous avions mis 100µL, il a fallu mettre les résultats en mL en suivant la formule
donnée pour la boite de dilution -5 :
N = nombre de colonies * Facteur de dilution = 60 * 105 * 10 = 6 * 107 UFC/mL.
- Dénombrement par spectrophotométrie :
On fait une extraction totale d’ADN des 2 mL d’Escherichia Coli, puis on mesure la densité
optique à 600 nm à des temps différents. Les résultats sont les suivant :

Temps (min)
DO (à 600 nm)
T=0
0,029
T=60
0,046
T=120
0,219
T=140
0,379
T=205
0,591
Sachant que 1 unité de DO à 600 nm correspond à environ 8x108 cellules par mL pour
Escherichia Coli.
On va tracer la courbe de DO= f (temps).
Voir la feuille millimétrée.
Calcule du nombre de bactéries / ml présentes dans la culture :
1 unité de DO 8x108
0,591 X
Bactéries / ml
Ce résultat est un peu élevé par rapport à ce qui est attendu mais il est cohérant avec les
résultats attendus car il est de l’ordre de 108.
Techniques de dénombrement des bactéries d’Escherichia Coli par qPCR :
Cette technique consiste à mesurer tout au long de la réaction et en temps réel la quantité
d’ADNr 16S, grâce a un marqueur fluorescent et cela indique le nombre d’amplicons d’ADN
qui se trouve dans la bactérie.
Voici les résultats de la q PCR après extraction d’ADN :
Discussion :
On a fait 2 dilutions une a 1/10 et une autre a 1/100 et on a obtenu une moyenne de
1,82x107 dans les 2µl, donc une moyenne de 9,10x106 par µl, et on multiplie par le facteur de
dilution 10 x 200 µl pour obtenir une valeur de 1,82x1010 dans la quantité d’ADN initiale, et
cela représente les 7 opérons d’ADNr 16 S totales présentes dans les bactéries E. Coli.

Le nombre de copies 16S d’E. Coli / ml est de 9,10x109 et on divise par 7 pour obtenir
le résultat final de 1,30x109 d’E. Coli / ml.
Par contre pour la deuxième dilution à 1/100 les résultats sont pareil que la première
dilution et cela est dû à une erreur de ma part dans la dilution d’on l’obtention des même
résultats et on a une seule courbe qui représente l’ADN1 et 2 en même temps.
Figure 1 : l’ARNr 16S correspondant à l’ADNr 16S un constituant de la petite sous-unité
ribosomales des procaryotes.
Dénombrement de levures par cellule de Malassez :
Principe
La numération cellulaire est la détermination du nombre de cellules contenues dans un
volume précis de milieu liquide. On exprime le résultat de la numération en concentration
cellulaire, c’est à dire en nombre de cellules par litre. La numération cellulaire est réalisée
directement par comptage au microscope, à l’aide de la cellule de Malassez.
La cellule de Malassez possède un quadrillage spécifique comportant 100 rectangles,
Parmi les 100 rectangles totaux, on trouve 25 rectangles qui sont divisés en 20 petits carrés afin
de faciliter le comptage.
- le volume correspondant au quadrillage total est égal à 1 mm3
- Chaque rectangle correspond à un volume 100 fois plus faible, soit 0,01 mm3.

Figure 2 : observation microscopique de la suspension des levures sur cellule de Malassez.
Résultat :
On a compté sur 10 rectangles un total de 276 levures. Donc une moyenne de 27,6 levures.
Nombre de cellule par ml = N x 100 x 103 x (inverse de la dilution)
Nombre de cellule par ml = 27,6 x 100 x 103 x 10 = 27,6 x 106.
Nombre de cellule = 2,76x107 levures / ml.
Conclusion :
Ces techniques nous ont permises de réaliser des dénombrements d’Escherichia coli. Et
la technique de qPCR reste la méthode la plus précise, mais également la plus chère à mettre en
œuvre. Par ailleurs, une simple erreur dans la manipulation peut avoir des conséquences sur le
résultat, il faut donc manipuler soigneusement l’ADN.
1
/
5
100%