POLY METABO LV303

POLY METABO LV303.pdf
UNIVERSITE PIERRE ET MARIE CURIE (PARIS 6)
“BIOCHIMIE FONDAMENTALE - LV303”
METABOLISME
1° Introduction. Transporteurs de sucres (perméases, PTS, ABC). Glycolyse (rappels).
2° Gluconéogénèse, métabolisme du glycogène.
3
°
Voie des pentoses
phosphates
3
°
Voie
des
pentoses
-
phosphates
.
4° Cycle de Krebs.
5° Acides gras : dégradation, biosynthèse. Corps cétoniques. Voie du glyoxylate.
6
°
Phosphorylations oxydatives photosynthèse (rappels) Navettes métaboliques
6
Phosphorylations
oxydatives
,
photosynthèse
(rappels)
.
Navettes
métaboliques
.
7° Métabolisme de l’azote. Acides aminés : biosynthèse, dégradation.Transaminations.
Cycle de l
'
urée.
Cycle
de
lurée.
8° Régulations, adaptation, et intégration du métabolisme.
Ouvrage recommandé : Lehninger Principles of Biochemistry
Fourth Edition, 2004 by W. H. Freeman & Company
K
eq
aA + bB cC + dD
K
eq
ENTHALPIE-ENTROPIE
0
[
C
]
c
[D]
d
0
K
eq
ΔG = ΔG'
0
+ RTln
[]
[D]
[A]a [B]b = ΔH - TΔS , avec ΔG'
0
= - RT ln Keq
Ared Aox + n e-
eq
REACTIONS REDOX
E =
E
'
0
+
RT
ln
[Aox ]
Δ
E
'
0
=
E
'
0
E
'0
Δ
G
'
0
=
nF
Δ
E
'
0
1
2
ENERGETIQUE DU TRANSPORT
E =
E
0
+
nF
ln
[Ared]
Δ
E
0
=
E
ox
0
-
E
red
Δ
G
0
=
-
nF
Δ
E
0
S
1
2
ENERGETIQUE DU TRANSPORT
ΔG = ZF ΔΨ + RT ln [S2]
[S1]
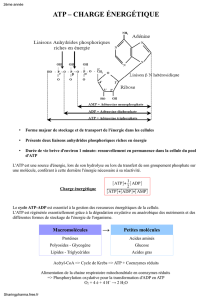
Rappels d’énergétique
Rappels
d’énergétique
2
ATP A
D
PA
M
P
NN
NH2
D
M
O
O
O
SYSTEME ADENYLATE
NN
O
-O-P-O-P-O-P-O-CH2
O
O
O
O-O-O-
OH OH
CONH
2
NAD(P)
+
/NAD(P)H
O
AMP-O-P-O-CH2
(P)
2
N
CONH2
N+
NAD(P)
/NAD(P)H
OH OH
NAD(P)+RNAD(P)H
NH
N
O
NH
NH
O
FAD/FADH2
N N O
CH2-(CHOH)3-CH2O-ADP
FAD
NNH O
R
FADH2
Système adénylate, NAD, FAD 3
Transporteurs
(A) (B)
Annu Rev
(C)
Annu
.
Rev
.
Biochem. 2004.
73:241–68
4
J. Bact., 2000, 182 : 5029–5035 Encyclopedia of life sciences © 2001, John Wiley
& Sons, Ltd. www.els.net

Cellule épithéliale
face apicale
face basale
Glucose
Glucose
2 K
+
[K+] = 4 mM [K+] = 140 mM
2 K
+
ATP
[Na+] = 12 mM
2 Na+
3 Na+ADP + Pi
[Na+] = 145 mM
Lumière
intestinale
Sang
Transport du glucose dans les cellules épithéliales
5
Métabolisme du glucose
Encyclopedia of life sciences © 2005, John Wiley & Sons, Ltd. www.els.net
6
Métabolisme
du
glucose
O
CH2OH
O
CH2OH
O
CH2OH
Pi
O
O
OP
O
O
ATP
1
1b
1a
Glc Glc-1P
G
l
è
U
T
P
O
CH2OP
P
O
H
2
C
O
C
H
2
O
H
ADP
1
G
l
y
c
o
g
è
n
e
P
O
H
C
O
C
H
O
P
UDP-Glc
U
T
P
UDPPPi
14 15
P
O
H
2
C
C
H
2
O
H
A
T
PADP
2
3
G
l
c
-
6
P
F
-
6
P
P
O
H
2
C
O
C
H
2
O
P
F
-
1
,
6
P
2
CH2OH-CO-CH2OP
5
6
7
Pi
DHAP
F
6
P
2
CHO-CHOH-CH2OPPOOC-CHOH-CH2OP
-OOC-CHOH-CH2OP
6
7
8
1
0
NAD+
NADH H+
ATP ADP
G-3P
1,3P 2
GA
3PGA
P
Y
R
-OOC-CHOP-CH2OH -OOC-COP=CH2-OOC-CO-CH3
9
1
0
H2OATP
ADP
NADH H+
N
A
D
+
11
12
1
3
2PGA PEP
P
Y
R
CO2
-OOC-CHOH-CH3
CH2OH-CH3CHO-CH3
N
A
D
NADH H+
NAD+
1
3
LACT
EtOH
7
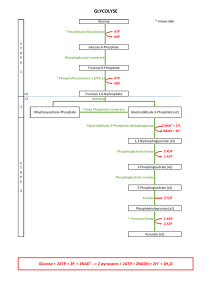
Glycolyse anaérobie et fermentations
GLU GLU-6P FRU-6P
FRU 1 6P
(GLU)n
FRU
-
1
,
6P
2
G3P DHAP
PEPOAA GLYC (TG)
Ami
OAA PYR PYR LACT
Ami
no
acides
ATP
Gluconéogénèse
CH3-CO-COO- + HCO3--OOC-CH2-CO-COO-
ATP
ADP + Pi
P
C
PYR OAA
MITO :
-OOC-CH2-CO-COO-
GTP
GDP
PEPCK
CH2=C(OP)—COO- + HCO3-
OAA
PEP
CYTO :
GDP
OAA
PEP
8
Transformation PYR / PEP

C
O
Biotine
N
H
C
O
(
C
H
)
C
O
HN NH
p
uis transfert du "COO" sur
divers
CO
HN N-COO-
N
H
—
C
O
—
(
C
H
2
)
4—
S
HCO3-
A
T
P
p
accepteurs
S
A
T
P
Biotine et carboxylations (Pyr carboxylase…)
GLU
Extérieur
Ca
2+ Cytoplasme
T1
T2
T3
Ca
SP
GLU-6Pase
GLU-6P PiGLU
T2
+
Lumière du RE
9
Glucose-6 phosphatase
ATP ADP + Pi
FRU
-
26
-
P2
ATP
PFK
FRU 2
,
6P2
AMP Citrate
H+
+-
FRUCTOSE 6 P
FRUCTOSE 1 6 bi h h t
FRUCTOSE
-
6
-
P
FRUCTOSE
-
1
,
6
-
bi
s
p
h
osp
h
a
t
e
FDPase
FRU
-
26
-
P2
H2O
P
i
FRU 2
,
6P2
AMP -Citrate
+
i
Cycle phosphofructokinase/fructose
16
bisphosphatase
10
Cycle
phosphofructokinase/fructose
-
1
,
6
-
bisphosphatase
11
Régulations de la PFK (1)
11
d’après Lehninger Principles of Biochemistry
Fourth Edition, 2004 by W. H. Freeman & Company
Régulations de la PFK (2)
12
d’après Lehninger Principles of Biochemistry
Fourth Edition, 2004 by W. H. Freeman & Company

ATP ADP
FRU-6P FRU-2,6-P2
KINASE
F2,6 P/K
KINASE
ATP Pi
cAMP/PKA PrPase cAMP/PKA
F2,6 P/K-P
PHOSPHATASE
ADP H2O
FRU-6P FRU-2,6-P2
H
2
O
P
i
H
2
O
P
i
FRU-2,6-P2
PFK FDPase
13
Fructose-2,6-bisphosphatase/kinase et AMPc
1) α-1,6-glucosidase
2
)
p
hos
p
hor
y
lase
P
i
phosphorylase
transférase
)
p
p
y
61-P
1
P
i
1
-P
3
Dégradation du glycogène
CH
2
OH
GLU-1P + UTP
O
2
O-P-O-P-O-uridine
(
U
D
PG
)
(D )
+ PPi2 Pi
pyrophosphatase
UDPG-pyrophosphorylase
pyrophosphatase
UDPG + (GLU)
—> UDP + (GLU)
UDPG + (GLU)
n
—> UDP + (GLU)
n+1
glycogène synthétase
14
Biosynthèse du glycogène
UTP PP
i
2 P
i
2
i
i
1
2
1
UDPG h h l
GLU-1P UDPG
1
1
2
UDPG
-pyrop
h
osp
h
ory
l
as
e
P
y
ro
p
hos
p
hatas
e
P
i
UDP
3
4
3
yp p
Glycogène synthétase (GS)
GLUn+1
P
i
UDP
4Glycogène phosphorylase (GP
)
GLUn
Cycle biosynthèse/dégradation du glycogène
15
adrénaline ou glucagon
adénylate
cyclase (i)
adénylate
cyclase (a)
cyclase (i)
cyclase (a)
cAMP
ATP
cAMP
ATP
PKA
(
a
)
PKA
(
i
)
()
()
ATP
ATP
(glycogène-
phosphorylase)
kinase
(
i
)
(GP)K-P(a) glycogène-
synthétase(a) GS-P(i)
ATP
ATP
()
glycogène-
phosphorylase(i) GP-P(a)
GLYCOGENE
ATP
GLUCOSE 1-P
16
Cascade AMPc-dépendante

ATP ADP
PKA
GPK
(inactive)
Ca2+GPK
(part active)
Ca2+GPK-P
(active)
Ca2+ Régulations de la glycogène
phosphorylase kinase
(inactive)
(part
.
active)
(active)
Pr.Pase
phosphorylase
kinase
Pr.Pase
H2O
Pi
2 ATP
GPK
ATP
G6P
P
P
2 ADP
GPK
Régulations de la
glycogène
GP B,
R
()
GP B,
T
(i ti )
AMP
GP A
,
T
(
P
)
GP A
,
R
(
P
)
GLU
P
P
2 Pi
glycogène
phosphorylase
(
active
)
(i
nac
ti
ve
)
,
()
(inactive)
,
()
(active)
2 H2O
Pr.Pas
e
Glycogène-synt. A
(active) Pr.
Pase
PKA
ATP PiRégulations de la
glycogène synthétase
Glycogène-synt. B (P)
(
inactive
)
ADP H2O
17
glycogène
synthétase
GS
(GLU)
n
Dé
g
radation de
(
GLU
)
n
GS
n
GP
g()
n
Biosynthèse de (GLU)n
UDPG
G1P
Accélération de l'exportation de GLU
G6P
F6P GLU GLU sanguin
F2 6P
2
PFK
FDPase
Dégradation de F2,6P2
F2
,
6P
2
PFK
FDPase
Inactivation de la PFK (défaut d
'
activation)
Biosynthèse de F2,6P2
+-
F1,6P2
Inactivation de la PFK (défaut d activation)
Activation de la FDPase (défaut d'inhibition)
PYR Accumulation de F6P, puis de G6P
Blocage de la glycolyse
18
Signal β-adrénergique et métabolisme du glycogène hépatique
O
CH 2OP
O
CH 2OP
NADP +H2O
NADP +
COO -
CO 2CH 2OH
O
GLU-1P
1
23
O
NADH, H +NADH, H +
CH 2OP CH 2OP
GLU GLU-6P 6-P-Gluconolactone 6-P-Gluconate Ribulose-5P
1
CHO
CH 2OH
O
4
CHO
CH
2
OP
CH 2OP
Sedoheptulose 7P
FRU-6P
CHO
5
6a
7
2
Ribose-5P
CH 2OH
O
Sedoheptulose
-
7P
CHO
FRU-1,6P 2CH 2OP
6b
Erythrose-4P
CH 2OP
O
Xylulose 5P
CH 2OP
Glycéraldéhyde-3P
(G3P)
DHAP G3P
Enzymes :
1 Glucose 6 P déshydrogénase
CH2OH
O
CHO
CHO
CH2O
H
O
CH2OH CH2OH
Xylulose
-
5P
1
.
Glucose 6
-
P déshydrogénase
2. Lactonase
3. 6-P gluconate déhydrogénas
e
4. Pentose-P isomérase
5 Pentose
-
P épimérase
O
R
CHO
R'
CHO
R
O
R'
cétose aldose aldose cétose
O
R
CHO
R'
CHO
R
O
R'
cétose
aldose
aldose
cétose
5
.
Pentose P épimérase
6a,b. Transcétolases
7. Transaldolase
Transaldolisation
19
cétose
aldose
aldose
cétose
Transcétolisation
Voie des pentoses phosphate
CH
3
-CO-COO-
PYR
CH2-COO-CH2-COO-CH2-COO-
3
NADH,H+
CO2
CO
2
CoASH 1
10 CH2-COO-
-COO-
HO-C
Cit
HO-CH-COO-
CH-COO-
iCit
O=C-COO-
CH
α-KG
2
CH3-CO-SCoA
CO
2
2
Cit
CH2-COO-
O=C SCoA
CH2
CH2-COO-
CH
2
-COO-
CH-COO-
-OOC-CH
OAA
NADH H
+
NADH,H+
9
CI
T
CoASH
iCIT
3
O=C
-
SCoA
Succ-CoA Succ
2
Fum
HO CH COO
-
O=C-COO
-
α-KG
CO2
NADH
,
H
9
8
MAL
CH
4
HO
-
CH
-
COO
Mal
CH2-COO-
O=C-COO
OAA
CH2-COO-
SCA
NADH,H+
FUM
C
oAS
H
5
6
7
S
ucc-
C
o
A
CO2
SUCC
GTP
FADH2
6
ENZYMES :
1. P
y
r. desh
y
dro
g
énase 2. Cit. s
y
nthétase 3. Aconitase
GTP
CoASH
yyg
y
4. iCit. deshydrogénase 5. α-KG. deshydrogénase
6. Succ-CoA synthétase 7. Succ. deshydrogénase
8. Fumarase 9. Mal. déshydrogénase 10. Pyr. carboxylase
20
Cycle de Krebs (CK)
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
1
/
14
100%