Document annexe

Pr. Catherine Michaux et Pr. Eric Perpète
Laboratoire de Chimie Physique des Biomolécules
Unité de Chimie Physique Théorique et Structurale
Département de Chimie
rue de Bruxelles, 61
B-5000 Namur
e-mail : [email protected]
3 propositions de stage dans le domaine des
biotechnologies, de la biochimie ou de la
modélisation moléculaire
1) Compréhension et optimisation d'une méthode de
renaturation de protéines et de peptides.
2) Production, purification et caractérisation de protéines de
transport pour des applications nanotechnologiques.
3) Modélisation moléculaire de la structure et de la dynamique
de protéines membranaires.

1) Compréhension et optimisation d'une méthode de
renaturation de protéines et de peptides.
La compréhension des phénomènes de repliement des protéines est un
défi majeur en biochimie car la structure tridimensionnelle d’une protéine à
l’état natif lui confère souvent une fonction spécifique. De nombreuses
protéines produites en laboratoire ou en industrie ne se trouvent pas dans leur
état fonctionnel; elles forment en effet des agrégats biologiquement inactifs et
sont donc non exploitables comme telles. A ce jour, il n’existe pas d’approche
expérimentale universelle et performante favorisant un repliement optimal.
Dans ce cadre, une méthode originale, basée sur l’association du
détergent SDS et d’un cosolvant, a été mise au point par notre groupe et
semble pouvoir s’appliquer à plusieurs types de protéines.
L’efficacité qualitative et quantitative de la méthode sera évaluée sur des
peptides et/ou protéines modèles par diverses méthodes analytiques,
physicochimiques et spectroscopiques. Nous pourrons ainsi évaluer l’influence
des concentrations respectives en détergent et cosolvant, ainsi que celle de
l’environnement physico-chimique.
Ce projet original et innovant ouvre de nombreuses perspectives dans
les domaines biotechnologique, médical et industriel.
Techniques utilisées : Spectroscopie UV/vis, Fluorescence, Diffusion
dynamique de la lumière, dichroïsme circulaire.

2) Production, purification et caractérisation de protéines de
transport pour des applications nanotechnologiques.
Ce projet s’inscrit dans une thématique multidisciplinaire s’intéressant à
la conception de nouveaux matériaux incorporant des protéines de transport
facilitant la diffusion de solutés. Les nanomatériaux composites résultants
offrent de nombreuses applications potentielles comme les nano-réacteurs,
nanosenseurs ou les systèmes de libération de médicaments.
Afin de considérer la mise en oeuvre de ces protéines vers de nouveaux
milieux imitant leur membrane naturelle, elles seront d’abord produites en
quantité suffisante et purifiées par chromatographie d’affinité. Puis, leur
transférabilité sera évaluée par leur reconstitution au sein de vésicules
composées de lipides ou de copolymères. L’activité et la diffusion relative de
différents solutés à travers le canal seront déterminées par diverses
techniques.
Techniques utilisées : Culture bactérienne in vitro, Purification de protéines
par chromatographie, Electrophorèse, Spectroscopies, Diffusion dynamique de
la lumière.

3) Modélisation moléculaire de la structure et de la dynamique
de protéines membranaires.
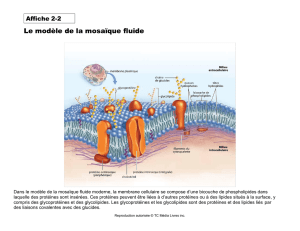
Les protéines membranaires sont des systèmes biologiques complexes.
Elles sont de première importance en biologie cellulaire et interviennent par
exemple comme canaux ioniques, récepteurs de médicaments ou transporteurs
de solutés. Au sein du génome humain, elles représentent approximativement
25% des séquences codantes, mettant en évidence leur importance,
notamment en tant que cibles potentielles pour le développement de nouveaux
médicaments. Néanmoins, il n'existe que très peu d'information structurale sur
ces protéines. La proportion de structures tridimensionnelles (3D) connues pour
ce type de protéines n'est d'ailleurs que de quelques pourcents, montrant la
difficulté de les isoler et de les caractériser.
Dans ce cadre, nous utiliserons d’abord des méthodes prédictives pour
construire un modèle 3D de ces protéines. Ensuite, nous ferons appel à la
dynamique moléculaire pour mettre en évidence les propriétés clés et la
structure des protéines insérées dans leur membrane biologique ou dans des
systèmes les mimant, comme des micelles de détergents. Les interactions
entre ces différents éléments seront analysées au niveau moléculaire.
Techniques utilisées : Méthodes de prédiction de structure, Dynamique
moléculaire.
1
/
4
100%