Document

1
Objectifs méthodologiques : raisonner, exploiter des arbres phylogénétiques, réaliser des comparaisons
Tous les êtres vivants actuels et fossiles sont plus ou moins apparentés. La phylogenèse est la reconstitution de l’histoire évolutive de différentes
lignées depuis leur divergence à partir d’un ancêtre commun. L’établissement des liens de parenté entre les êtres vivants permet de construire des
arbres phylogénétiques et de reconstituer l’évolution du monde vivant.
Problème : comment peut-on déterminer les relations de parenté ? Quels critères doit-on utiliser pour construire un
arbre phylogénétique ?
I. Comparaison de caractères chez les Vertébrés
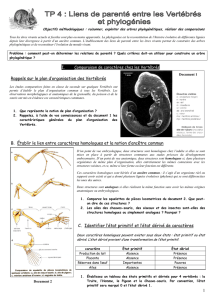
A. Rappels sur le plan d’organisation des Vertébrés
Les études comparatives faites en classe de seconde sur quelques Vertébrés
ont permis d’établir le plan d’organisation commun à tous les Vertébrés. Les
observations morphologiques et anatomiques de la grenouille, du poisson et de
la souris ont mis en évidence ces caractéristiques communes.
1. Que représente la notion de plan d’organisation ?
2. Rappelez, à l’aide de vos connaissances et du document 1 les
caractéristiques générales du plan d’organisation des Vertébrés.
B. Établir le lien entre caractères homologues et la notion d’ancêtre commun
D’un point de vue embryologique, deux structures sont homologues chez l’adulte si elles se sont
mises en place à partir de structures communes aux stades précoces du développement
embryonnaire. D’un point de vue anatomique, deux structures sont homologues si, dans
plusieurs organismes de même plan d’organisation, elles entretiennent les mêmes connexions
avec les structures voisines, et ce, même si leur forme ou leur fonction est différente.
Ces caractères homologues sont hérités d’un ancêtre commun ; il s’agit d’un organisme réel ou
supposé avoir existé et qui a donné plusieurs lignées évolutives (phylums) qui se sont
différenciées les unes des autres.
Deux structures sont analogues si elles réalisent la même fonction sans avoir les mêmes origines
anatomiques ou embryologiques.
3. Comparez les squelettes de pièces locomotrices du document 2. Que peut-on dire de
ces structures ?
4. Les ailes des chauves-souris, des oiseaux et des insectes sont-elles des structures
homologues ou simplement analogues ? Pourquoi ?
C. Identifier l’état primitif et l’état dérivé de caractères
Deux caractères homologues peuvent exister sous deux états : état primitif ou état dérivé. L’état
dérivé provient d’une transformation de l’état primitif.
caractère
État primitif « 0 »
État dérivé « 1 »
Production de lait
Absence
Présence
Placenta
Absence
Présence
Réserves dans l’œuf
Importantes
Pauvres
Ailes
Absence
Présence
5. Établissez un tableau des états primitifs et dérivés pour 4 vertébrés : la Truite,
l’Homme, le Pigeon et la Chauve-souris. Par convention, l’état primitif sera marqué
« 0 » et l’état dérivé « 1 ».
Document 2
Document 1

2
6. Sur chacun des trois arbres ci-dessous, placez les transformations évolutives des caractères étudiés au niveau des branches des lignées.
7. Pour choisir un arbre plutôt qu’un autre, on fait appel au principe d’économie d’hypothèses de transformations (= principe de
parcimonie) : un caractère ne doit apparaître ou disparaître qu’une fois. Choisissez un arbre en l’expliquant.
8. Quels caractères devait présenter l’ancêtre commun à l’Homme et à la Chauve-souris ?
II. Construction d’un arbre phylogénétique à partir de données anatomiques
Afin de comparer différentes espèces de Vertébrés et de construire des arbres phylogénétiques, nous allons utiliser le logiciel phylogène. Pour cela,
lancez le raccourci vers phylogène : « Phylo.exe ».
A. Établir un tableau de comparaison
Utilisez la commande « fichier », « sélectionner une collection » et choisissez la collection « vertébrés lycée »
La commande « comparer » permet de comparer différentes données morphologiques et anatomiques de différents animaux (4 au maximum).
9. Comparez (données anatomiques) le squelette puis l’organe locomoteur du Dauphin, du Requin, de l’Homme et de la Vipère. Quels sont
l’état primitif du caractère « organe locomoteur » et les états dérivés ?
10. Le caractère présence ou absence de nageoire sera-t-il un bon moyen de comparaison pour établir les liens de parenté ? Pourquoi ?
Utilisez la commande « construire » afin de réaliser le tableau de comparaison ci-dessous.
11. Remplissez le tableau sous phylogène et vérifiez qu’il est exact. Complétez le tableau ci-dessous.

3
Utilisez la commande « polariser » afin de déterminer les états primitifs et dérivés : il faut d’abord choisir l’animal de référence (« extragroupe »)
ne présentant aucun état dérivé parmi les caractères choisis (étape 1) puis cliquer sur « colorer les états primitifs suivant l’extragroupe ». Enfin, il
faut sélectionner et colorer tous
les états dérivés (utiliser la
commande « colorer de la
même manière les états
identiques) ; il peut y avoir
plusieurs états dérivés pour un
même caractère (choisir
différentes couleurs). Terminez
en vérifiant le codage.
Utilisez ensuite la commande « classer » afin de visualiser un aperçu des innovations évolutives et de créer l’emboîtement des groupes
monophylétiques. Sélectionnez l’option « afficher les boîtes ». Choisissez les caractères appropriés de classement pour réaliser une phylogénie.
Vérifiez toujours que le principe de parcimonie s’applique : une innovation évolutive identique pour deux groupes (= clades) différents est très peu
probable. Le logiciel interdit toute erreur de classement. Avec un clic droit sur les boîtes, on peut nommer les groupes.
12. Réalisez un classement le plus judicieusement possible. Celui-ci doit faire apparaître l’ordre d’apparition des caractères. Certains
caractères ne doivent pas être pris en compte pour établir le classement. Lesquels ? Discutez de la pertinence de l’existence du groupe
des « reptiles ».
B. Construction de l’arbre
Sélectionnez la commande « établir des parentés » afin de réaliser et améliorer visuellement l’arbre obtenu (vous pouvez déplacer des
branches et faire pivoter des groupes autour de chaque nœud). L’arbre obtenu est plus complet : il comprend les espèces possédant
l’ensemble des caractères à l’état primitif. Une fois l’arbre fait, cliquez sur l’onglet « choix options » et sélectionnez « explorer
l’arbre ». Vous pouvez ainsi avec un clic droit sur l’arbre faire apparaître les groupes et éventuellement leurs noms s’ils ont été
nommés à l’étape précédente.
Application : Complétez l’arbre ci-joint en indiquant les transformations évolutives.
TITRE :

4
III. Construction d’un arbre phylogénétique à partir de données moléculaires
Choisissez la commande « ouvrir un tableau de séquences », « myoglobine ». On utilise les symboles à
une lettre pour les acides aminés. Les identités sont signalées par des lettres rouges, les différences par
des lettres noires. Ne conserver que les animaux suivants : Thon, alligator, dauphin, homme, manchot
et requin en désélectionnant les autres
Le tableau de commande spécifique permet de demander l'affichage de la matrice mais pour l’instant
on s’intéresse à la représentation de type « arbre clade » : cliquez sur l’onglet « options » puis
choisissez « arbre clade ». Réalisez l’arbre phylogénétique par la comparaison des quelques acides
aminés suivants : positions 5, 6, 7, 17, 21, 41, 151.
13. Recopiez sur le compte-rendu l’arbre obtenu.
14. Expliquez pourquoi on peut retrouver le même acide aminé (par exemple en position 143) chez le
dauphin et le requin pourtant très éloignés génétiquement ?
Afin de comparer efficacement des molécules chez plusieurs animaux, il convient d’analyser la totalité
des différences entre les acides aminés qui la composent. Le logiciel peut faire cette comparaison grâce
au calcul des distances. Affichez la matrice des distances après avoir changé dans les options le type
d’arbre en UPGMA.
15. Recopiez la matrice des distances obtenue et classez les animaux par rapport à l’Homme depuis le plus
proche au plus éloigné génétiquement.
Cliquez sur l’onglet « arbre ». L’arbre obtenu tient compte de la totalité de la séquence étudiée et est fonction des distances
génétiques entre chaque espèce.
16. Quelle(s) remarque(s) pouvez-vous faire en comparant cet arbre avec celui établi par les données anatomiques ?
17. Y a-t-il une méthode plus fidèle que l’autre ? Pourquoi ?
BILAN
À l’aide de la commande affichage des distances (onglet ) les distances moléculaires relatives s’affichent.
Réalisez un arbre réaliste en complétant l’ébauche suivante, en tenant compte à la fois des données moléculaires et anatomiques. tenez
compte des distances, affichez également les innovations évolutives et les noms des différents clades (ex : la lamproie n’a pas mâchoire et
appartient au groupe des agnathes).
Code international des
acides aminés :
G - Glycine (Gly)
P - Proline (Pro)
A - Alanine (Ala)
V - Valine (Val)
L - Leucine (Leu)
I - Isoleucine (Ile)
M - Methionine (Met)
C - Cysteine (Cys)
F - Phenylalanine (Phe)
Y - Tyrosine (Tyr)
W - Tryptophan (Trp)
H - Histidine (His)
K - Lysine (Lys)
R - Arginine (Arg)
Q - Glutamine (Gln)
N - Asparagine (Asn)
E - Glutamic Acid (Glu)
D - Aspartic Acid (Asp)
S - Serine (Ser)
T - Threonine (Thr)
1
/
4
100%