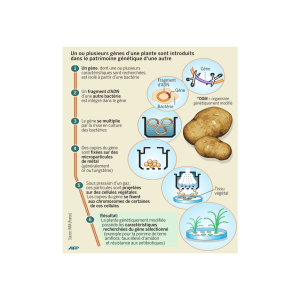
Comment identifier les gènes impliqués dans des maladies

Comment identifier les gènes
impliqués dans des maladies ?
•Trouverdes
familles où la
maladie ségrège
• Trouver le gène
candidat
• Confirmer le gène
candidat

Analyse des familles
Principe: transmission plus fréquente de la maladie
avec un marqueur génétique

Analyse des malades
Malades non malades
Principe: la maladie apparaît avec une fréquence
plus élevée pour un allèle spécifique d’un
marqueur donné

Les différents cas de recherche du
gène candidat

Approche biochimique : la fonction de la protéine
impliquée est connue exemple du facteur VIII
de coagulation et de l’hémophilie
Cascade
d’activation
lorsqu’un
vaisseau sanguin
est lésé
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
1
/
30
100%